Programme intégré canadien de surveillance de la résistance aux antimicrobiens (PICRA) 2017: Design et méthodes
Télécharger la version PDF, 2074 Ko
Promouvoir et protéger la santé des canadiens grâce au leadership, aux partenariats, à l'innovation et aux interventions en matière de santé publique, Agence de la santé publique du Canada.
Préserver l'efficacité des antimicrobiens utilisés chez les humains et les animaux, Programme intégré canadien de surveillance de la résistance aux antimicrobiens.
Also available in English under the title:
Canadian Integrated Program for Antimicrobial Resistance Surveillance (CIPARS) 2017: Design and Methods
Pour obtenir plus d'information, veuillez communiquer avec :
Dolly Kambo
Adjointe exécutive
Agence de la Santé Publique du Canada
370 Speedvale Avenue Ouest, Guelph, ON N1H 7M7
Téléphone : 519-826-2174
Télécopieur : 519-826-2255
Courriel : phac.cipars-picra.aspc@canada.ca
On peut obtenir, sur demande, la présente publication en formats de substitution.
Sa Majesté la Reine du chef du Canada, représentée par la ministre de la Santé, 2020
Date de publication : décembre 2020
La présente publication peut être reproduite sans autorisation pour usage personnel ou interne seulement, dans la mesure où la source est indiquée en entier.
Cat. : HP2-4/2017F-3-1-PDF
ISBN : 978-0-660-36794-1
Pub. : 200318
Citation suggérée :
Gouvernement du Canada. Programme intégré canadien de surveillance de la résistance aux antimicrobiens (PICRA) 2017 : Design et méthodes. Agence de la santé publique du Canada, Guelph (Ontario), 2020.
Table des matières
- Nouveautés du PICRA en 2017
- Design et méthodes
- Utilisation des antimicrobiens
- Résistance aux antimicrobiens
- Surveillance chez les humains
- Surveillance de la viande vendue au détail
- Surveillance à l'abattoir
- Surveillance à la ferme
- Surveillance des isolats cliniques animaux
- Aliments et ingrédients des aliments pour animaux
- Méthodes d'isolement des bactéries
- Méthodes de sérotypage et de lysotypage
- Méthodologies des tests de sensibilité aux antimicrobiens
- Valeurs seuils de la sensibilité aux antimicrobiens
- Analyse des données
- Classification des antimicrobiens
- Annexe
Nouveautés du PICRA en 2017
Utilisation des antimicrobiens chez les animaux
- En 2017, Pêches et Océans Canada a fourni les données sur les quantités d'antimicrobiens utilisés en aquaculture chez les poissons de mer et d'eau douce.
Résistance aux antimicrobiens
- En 2017, nous avons effectué, dans le cadre de la vente au détail, un échantillonnage en Ontario et dans les Prairies pour une partie de l'année seulement et aucun échantillonnage n'a été effectué dans la région de l'Atlantique.
- Le prélèvement d'échantillons de dindon haché vendu au détail, pour la détection de Campylobacter spp., a été interrompu de façon définitive en 2017.
- En 2017, les échantillons de 2017 communs à FoodNet et au PICRA ont été séquencés à l'aide de la plateforme MiSeq d'IlluminaMD; le sérotype prédictif a été déterminé en utilisant SISTR (Salmonella in silico Typing Resource).
- Pour 2017, les isolats de Salmonella de sérotype Enteritidis, Heidelberg et Typhimurium ont été soumis à un test de lysotypage s'ils étaient envoyés avant le 28 septembre 2017; tous les isolats soumis après cette date n'ont pas été lysotypés.
Design et méthodes
Utilisation des antimicrobiens
Les activités de surveillance de l'utilisation d'antimicrobiens chez les humains réalisées par l'Agence de la santé publique du Canada (ASPC) sont présentées dans la mise à jour de 2018 du Système canadien de surveillance de la résistance aux antimicrobiens (SCSRA)Note de bas de page 1. Une sélection de certaines données provenant de IQVIA (anciennement connu sous le nom de QuintilesIMS) et présentées dans le rapport de 2017 du SCSRA ont été ajoutées dans les résultats intégrés de ce rapport (entente verbale avec le SCSRA).
Quantités d'antimicrobiens distribués pour la vente aux fins d'utilisation chez les animaux
Les données sur les ingrédients actifs distribués pour la vente ont été regroupées et transmises à l'Agence de la santé publique du Canada par l'Institut canadien de la santé animale (ICSA) à titre d'estimation des antimicrobiens utilisés chez les animaux. L'ICSA est une association commerciale qui représente les fabricants et distributeurs de médicaments destinés aux animaux de compagnie, aux animaux utilisés dans les sports et aux animaux destinés à l'alimentation (y compris les poissons) au Canada. Selon cette association, les ventes de ses membres représentent approximativement 95 % de toutes les ventes de produits pharmaceutiques vétérinaires homologués au CanadaNote de bas de page 2. L'ICSA coordonne la collecte électronique de données provenant de ses membres. La collecte et l'analyse des données sont effectuées par une tierce partie, Impact Vet. Les données de l'ICSA comprennent des renseignements provenant de 17 fabricants de produits antimicrobiens destinés à l'utilisation chez les animaux au Canada et de 4 grossistes ou distributeurs importants. Les données de l'ICSA relatives à la distribution des antimicrobiens utilisés chez les animaux offrent un contexte qui permet d'interpréter d'autres données sur l'utilisation des antimicrobiens chez les animaux qui sont recueillies dans le cadre de la surveillance à la ferme ou la recherche. Elles constituent également un moyen d'estimer les variations temporelles globales associées à l'utilisation d'antimicrobiens chez les animaux.
Le nombre de kilogrammes d'ingrédients actifs est déclaré au PICRA à l'échelle du fabricant d'aliments et de la clinique vétérinaire dans la chaîne de distribution. Les fabricants ont attribué l'utilisation d'antimicrobiens aux animaux d'élevage (y compris les chevaux) ou aux animaux de compagnie selon les allégations figurant sur l'étiquette, et dans le cas où l'étiquette indiquait les deux types d'animaux, le fabricant a attribué (estimé) son utilisation aux animaux de compagnie ou aux animaux d'élevage.
Ces données ne représentent pas l'utilisation réelle d'antimicrobiens pour une année donnée, mais le volume d'antimicrobiens distribués par les fabricants et les grossistes. Les quantités distribuées devraient correspondre approximativement aux quantités utilisées, en particulier lorsque les données incluses couvrent plus d'une année. Toutefois, lorsqu'il s'agit de données couvrant une seule année, les volumes distribués peuvent varier par rapport aux quantités réellement utilisées en raison de l'intervalle de temps entre la distribution des médicaments et leur utilisation réelle, ainsi qu'en raison de la constitution de stocks d'antimicrobiens à différentes étapes du système de distribution. Les données des ventes ne représentent pas non plus les pertes de médicaments en raison de leur péremption.
Les données n'incluent pas les antimicrobiens importés pour usage personnel en vertu de la disposition pour approvisionnement personnel de la Loi sur les aliments et drogues du gouvernement fédéral et de son Règlement. Elles n'incluent pas non plus les ingrédients pharmaceutiques actifs importés en vrac et préparés par un pharmacien ou un vétérinaire accrédité. Les derniers renseignements provenant d'une étude Ipsos/Impact Vet, menée pour l'ICSA, indiquent que les occasions manquées d'obtenir des données en raison de l'importation pour usage personnel et de l'importation d'ingrédients pharmaceutiques actifs sont estimées à 13 % du total des ventes de produits de santé animale (communication personnelle, Jean Szkotnicki). Les données de l'ICSA ne comprennent pas les ordonnances d'antimicrobiens à usage humain exécutées par les pharmaciens pour des animaux de compagnie. Par conséquent, les données de l'ICSA sous-estiment le volume réel d'antimicrobiens utilisés chez les animaux au Canada. De plus, les données de l'ICSA ne représentent pas ce qui se produit après l'achat des médicaments; par conséquent, ces données ne fournissent pas de renseignements sur les pratiques réelles d'utilisation des antimicrobiens, comme le dosage, la durée d'utilisation, la justification de l'utilisation, les renseignements détaillés propres à un médicament ou l'utilisation en dérogation des directives de l'étiquette.
Les données de l'ICSA comprennent des médicaments vendus directement aux pharmaciens qui se spécialisent dans les médicaments destinés aux animaux d'élevage. Elles n'incluent pas les agents antimicrobiens que les vétérinaires transmettent aux pharmacies et qui sont ensuite utilisés par les pharmacies. Cette dernière distribution se reflète dans les données à l'échelle des cliniques vétérinaires.
L'ICSA fournit ces renseignements regroupés en catégories, et certains antimicrobiens ne sont pas déclarés de façon indépendante. Il s'agit d'une « règle de 3 par entreprise » établie par l'ICSA pour se conformer à la réglementation anticoncurrentielle de l'Union européenne et des États-Unis. Dans certains cas, l'ICSA ajoute une « règle de 90 % » afin de ne pas contrevenir aux règlements des États-Unis. Ces règles comptables peuvent entraîner des modifications de la catégorisation d'antimicrobiens précis au cours du temps. Pour l'année 2017, les antimicrobiens sont catégorisés selon le tableau 1.
| Classe d'antimicrobiens | Ingrédient |
|---|---|
| Aminoglycosides | Amikacine, apramycine, dihydrostreptomycine, sulfate de framycétine, gentamicine, néomycine, spectinomycine, streptomycine |
| β-Lactames/pénicillines | Amoxicilline, ampicilline, acide clavulanique, cloxicilline, pénicilline |
| Céphalosporines | céfadroxil, céfalexine, céphapirine, céfazoline, céfovecin, céfoxitine, cefpodoxime, ceftiofur |
| Fluoroquinolones | Ciprofloxacine, danofloxacine, enrofloxacine, marbofloxacine, orbifloxacine, pradofloxacine |
| Anticoccidiens de synthèse et arsénicaux | Amprolium, clopidol, décoquinate, diclazuril, halofugione, narasin, pyriméthamine, robénidine, toltrazuril |
| Anticoccidiens ionophores | Lasalocide, maduramicine, monensin, salinomycine |
| Lincosamides | Clindamycine, lincomycine, pirlimycine |
| Macrolides | Érythromycine, gamithromycine, tildipirosine, tilmicosine, tulathromycine, tylosine |
| Autres antimicrobiens | Avilamycine, bacitracine, bambermycine, chloramphénicol, gluconate de chlorhexidine, florfénicol, acide fusidique, novobiocine, polymixine B, tiamuline, virginiamycine |
| Tétracyclines | Chlortétracycline, doxycycline, minocycline, oxytétracycline, tétracycline |
| Triméthoprime et sulfamides | Sulfadiazine, sulfadoxine, sulfamérazine, sulfaméthazine, sulfaméthoxazole, sulfaquinoxaline, sulfathiazole, triméthoprime |
Figures temporelles et tableaux de données pour les tests d'analyse statistique
Puisque les données de l'ICSA représentent des informations de recensement, aucune évaluation n'a été effectuée à l'égard des différences statistiques entre les années (c.-à-d. que les données de l'ICSA ne sont pas des données qui proviennent d'échantillons). Toute différence observée entre les résultats devrait refléter une différence réelle quant aux quantités d'antimicrobiens distribués pour la vente par les sociétés membres.
« Population correction unit »
Les variations de la quantité globale d'antimicrobiens distribués au cours du temps peuvent refléter différentes situations : une évolution réelle des pratiques d'utilisation, une évolution du nombre ou du type d'animaux dans la population (qui nécessitent des antimicrobiens), une évolution de la prévalence des maladies qui nécessitent l'utilisation d'antimicrobiens, ou une évolution des types d'antimicrobiens administrés. Afin d'ajuster les données des ventes en fonction de l'évolution des populations animales au cours du temps, on a appliqué un dénominateur qui représente le nombre d'animaux et leurs poids (biomasse animale). Ce dénominateur est fondé sur la méthodologie actuellement utilisée par le réseau de Surveillance européenne de la consommation d'antibiotiques à usage vétérinaire (ESVAC)Note de bas de page 3.
L'ESVAC ajuste les données des ventes au moyen d'une unité corrigée de la population (PCU) qui est un indicateur de la biomasse animale susceptible de recevoir un traitement par des antimicrobiens. Il s'agit d'une mesure technique seulement, où 1 PCU = 1 kg de différentes catégories d'animaux vivants et abattus. La méthodologie de l'ESVAC a été utilisée le plus possible, mais les renseignements sur la population recueillis par Statistique Canada et Agriculture et Agroalimentaire Canada présentent une structure légèrement différente de celle des données auxquelles l'ESVAC a accès (Eurostat et TRACES). Par conséquent, les comparaisons directes des PCU ou des mg/PCU avec les données des pays qui participent à l'ESVAC doivent être établies avec prudence.
Le PCU est calculé en multipliant le nombre d'animaux vivants et abattus de chaque espèce et de chaque stade de production par le poids théorique (normalisé) au moment le plus probable du traitementNote de bas de page 4,Note de bas de page 5.
Équation 1 Formule pour le calcul du PCU
Équation 1a. Formule pour le calcul du PCU
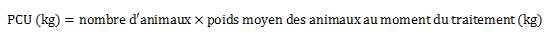
Équation 1a - Équivalent textuel
Le PCU (en kilogramme) est calculé en multipliant le nombre d'animaux par le poids moyen des animaux au moment du traitement (en kilogramme)
Équation 1b. Formule pour le calcul du PCU

Équation 1b - Équivalent textuel
Les milligrammes par « Population correction unit » (PCU) est calculée en divisant la quantité d'antimicrobiens distribuée (en mg) par la valeur du PCU (en kilogramme)
Les données du dénominateur national à propos du nombre d'animaux vivants et abattus ont été obtenues des sites Web de Statistique Canada, d'Agriculture et Agroalimentaire Canada, de Pêches et Océans Canada, de l'Institut canadien de la santé animale et de Canada Équestre (anciennement nommé Canada Hippique). Il convient de noter que certains sites Web mettent à jour périodiquement leurs données historiques; par conséquent, les données sont considérées comme les plus exactes possible à la date où elles sont consultées.
À l'automne 2013, le PICRA a rencontré des personnes volontaires affiliées à des groupes de production animale, à l'industrie pharmaceutique et certains représentants des gouvernements provinciaux de l'agriculture pour discuter sur les poids moyens canadiens au moment du traitement. Au moyen des données du PICRA qui étaient disponibles et des commentaires recueillis par ces participants (avis du comité d'experts), nous avons déterminé conjointement les poids moyens canadiens au moment du traitement. Cette démarche repose sur le principe que les animaux canadiens, dans le cas de certaines espèces animales ou certains stades de production, pourraient être plus lourds ou plus légers que les animaux européens.
En 2017, sur la base d'une consultation avec un expert de l'industrie, le PICRA a modifié le poids des porcs exportés par le Canada (destinés à l'alimentation) pour le calcul du PCUCANADA. Le PICRA a également appliqué le poids de 1 kg pour la volaille importée et exportée pour le calcul du PCUESVAC, mais a utilisé les catégories de poids canadiennes déclarées pour déterminer le PCUCANADA.
Inclusions et exclusions détaillées pour le dénominateur du PCU
Conformément à la méthodologie de l'ESVAC, les animaux exportés ont été ajoutés au PCU, tandis que les animaux importés en ont été soustraits, selon l'hypothèse de l'ESVAC que les animaux sont traités dans leur pays d'origine. Cependant, il a été noté que dans le contexte canadien, cette situation varierait en fonction du stade de production dans lequel se trouvent les animaux qui traversent la frontière.
Aux fins de calcul du PCU, les espèces d'animaux d'élevage dont les populations sont les plus importantes ont été incluses, à l'aide des mêmes catégories d'élevage que l'ESVAC (pour la plupart, en fonction de la disponibilité des données), à l'exception notable de l'inclusion supplémentaire des vaches de boucherie (qui ne sont pas incluses par l'ESVAC).
Les espèces actuellement exclues des calculs du PCU comprennent les gibiers (p. ex. orignal), les animaux de compagnie de « poche » (p. ex. hamsters, cochons d'Inde, oiseaux de compagnie), les reptiles et les amphibiens.
Pour certains stades de production, les données d'importation et d'exportation pour les volailles sont incluses dans une structure différente avant et après 2009, selon les données accessibles de Statistique Canada. Les importations et les exportations de volailles pour certaines catégories de poids ont été ajoutées, alors qu'elles ne sont pas incluses dans la méthodologie de l'ESVAC.
Stratification provinciale du numérateur et du dénominateur
Il est possible que des antimicrobiens soient redistribués dans d'autres provinces après leur distribution dans les cliniques vétérinaires (en particulier la redistribution d'aliments médicamentés : par exemple, des renseignements invérifiables indiquent que le Nouveau-Brunswick dispose d'une industrie négligeable quant à ses meuneries et qu'il achète généralement ses aliments médicamentés au Québec). Par conséquent, il faut être prudent lorsqu'on interprète les quantités d'antimicrobiens distribués pour la vente au sein de chaque province. Les calculs provinciaux/régionaux du PCU doivent faire l'objet d'une discussion plus poussée.
Discussion globale des forces et des limites
Les données de l'ICSA fournissent une mesure approximative des antimicrobiens distribués pour la vente et destinés à toutes les espèces animales, y compris ceux qui ne font pas l'objet de la Surveillance à la ferme du PICRA (avec les réserves appropriées concernant les importations pour usage personnel et les importations d'ingrédients pharmaceutiques actifs). Actuellement, la mesure du PCU ne tient pas compte de la durée de vie de l'animal, qui peut influencer l'interprétation des quantités d'antimicrobiens administrés aux animaux. De plus, l'utilisation d'un poids standard statique peut ne pas refléter un changement de la production dans l'industrie influant sur les poids moyens des animaux traités et attribuable à des conditions météorologiques, au commerce ou à d'autres raisons. Les mesures de l'utilisation d'antimicrobiens déclarées selon de grandes catégories et selon un dénominateur, le PCU, ne tiennent pas compte de la quantité de médicament nécessaire pour atteindre un succès thérapeutique. Ceci pourrait affecter l'interprétation des tendances. Par exemple, une diminution de la valeur des mg/PCU pourrait refléter un changement d'utilisation au profit d'un médicament ayant une plus petite dose quotidienne, plutôt qu'une diminution de l'exposition réelle des animaux aux antimicrobiens. Les données de l'ICSA doivent être interprétées comme une mesure décrivant l'utilisation d'antimicrobiens chez les animaux. Une extrême prudence est de mise lorsqu'on procède à des inférences en vue de toute utilisation chez une espèce animale précise.
Au cours des dernières années, plusieurs progrès ont été réalisés à l'égard du raffinement de ces données. En effet, depuis 2011, les données sont stratifiées par province; depuis 2012, elles sont stratifiées par animaux de compagnie/animaux d'élevage; et, depuis 2013, elles sont stratifiées par voie d'administration.
Quantités d'antimicrobiens distribués pour la vente aux fins d'utilisation dans les cultures
L'Agence de réglementation de la lutte antiparasitaire (ARLA) de Santé Canada recueille les données sur les ventes canadiennes annuelles auprès de tous les fabricants de pesticides. L'ARLA a gracieusement fourni au PICRA les renseignements sur les ventes d'antimicrobiens homologués aux fins d'utilisation comme pesticides sur les cultures. Ces données représentent les antimicrobiens ayant été administrés pour les raisons suivantes : brûlure bactérienne sur les fruits à pépins (pommes, poires, coings), les framboises, les mûres et les baies de Saskatoon; flétrissement des bourgeons et chancre bactérien sur les cerises; chancre de la tige et tache bactérienne sur les légumes-fruits en serre ou en champ (poivrons, tomates et aubergine); et bactériose du noyer sur les noix de Grenoble. Dans le but de protéger les renseignements commerciaux confidentiels, les données sont présentées uniquement en combinaison avec les données pour les humains et les animaux.
Quantités d'antimicrobiens utilisés en aquaculture chez les poissons de mer et d'eau douce
Pêches et Océans Canada (MPO) exige des exploitants de l'industrie d'aquaculture qu'ils signalent leur utilisation de médicaments, y compris les antimicrobiens (comme l'autorise la Loi sur les Aliments et drogues) et les pesticides (enregistrés en vertu de la Loi sur les produits antiparasitaires) en vertu de la Loi sur les produits antiparasitaires encadrée par l'autorité du Règlement sur les activités d'aquaculture autorisées en vertu de la Loi sur les pêches. Dans un rapport annuel, les exploitants aquacoles sont tenus de déclarer la quantité de médicaments et de pesticides utilisés tout au long de l'année à chaque endroit (c.-à-d. site agricole). À partir de ces données, le nombre d'ordonnances et la fréquence des périodes de traitement sont calculés, en plus des mesures prises pour éviter la nécessité d'une telle utilisation. Ces données couvrent toutes les installations aquacoles de poissons d'eau douce et de mer au Canada. Vous trouverez de plus amples renseignements sur l'utilisation d'antimicrobiens et d'autres produits par l'industrie aquacole au Canada sur le site Web du MPO sur l'aquacultureNote de bas de page 6.
Surveillance à la ferme
Questionnaire pour les fermes d'élevage
Poulets de chair
Dans la composante Surveillance à la ferme des poulets de chair du Programme intégré canadien de surveillance de la résistance aux antimicrobiens (PICRA), les données sur les fermes sentinelles étaient recueillies par l'intermédiaire de questions posées par le vétérinaire en médecine aviaire (ou le personnel technique désigné) au producteur (ou au personnel agricole désigné). Les questionnaires permettaient de recueillir des renseignements liés aux couvoirs et aux fermes d'élevage de poulets de chair. Le vétérinaire a demandé au producteur les reçus de livraison des poussins qui renferment les renseignements nécessaires pour remplir la portion du questionnaire qui concerne les couvoirs, comme les renseignements sur le troupeau reproducteur, y compris l'origine (c.-à-d. la province d'origine ou d'importation), l'intervalle d'âge du troupeau reproducteur, si le couvoir a acheté les poussins sous forme d'œufs d'incubation ou de poussins, les antimicrobiens utilisés et les voies d'administration, le dosage, les raisons principales (traitement, prévention, troupeau reproducteur à haut risque, demande du producteur), les raisons secondaires ou selon la maladie diagnostiquée (infection à E. coli aviaire pathogène, Enterococcus cecorum, Salmonella spp., Staphylococcus spp., infections clostridiales précoces et d'autres maladies) ainsi que tous les vaccins administrés in ovo ou à l'éclosion. Le vétérinaire ou un membre du personnel désigné a confirmé les renseignements en appelant les couvoirs.
On a complété à la portion du questionnaire qui concerne les fermes à l'aide des reçus de livraison des aliments (moulée), des dossiers de la ferme, des ordonnances ou de questions posées au producteur. On a également obtenu les renseignements démographiques de la ferme (p. ex. période contingentaire, âge et estimation du poids des oiseaux au moment de la visite, capacité de la ferme, du poulailler et de l'étage), ainsi que les renseignements en matière de biosécurité et de santé animale (p. ex. vaccins administrés à la ferme).
On a questionné le producteur ou le membre du personnel agricole désigné à propos de l'utilisation d'antimicrobiens administrés par les aliments et l'eau. Des données ont été recueillies à propos de chaque type d'aliment servi au troupeau, y compris les aliments médicamentés et non médicamentés (les moulées non médicamentées ne comprenaient pas d'antimicrobiens), le nombre total de jours au cours desquelles chaque type d'aliment était servie et l'âge des troupeaux au début et à la fin de chaque ration. D'autres renseignements ont été recueillis à propos des aliments contenant des antimicrobiens tels que le ou les ingrédients actifs, leur concentration dans les aliments et les raisons principales de cette utilisation d'antimicrobiens (stimulation de la croissance, prévention ou traitement de maladies). Les raisons secondaires d'utilisation d'antimicrobiens ou selon la maladie diagnostiquée ont été recueillies lorsque la raison principale de l'utilisation était le traitement ou la prévention de maladies. La liste des raisons secondaires comprenait les maladies les plus fréquemment diagnostiquées chez les poulets de chair : omphalite, septicémie, maladies musculosquelettiques, maladies respiratoires, entérite nécrotique, coccidiose et d'autres maladies (p. ex. toute étiologie non bactérienne, comme les étiologies virales et métaboliques).
Les données recueillies sur l'exposition aux antimicrobiens par l'eau comprenaient les ingrédients actifs dans les médicaments utilisés, le dosage (g ou mL/L d'eau de consommation), l'âge au début et à la fin de chaque médication administrée par l'eau, la proportion de troupeaux exposés et les raisons de l'utilisation. Les raisons principales et secondaires pour la prévention et le traitement pour l'administration d'antimicrobiens par l'eau étaient semblables à celles décrites pour l'administration d'antimicrobiens par les aliments. On a également demandé au producteur s'il avait reçu une ordonnance d'un vétérinaire si le médicament utilisé dans l'eau était un achat en vente libre.
Selon les éléments exigés par la Norme nationale de biosécurité pour les fermes avicolesNote de bas de page 7, des questions pertinentes ont été posées à propos du niveau de biosécurité. Des questions sur la gestion de l'accès, la gestion de la santé animale et la gestion opérationnelle ont été incluses. On a également recueilli des données sur l'état de santé du troupeau (p. ex. diagnostic des maladies bactériennes et virales les plus courantes) et l'administration de vaccins depuis le placement des poussins.
Porcs en croissance-finition
Dans la composante Surveillance à la ferme des porcs en croissance-finition du PICRA, les données sur les fermes sentinelles étaient recueillies par l'intermédiaire de questions posées par le vétérinaire en médecine porcine (ou le personnel technique désigné) au producteur (ou au personnel agricole désigné). Les questionnaires incluaient des sections demandant de l'information sur l'utilisation d'antimicrobiens, les caractéristiques démographiques des troupeaux et la santé des animaux.
Les questions portant sur le nombre de porcs dans la population à l'étude différaient selon le système de gestion : en rotation ou en tout plein-tout vide. La gestion en tout plein-tout vide est un système de production dans lequel les porcs arrivent dans la porcherie et en sortent, en lots distincts. En évitant de mélanger les lots, on espère réduire les risques de propagation des maladies. Habituellement, les installations sont entièrement nettoyées et désinfectées entre chaque lot. Cette méthode d'élevage se fait généralement par salle ou par bâtiment. Dans le système de gestion en rotation, des animaux sont continuellement enlevés et ajoutés du système de production.
Le questionnaire sur l'utilisation d'antimicrobiens était conçu pour recueillir des données sur les troupeaux de porcs en croissance-finition. Aucune donnée individuelle sur les porcs n'a été recueillie. Six parcs de porcs représentatifs de cette population ont été sélectionnés et des échantillons de matière fécale y ont été prélevés à des fins d'isolement bactérien et pour faire l'objet de tests de sensibilité aux antimicrobiens. Par conséquent, dans le cas des troupeaux en tout plein-tout vide, la population à l'étude comprenait tous les porcs qui sont arrivés dans la porcherie et en sont sortis et qui faisaient partie du même groupe que les porcs échantillonnés. La population à l'étude dans les troupeaux en rotation concernait les porcs qui sont entrés dans l'unité de croissance-finition avec les porcs échantillonnés.
Des questions ont également été posées aux propriétaires et aux gérants des troupeaux sur leur utilisation d'antimicrobiens ajoutés aux aliments, à l'eau ou administrés par injection. Les renseignements recueillis sur chaque type d'aliment administré au cours de la période de croissance-finition comprenaient la présence d'antimicrobiens dans les aliments (moulée médicamentée) ou l'absence d'antimicrobiens (moulée non médicamentée), le nombre moyen de semaines au cours desquelles chaque ration était servie et les poids des porcs au début et à la fin de cette période. D'autres renseignements ont été recueillis à propos des rations contenant des antimicrobiens : les ingrédients antimicrobiens actifs, la concentration des ingrédients actifs dans la moulée et les raisons principales de cette utilisation d'antimicrobiens (une parmi la stimulation de la croissance, la prévention et le traitement de maladies). Si la justification principale d'utilisation d'antimicrobiens était la prévention et le traitement de maladies, les répondants pouvaient choisir l'une des raisons secondaires suivantes de l'utilisation d'antimicrobiens dans les aliments : maladie respiratoire, maladie entérique, boiterie ou autres maladies. On a également noté la proportion de porcs ayant reçu chaque aliment.
Les données recueillies sur l'exposition aux antimicrobiens par l'eau ou des injections comprenaient les ingrédients actifs dans les médicaments utilisés, les raisons de l'utilisation et la proportion de porcs exposés. Les raisons principales d'utilisation d'antimicrobiens dans l'eau comprenaient la prévention et le traitement de maladie, ainsi que les raisons secondaires connexes d'utilisation suivantes : maladie respiratoire, maladie entérique, boiterie ou autres maladies. Seules les raisons de traitement de maladie ont été recueillies pour l'utilisation d'antimicrobiens administrés par injection. Le nombre de porcs exposés à l'utilisation d'antimicrobiens dans l'eau ou par injection a été recueilli sous forme de données de catégorisation parmi les intervalles suivants : 1 à 25 %, 26 à 50 %, 51 à 75 % ou de 76 à 100 % des porcs. Aucune donnée sur l'utilisation d'antimicrobiens n'a été recueillie pour les stades de production antérieurs au stade de croissance-finition.
Aucune donnée sur l'utilisation d'antimicrobiens n'a été recueillie pour les stades de production antérieurs au stade de croissance-finition. Toutes les données relatives à l'utilisation d'antimicrobiens chez les porcs pesant moins de 15 kg (33 lb) ont été exclues, puisqu'à ce poids, car il est inférieur au standard de l'industrie des porcs en croissance-finition.
Dindons
Dans la composante Surveillance à la ferme des dindons du PICRA, les données sur les fermes sentinelles étaient recueillies par l'intermédiaire de questions posées par le vétérinaire en médecine aviaire (ou le personnel technique désigné) au producteur (ou au personnel agricole désigné). Des données ont été recueillies sur le marché visé pour les oiseaux échantillonnés. Les marchés potentiels étaient les suivants : dindons de chair âgés entre 64 et 71 jours et pesant en moyenne 5,5 kg, dindons femelles légères âgées entre 76 et 83 jours et pesant en moyenne 7,2 kg, dindons femelles lourdes âgées entre 99 et 106 jours et pesant en moyenne 9,4 kg, dindons mâles légers âgés entre 97 et 104 jours et pesant en moyenne 12,2 kg et dindons mâles lourds âgés entre 109 et 116 jours et pesant en moyenne 15,1 kg.
Des renseignements concernant l'utilisation de médicaments au couvoir ont été obtenus à l'aide des reçus de livraison des dindonneaux ou en appelant les couvoirs (s'ils étaient d'origine canadienne). Les données recueillies comprenaient des renseignements sur le troupeau reproducteur, y compris l'origine (p. ex., la province d'origine ou d'importation), l'intervalle d'âge du troupeau reproducteur, si le couvoir a acheté les dindonneaux sous forme d'œufs d'incubation ou de dindonneaux, les antimicrobiens utilisés et les voies d'administration et le dosage. De plus, la raison principale de l'utilisation d'antimicrobiens, telle que le traitement, la prévention, le risque élevé associé au troupeau reproducteur source ou la demande du producteur, a été fournie. La bactérie ou la maladie ciblée a aussi été mentionnée : E. coli, Salmonella spp., Staphylococcus spp. ou autres.
La portion du questionnaire qui concerne l'utilisation d'antimicrobiens à la ferme a été remplie à l'aide des reçus de livraison des aliments, des dossiers de la ferme, des ordonnances ou de questions posées au producteur. Des renseignements démographiques ont été recueillis au sujet de la ferme, de l'âge et du poids estimé des oiseaux au moment de la visite, de la capacité de la ferme, du poulailler et de l'étage, ainsi que des renseignements en matière de biosécurité et de santé animale (c.-à-d., les vaccins administrés à la ferme).
Le producteur ou le membre du personnel agricole désigné a été questionné sur l'UAM par les aliments et l'eau. Des données ont été recueillies à propos de chaque type d'aliment servi au troupeau. Ces données indiquaient si l'aliment contenait ou non des antimicrobiens (aliment médicamenté et non médicamenté), le nombre total de jours au cours desquels chaque type d'aliment était servi et l'âge des troupeaux au début et à la fin de chaque ration. D'autres renseignements ont été recueillis à propos des aliments contenant des antimicrobiens tels que le ou les ingrédients actifs, leur concentration dans les aliments et les raisons principales de cette utilisation d'antimicrobiens (stimulation de la croissance, prévention ou traitement de maladies). Les raisons secondaires d'utilisation d'antimicrobiens ont été mentionnées lorsque la raison principale de l'utilisation était la prévention ou le traitement de maladies. La liste des raisons secondaires comprenait les maladies les plus fréquemment diagnostiquées chez les dindons : omphalite, septicémie, maladies musculosquelettiques, maladies respiratoires, maladies entériques, coccidiose et d'autres maladies (p. ex. toute étiologie non bactérienne, comme les étiologies virales et métaboliques).
Les données recueillies sur l'exposition aux antimicrobiens par l'eau comprenaient les ingrédients actifs dans le ou les médicament(s) utilisé(s), le dosage (g ou ml/L d'eau de consommation), l'âge au début et à la fin de chaque médication administrée par l'eau, la proportion du troupeau exposé et la ou les raison(s) de l'utilisation. Les raisons principales et secondaires pour la prévention et le traitement pour l'administration d'antimicrobiens par l'eau étaient semblables à celles décrites pour l'administration d'antimicrobiens par les aliments. On a également demandé au producteur s'il avait reçu une ordonnance d'un vétérinaire ou si le médicament utilisé dans l'eau était un achat en vente libre.
Selon les éléments exigés par la Norme nationale de biosécurité pour les fermes avicolesNote de bas de page 8des questions pertinentes ont été posées à propos du niveau de biosécurité. Des questions sur la gestion de l'accès, la gestion de la santé animale et la gestion opérationnelle ont été incluses. On a également recueilli des données sur l'état de santé du troupeau (c.-à-d., le diagnostic des maladies bactériennes et virales les plus courantes) et l'administration de vaccins depuis le placement des dindonneaux.
Analyse des donnéesNote de bas de page 9
Les données ont été saisies dans une base de données PostGreSQL et des statistiques descriptives en ont été tirées au moyen d'un logiciel offert sur le marchéNote de bas de page 10.
Poulets de chair
Les expositions aux antimicrobiens de l'éclosion à la fin de la croissance ou au stade d'échantillonnage de fin de production (plus grand que ou égal à 30 jours) ont été résumées pour chaque troupeau. Par exposition, on entend toute utilisation déclarée d'un ingrédient actif par une voie d'administration donnée. Les données sont déclarées sous forme d'exposition à un ingrédient actif par une voie d'administration donnée, ainsi que par exposition à un ingrédient actif par toute voie d'administration. Ces expositions sont résumées par classe d'antimicrobiens.
Consommation d'aliments
Les estimations relatives à la consommation d'aliments reposaient sur une régression simple et un calcul intégral. Les estimations de consommation d'aliments provenant des standards de performance les plus récents (souches Ross et Cobb) et des objectifs de performance définis par les entreprises spécialisées dans la nutritionNote de bas de page 11,Note de bas de page 12,Note de bas de page 13,Note de bas de page 14,Note de bas de page 15 ont été enregistrées dans MicrosoftMC Excel. À partir de ces données, la consommation cumulative d'aliments a été calculée en utilisant la moyenne des normes relatives à l'alimentation des animaux pour les 2 souches les plus communes de poulets de chair ainsi que les standards définis par les entreprises spécialisées dans la nutrition (c.-à-d., qui ne sont pas propres à la souche) pour les poulets de chair dès leur éclosion (c.-à-d., mâles et femelles combinés). Cela a permis de créer une courbe pour la consommation d'aliments en gramme par oiseau par jour.
Le questionnaire sur les poulets de chair indiquait l'âge des oiseaux correspondant au début et à la fin de chaque ration. Étant donné que le dernier jour d'une ration était le premier jour de l'autre, un algorithme a été utilisé pour empêcher le chevauchement des jours pour chaque ration suivante. À l'aide de la courbe de consommation d'aliments, des paramètres de régression ont été calculés dans MicrosoftMC Excel. Une valeur minimale R au carré de plus de 0,99 était nécessaire pour être considérée comme un bon ajustement de la ligne de régression. Afin d'obtenir la ligne de régression la mieux ajustée, la courbe d'alimentation des poulets de chair a été divisée en 3 segments. Au segment 1, ou à la première ligne de régression, les estimations ont permis de calculer la consommation d'aliments lorsque l'âge des oiseaux au début ou à la fin de la ration était inférieur ou égal à 21 jours (c.-à-d., qui correspond à la couvaison et à la période d'engraissement précoce) (tableau 2). Les estimations de la deuxième ligne de régression (segment 2) ont été utilisées lorsque l'âge des oiseaux au début ou à la fin de la ration était supérieur ou égal à 35 jours (c.-à-d., qui correspond à la phase de finition ou à la période d'engraissement prolongée chez les poulets de chair) (tableau 2). La consommation d'aliments pour toutes les autres tranches d'âge était basée sur la représentation de la troisième ligne de régression (c.-à-d., la période d'engraissement) (tableau 2).
Les calculs de consommation d'aliments ont ensuite été réalisés en fonction des coefficients de régression calculés et présentés dans le tableau 2. Pour chaque ration, les coefficients de régression appropriés (basé sur l'âge des oiseaux au début et à la fin) et le nombre de jours au cours desquels la ration leur a été servie (tel qu'il est indiqué dans l'enquête) ont été introduits dans les formules de l'aire sous la courbe (tableau 2). Deux intégrales ont été calculées pour chacune des rations. L'intégrale inférieure « t » correspond au début de la ration et l'intégrale supérieure « t » à la fin de la ration. La différence entre l'intégrale supérieure et inférieure donne l'estimation de la consommation d'aliments en gramme par oiseau pour cette ration. La consommation d'aliments a été convertie de grammes en tonnes et multipliée par le nombre d'oiseaux à risque (c.-à-d., le nombre total d'oiseaux moins la moitié des mortalités) afin de fournir une estimation du nombre total de tonnes servies pour chaque ration. Le nombre d'oiseaux déclarés était le nombre total d'oiseaux livrés au bâtiment avicole concerné (poulailler ou étage) y compris la marge de tolérance de 2 % fournie par le couvoir. Cette valeur a ensuite été utilisée pour calculer les grammes d'antimicrobiens consommés par ration et intégrée à l'analyse quantitative.
| Segment de la courbe de consommation de moulée | Âge des oiseaux en jours | Coefficients de régression calculés | R2 | Calcul de l'aire sous la courbe et de la consommation de moulée | |||
| β0 | β1 | β2 | β3 | ||||
| 1 | ≤ 21 | 14,096 | 1,2095 | 0,228 | -0,003 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| 2 | ≥ 35 | -13,06 | 4,8777 | 0,085 | -0,0017 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| 3 | Tous les autres âges | -27,935 | 8,827 | -0,069 | -5,00E-05 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
Consommation d'eau
Les estimations relatives à la consommation d'eau reposaient sur une régression simple et un calcul intégral. Elles ont été saisies dans Microsoft™ Excel. Les estimations ont été calculées à l'aide d'un graphique représentant la consommation d'eau quotidienneNote de bas de page 16, ce qui a permis de créer une courbe pour la consommation en litres par oiseau par jour.
Le questionnaire sur les poulets de chair indiquait l'âge des oiseaux correspondant au début et à la fin de chaque traitement par l'eau. Un algorithme a été utilisé pour empêcher tout chevauchement possible de l'âge en jours pour les traitements consécutifs par l'eau utilisant différents antimicrobiens au sein d'un même troupeau. À l'aide de la courbe de consommation d'eau, des paramètres de régression ont été calculés dans Microsoft™ Excel. Une valeur minimale R au carré supérieure à 0,99 était nécessaire pour être considérée comme un bon ajustement de la courbe de régression. Afin d'obtenir les valeurs de régression les mieux ajustées, la courbe de consommation d'eau a été divisée en 3 segments. Lorsque l'âge des oiseaux au début et à la fin du traitement par l'eau était inférieur ou égal à 21 jours, la consommation d'eau était calculée en fonction de la ligne de régression correspondant au premier segment de la courbe (tableau 3). Lorsque l'âge des oiseaux au début ou à la fin du traitement par l'eau était inférieur ou égal à 38 jours, la consommation d'eau était calculée en fonction de la ligne de régression correspondant au deuxième segment de la courbe (tableau 3). Pour toutes les autres tranches d'âge, la consommation d'eau a été calculée d'après la ligne de régression correspondant au troisième segment de la courbe. À partir des coefficients de régression, la consommation d'eau pouvait ensuite être calculée à l'aide du calcul intégral et de la formule de l'aire sous la courbe telle qu'elle a été décrite plus haut pour la consommation d'aliments des poulets de chair (tableau 3).
| Segment de la courbe de consommation d'eau | Âge des oiseaux en jours | Coefficients de régression calculés | R2 | Calcul de l'aire sous la courbe et de la consommation d'eau | |||
| β0 | β1 | β2 | β3 | ||||
| 1 | ≤ 21 | 0,0322 | 8,00E-05 | 0,0005 | -7,00E-06 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| 2 | ≥ 38 | 0,0335 | -0,0003 | 0,0005 | -7,00E-06 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| 3 | Tous les autres âges | -0,4475 | 0,0417 | -0,0007 | 4,00E-06 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
Quantité d'antimicrobiens utilisés chez les poulets de chair
D'après les calculs propres aux espèces ci-dessus, les milligrammes d'ingrédient actif ont été obtenus pour chaque voie d'administration, déclarés par voie et l'ensemble de toutes les voies. Pour l'équation 2 à l'équation 4, le nombre total d'animaux correspond à la population du troupeau ou de l'élevage de départ moins la moitié du nombre des mortalités.
Équation 2. Estimation de la quantité totale de milligrammes administrés dans les aliments (poulets de chair, porcs et dindons)
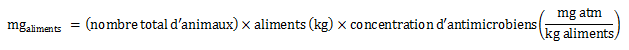
Atm = antimicrobiens.
Équation 2 - Équivalent textuel
La quantité totale des milligrammes dans les aliments est calculée en multipliant le nombre total d'animaux par la quantité d'aliment consommés (en kilogramme) et par la concentration d'antimicrobiens (les milligrammes d'ingrédient actif divisés par les kilogrammes d'aliments).
Équation 3. Estimation du total en milligrammes dans l'eau (poulets de chair, porcs et dindons)

Concentration d'antimicrobiens* = taux d'inclusion indiqué sur l'étiquette x la concentration de l'antimicrobien.
Équation 3 - Équivalent textuel
La quantité totale des milligrammes dans l'eau des volailles est calculée en multipliant le nombre total d'animaux par la quantité d'eau consommée (en litre) et par la concentration d'antimicrobiens* (en milligramme par litre).
La quantité totale des milligrammes dans l'eau des porcs est calculée en additionnant la quantité totale d'antimicrobiens en gramme administrés pendant la durée du traitement.
Équation 4. Estimation de la quantité totale de milligrammes administrés par injection in ovo ou sous‐cutanée au couvoir (poulets de chair, porcs et dindons)

Le nombre total d'animaux se rapporte à la population du troupeau de départ moins la moitié du taux de mortalité signalé au moment de l'échantillonnage, multiplié par la proportion de porcs exposés.
Équation 4 - Équivalent textuel
La quantité totale des milligrammes par injection des volailles est calculée en multipliant le nombre total de poulets de chair par la quantité de milligrammes injectés et par oeuf d'incubation ou par poussin.
La quantité totale des milligrammes par injection des porcs est calculée en additionnant les résultats obtenus par la multiplication du nombre total d'animaux par le résultat de la multiplication de la concentration du médicament (en milligramme par millilitre) par le poids moyen au moment du traitement (en kilogramme) et par le nombre de jours durant lesquels le médicament a été administré.
En fonction de la quantité d'aliments ou d'eau consommée et de la quantité administrée par injection (pour les poulets de chair et les dindons uniquement) qui provenait des calculs ci-dessus, les mesures ou indicateurs d'utilisation d'antimicrobiens suivants ont été présentés :
Milligrammes d'ingrédient actif/population correction unit (mg/PCU): Le nombre total de milligrammes (les injections, les aliments et l'eau combinés pour les poulets de chair et les dindons, et les aliments uniquement pour les porcs) pour chaque antimicrobien/classe et globalement, ajusté pour la population animale (1 cycle d'engraissement) et le poids.
- Étape 1 population correction unit (PCU) ou biomasse (Équation 5): Le PCU a été calculée en multipliant le nombre total d'animaux déclarés dans le questionnaire (correspondant à un cycle d'engraissement; population moins la moitié des animaux morts) par le poids théorique (standardisé) au moment le plus probable du traitement (le poids standard de l'ESVAC de 1 kg pour les poulets de chair, de 6,5 kg pour les dindons et de 65 kg pour les porcs a été utilisé).
- Étape 2 mg/PCU (Équation 6): L'estimation des mg/PCU pour chaque ingrédient actif de l'antimicrobien, compilé ensuite par classe et globalement afin de générer une estimation propre à une année donnée par espèce.
Équation 5. Formule pour le calcul du PCU
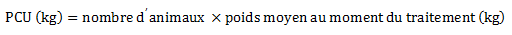
Équation 5 - Équivalent textuel
Le PCU (en kilogramme) est calculé en multipliant le nombre d'animaux par le poids moyen au moment du traitement (en kilogramme).
Équation 6. Formule pour le calcul du mg/PCU
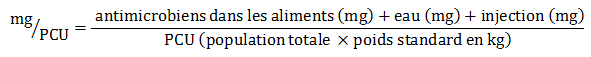
Équation 6 - Équivalent textuel
La mesure des milligrammes par « Population correction unit » (PCU) est calculée en divisant la quantité d'antimicrobiens administrée dans les aliments, dans l'eau et par injection (en mg) par le PCU (population totale multipliée par le poids standard en kilogramme).
Doses définies journalières canadiennes (Defined Daily Doses) selon les doses canadiennes (DDDvetCA) : Les doses journalières moyennes canadiennes de chaque antimicrobien ont été, à quelques exceptions près, attribuées selon une méthodologie semblable à celle des DDDvet de l'ESVACNote de bas de page 17.
- Étape 1 Dose journalière moyenne (équation 7): La dose journalière moyenne a été déterminée comme suit : un DDDvetCA a été attribué à chaque antimicrobien en obtenant toutes les doses approuvées pour les poulets, les porcs et les dindons (aux fins de prévention et de traitement) selon 2 références canadiennesNote de bas de page 18,Note de bas de page 19 ou l'avis d'expert, en l'absence de produit étiqueté (utilisation des médicaments en dérogation des directives de l'étiquette, UMDDE)Note de bas de page 20. La somme de l'ensemble des doses a ensuite été divisée par le nombre total de doses uniques.
- Étape 2 DDDvetCA (équation 8) : Puisque la dose étiquetée (taux d'inclusion) variait selon la forme pharmaceutique (p. ex., g/tonne de produits administrés par les aliments, g/L d'eau pour les produits administrés par l'eau potable, mg/poussin ou œufs d'incubation pour les produits injectables), les valeurs ont été standardisées en mgmédicament/kganimal/jour selon l'approche de l'ESVAC. Comme dans la méthodologie de l'ESVACNote de bas de page 21, un DDDvetCA a été établi pour chaque composant antimicrobien des produits composés. Chez les poulets de chair et les dindons, cela s'applique aux médicaments en association suivants : lincomycine-spectinomycine et triméthoprime-sulfadiazine. Les valeurs pour les porcs et les poulets sont résumées dans le tableau A. 3 et le tableau A. 4. Veuillez noter que l'élaboration des valeurs de mesure est un processus itératif et, par conséquent, les valeurs peuvent changer (p. ex., nouveau produit sur le marché, changements apportés aux étiquettes des produits ou aux allégations approuvées, amélioration des mesures).
Équation 7. Calcul de la dose journalière moyenne
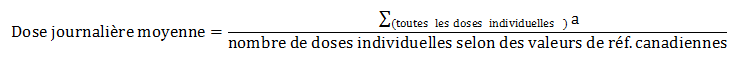
Réf. = référence.
- Équation 7 note a
-
Toutes les doses individuelles indiquées pour le traitement et la prévention et la stimulation de la croissance étaient utilisées pour le calcul de la dose journalière moyenne d'un antimicrobien; un antimicrobien peut avoir plus d'une dose unique par format ou indication du produit.
Équation 7 - Équivalent textuel
La dose journalière moyenne est calculée en additionnant toutes les doses individuelle puis divisées par le nombre de doses individuelles selon des valeurs de référence canadiennes
Équation 8. Standardisation de la dose journalière moyenne pour obtenir le DDDvetCA en mg de médicament par kilogramme de poids corporel (animal) par jour
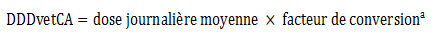
- Équation 8 note a
-
Un facteur de conversion est utilisé pour standardiser l'unité du DDDvetCA en mgmédicament/kganimal/jour comme dans l'approche de l'ESVAC; veuillez consulter les tableaux A. 3 et A. 4 pour les facteurs de conversion propres aux poulets de chair et dindons et aux porcs en croissance-finition, respectivement.
Équation 8 - Équivalent textuel
Le DDDvetCA a été calculé en multipliant la dose journalière moyenne par le facteur de conversion.
Le nDDDvetCA (équation 9): Chaque ingrédient actif de l'antimicrobien et l'ensemble de tous les ingrédients actifs des antimicrobiens (total annuel) font l'objet d'ajustements en fonction de diverses unités techniques de mesure propres à l'espèce (p. ex., la population, le poids, les jours à risque) telles qu'elles sont décrites à l'équation 9 et l'équation 10. Comme pour les mg/PCU, ces indicateurs sont également utilisés pour les comparaisons au fil du temps entre les classes d'antimicrobiens et entre les espèces.
Équation 9. Calcul du nombre de doses journalières en utilisant les standards canadiens (nDDDvetCA)

- Équation 9 note a
-
Il s'agit du numérateur, la combinaison des milligrammes consommés par les aliments par l'eau et administrés par injection.
Équation 9 - Équivalent textuel
Le nDDDvetCA a été calculé en divisant le total des milligrammesa par le DDDvetCA standard en milligrammes par kilogramme par jour.
Nombre de doses journalière définies canadiennes (nDDDvetCA)/1 000 animaux-jours à risque (équation 10) : Également connu sous le nom d'incidence du traitementNote de bas de page 22,Note de bas de page 23,Note de bas de page 24,Note de bas de page 25. Cet indicateur a été calculé en divisant la valeur du nDDDvetCA (équation 9) par la valeur du dénominateur (population du troupeau ou de l'élevage moins la moitié du nombre d'animaux morts multiplié par le poids standard de l'ESVAC et le nombre moyen de jours pour un cycle de production pour les troupeaux ou élevages surveillés). Le nombre de jours à risque est propre à une année donnée (p. ex., 2017 : 34 jours pour les poulets de chair, 114 jours pour les porcs en croissance-finition, 90 jours pour les dindons). La dernière étape consistait à multiplier les valeurs par 1 000. Veuillez noter que l'équation 10 diffère légèrement du Rapport annuel du PICRA de 2016; le calcul ci-dessous a été modifié pour refléter les étapes séquentielles menant à l'indicateur final d'utilisation d'antimicrobien et correspondre à la méthodologie décrite dans la littérature.
Équation 10. Formule pour le nombre de DDDvetCA/1000 animaux-jours à risque
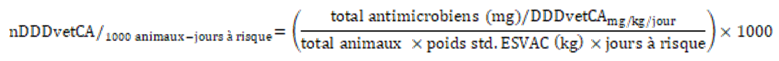
Std. = standard.
Équation 10 - Équivalent textuel
Le nDDDvetCA par 1000 animaux-jours à risque est calculé en divisant la quantité d'antimicrobiens (en milligrammes) par DDDvetCA (en milligrammes par kilogramme par jour) par la valeur obtenue en multipliant le nombre total d'animaux par le poids standard ESVAC (en kilogrammes) et le nombre de jours à risque. Le résultat de cette division est ensuite multiplié par 1 000.
Nombre de doses journalières canadiennes/population correction unit (nDDDvetCA/PCU) (Équation 11) : Cette valeur de mesure a ajusté le nDDDvetCA à la biomasse propre à l'espèce (voir l'étape 2 de l'équation 8) en fonction d'une méthode décrite ailleursNote de bas de page 26.
Équation 11. Formule pour le nombre de DDDvetCA/PCU
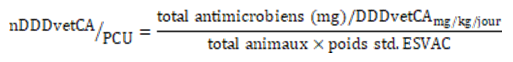
Std. = standard.
Équation 11 - Équivalent textuel
Le nDDDvetCA par le Population correction unit (PCU) est calculé en divisant la quantité d'antimicrobiens (en milligrammes) par DDDvetCA (en milligrammes par kilogramme par jour) par la valeur obtenue en multipliant le nombre total d'animaux par le poids standard ESVAC (en kilogrammes).
Porcs en croissance-finition
L'exposition aux antimicrobiens a été résumée pour chacun des troupeaux. Par exposition, on entend toute utilisation déclarée d'un ingrédient actif par une voie d'administration donnée en 2017. Les données ont été déclarées sous forme d'exposition à un ingrédient actif par une voie d'administration donnée, ainsi que d'exposition à un ingrédient actif par toute voie d'administration. Ces expositions sont résumées par classe d'antimicrobiens. Il est important de noter que les expositions aux antimicrobiens par la moulée touchent généralement un plus grand nombre de porcs pendant une plus longue durée d'utilisation que les expositions aux antimicrobiens par l'eau. Les antimicrobiens injectables sont généralement administrés de façon individuelle à un nombre de porcs limitéNote de bas de page 27.
Consommation d'aliments
Des données quantitatives sur l'utilisation d'antimicrobiens (dose et durée) ont été recueillies pour les antimicrobiens ajoutés aux aliments, mais pas pour ceux ajoutés à l'eau et administrés par injection. La quantité d'antimicrobiens consommés par la moulée a été estimée en multipliant la concentration d'antimicrobiens dans une ration donnée par la somme de tonnes consommées au cours de la période d'exposition. Les estimations de consommation de moulée sont fondées sur des équations de régression simple et le calcul intégral. Des graphiques de la consommation de moulée par jour ont été créés avec le logiciel MicrosoftMC Excel, à l'aide des tableaux du Conseil national de recherches (Nutrient Requirements of Swine : Onzième édition révisée, National Academy of Sciences, 2012) pour les porcs en croissance-finition. Trois graphiques ont été créés pour illustrer le rendement faible (dépôts de protéines par kg de moulée consommée 15 % plus faibles que ceux d'un porc moyen), moyen (porc moyen décrit par le Conseil national de recherches) et élevé (dépôts de protéines 15 % plus élevés que ceux d'un porc moyen). Le plus faible poids de départ observé pour toutes les rations indiquées dans un questionnaire a été choisi et le jour correspondant a été déterminé dans le tableau de consommation de moulée. Le nombre de jours où la ration a été servie a ensuite été ajouté au jour de départ pour obtenir le jour de fin de cette ration. Pour chaque ration successive, le nombre de jours pendant lesquels la ration a été servie a été ajouté au jour de fin de la ration précédente. Lorsque le jour de fin d'alimentation déclaré se situait à l'extérieur du tableau du Conseil national de recherches, les données étaient extrapolées jusqu'à un maximum de 50 jours supplémentaires.
Les paramètres de régression pour chaque niveau de performance du porc ont été calculés dans MicrosoftMC Excel à l'aide de la courbe de consommation d'aliments (tableau 4). Une valeur minimale R au carré supérieure à 0,99 était nécessaire pour être considérée comme un bon ajustement de la courbe de régression. À partir des coefficients de régression, la consommation d'aliments pouvait ensuite être calculée à l'aide du calcul intégral et de la formule de l'aire sous la courbe fournie dans le tableau 4, comparable à celle décrite ci-dessus pour la consommation d'aliments des poulets de chair. Toutefois, pour les porcs, 3 lignes de régression (performances basse, moyenne et haute) ont été créées par ration. Deux intégrales ont été calculées à l'aide de la formule du tableau 4. L'intégrale inférieure, « t » correspond à l'âge du porc au début de la ration et l'intégrale supérieure « t » correspond à l'âge du porc à la fin de la ration. La différence entre l'intégrale supérieure et inférieure donne l'estimation de la consommation d'aliments en kilogrammes par porc pour cette ration. Pour chaque troupeau de porcs en croissance-finition, un gain moyen quotidien (GMQ) a été calculé en fonction des données fournies dans le questionnaire; les poids au début et à la fin ainsi que le nombre de jours pendant lesquels les porcs étaient au stade de production croissance-finition. En utilisant les seuils générés par le partitionnement en tiers des données sur le GMQ présentées dans le questionnaire, les exploitations ont été classées selon la performance des porcs, c'est-à-dire : basse, moyenne ou haute. Les troupeaux à haute performance ont été définis comme des troupeaux dont le GMQ était supérieur à 0,8734, les troupeaux à moyenne performance ont eu un GMQ qui se situait entre 0,8734 et 0,8045 et les troupeaux à basse performance ont eu un GMQ inférieur à 0,8045. D'après ce classement, la ligne de régression et l'intégrale appropriées ont été appliquées pour calculer la consommation d'aliments. La consommation d'aliments a été convertie de grammes en tonnes et multipliée par le nombre de porcs à risque afin de fournir une estimation du nombre total de tonnes servies pour chaque ration. Cette valeur a ensuite été utilisée pour calculer les grammes d'antimicrobiens consommés par ration et intégrée aux analyses quantitatives.
| Performance des porcs | Coefficients de régression calculés | R2 | Calcul de l'aire sous la courbe et de la consommation de moulée | ||
|---|---|---|---|---|---|
| β0 | β1 | β2 | |||
| Basse | 0,901 | 0,0243 | -7,00E-05 | 0,99 | β0t+ β1t2/2+ β2t3/3 |
| Moyenne | 0,8974 | 0,0267 | -9,00E-05 | 0,99 | β0t+ β1t2/2+ β2t3/3 |
| Haute | 0,8945 | 0,0291 | -0,0001 | 0,99 | β0t+ β1t2/2+ β2t3/3 |
Consommation d'eau
La quantité totale de grammes d'ingrédient actif administré pour chaque course de traitement dans l'eau était disponible dans le questionnaire relatif aux porcs en croissance-finition. En obtenant le nombre total de grammes administrés par traitement, il n'était pas nécessaire de calculer la consommation d'eau pour la proportion et la taille des porcs exposés. Pour chaque troupeau, la quantité totale d'UAM dans l'eau a été obtenue en faisant la somme des grammes de l'ingrédient actif, de la classe d'antimicrobiens ou, aux fins d'analyse, de tout antimicrobien utilisé.
Injection
En ce qui a trait l'UAM par injection, la concentration du produit en mg/mL, le nombre de jours de traitement, le poids moyen des porcs au moment du traitement et la proportion de porcs exposés étaient disponibles dans le questionnaire relatif aux porcs en croissance-finition. À partir de ces paramètres, le total en mg d'antimicrobien peut être calculé pour la ferme. Pour chaque troupeau, l'UAM totale par injection a été obtenue en faisant la somme des milligrammes de l'ingrédient actif, de la classe d'antimicrobiens ou, aux fins d'analyse, de tout antimicrobien utilisé.
Quantité d'antimicrobiens utilisée chez les porcs en croissance-finition
Veuillez consulter la section « Quantité d'antimicrobiens utilisée chez les poulets de chair » (voir ci-dessus) pour connaître la quantité d'antimicrobiens utilisée dans les calculs relatifs aux porcs en croissance-finition.
Dindons
Les expositions aux antimicrobiens de l'éclosion à la fin de la croissance ou à l'étape d'échantillonnage en fin de production (environ une semaine avant l'abattage) ont été résumées pour chacun des troupeaux. Par exposition, on entend toute utilisation déclarée d'un ingrédient actif par une voie d'administration donnée. Les données ont été déclarées sous forme d'exposition à un ingrédient actif par une voie d'administration donnée, ainsi que d'exposition à un ingrédient actif par toute voie d'administration. Ces expositions sont résumées par classe d'antimicrobiens.
Consommation d'aliments
Les estimations relatives à la consommation d'aliments reposaient sur une régression simple et un calcul intégral. Les estimations de consommation d'aliments provenant des références les plus récentes dont les normes de performance pour les dindons Aviagen (Nicolas)Note de bas de page 28 et hybridesNote de bas de page 29 ont été enregistrées dans MicrosoftMC Excel. À partir de ces données, la consommation cumulative de moulée a été calculée en utilisant la moyenne des normes relatives à l'alimentation des animaux pour les 2 souches les plus communes de dindons de chair ainsi que les normes définies par les entreprises spécialisées dans la nutrition (c.-à-d., qui ne sont pas propres à la souche) pour les dindons de chair à la naissance. Les calculs de régression ont été effectués pour les dindons de chair, les dindons reproducteurs mâles et les dindons reproducteurs femelles.
La consommation d'aliments a été calculée par ration en utilisant la même méthodologie que celle décrite pour la consommation d'aliments des poulets de chair. Des coefficients de régression distincts ont été calculés pour les dindons de chair, les dindons reproducteurs mâles et les dindons reproducteurs femelles et appliqués de manière appropriée en fonction du choix du marché ciblé qui avait été indiqué dans l'enquête au moment de la saisie des données. Les coefficients de la ligne de régression et les formules de l'aire sous la courbe sont présentés dans le tableau 5.
| Type d'oiseau | Coefficients de régression calculés | R2 | Calcul de l'aire sous la courbe et de la consommation de moulée | |||
|---|---|---|---|---|---|---|
| β0 | β1 | β2 | β3 | |||
| Dindons de chair | -0,1085 | 0,1782 | 0,008 | -0,0003 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| Dindons mâles (reproducteurs) | -0,0545 | 0,1398 | 0,016 | -0,0005 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
| Dindons femelles (reproducteurs) | -0,1424 | 0,2016 | 0,002 | -0,0002 | 0,99 | β0t+ β1t2/2+ β2t3/3+ β3t4/4 |
Consommation d'eau
Les estimations relatives à la consommation d'eau reposaient sur une régression simple et un calcul intégral. Les estimations de consommation d'eau provenant des référencesNote de bas de page 30 les plus récentes ont été enregistrées dans MicrosoftMC Excel, ce qui a permis de créer un graphique représentant la consommation d'eau quotidienne et une courbe de consommation en litres par oiseau par jour.
La consommation d'eau a été calculée par durée de traitement en utilisant la même méthodologie que celle décrite pour la consommation d'eau des poulets de chair. Des lignes de régressions distinctes ont été calculées pour les oiseaux âgés de 13 semaines ou moins et pour ceux âgés de plus de 13 semaines afin d'obtenir la courbe la mieux ajustée. Les coefficients de la ligne de régression et les formules de l'aire sous la courbe sont présentés dans le tableau 6.
| Segment de la courbe de consommation d'eau | Âge des oiseaux en semaines | Coefficients de régression calculés | R2 | Calcul de l'aire sous la courbe et de la consommation d'eau | ||
| β0 | β1 | β2 | ||||
|---|---|---|---|---|---|---|
| 1 | ≤ 13 | -0,0131 | 0,0487 | 0,0019 | 0,99 | β0t+ β1t2/2+ β2t3/3 |
| 2 | > 13 | 0,8922 | 0,0018 | 0,0002 | 0,99 | β0t+ β1t2/2+ β2t3/3 |
Quantité d'antimicrobiens utilisée chez les dindons
Veuillez consulter la section « Quantité d'antimicrobiens utilisée chez les poulets de chair » (voir ci-dessus) pour connaître la quantité d'antimicrobiens utilisée dans les calculs relatifs aux dindons.
Résistance aux antimicrobiens
Surveillance chez les humains
Objectif(s)
La composante sur la Surveillance des isolats cliniques humains du PICRA a pour objectif de fournir une démarche représentative et dont les méthodes sont uniformisées en vue d'effectuer le suivi des variations temporelles de la prévalence de la résistance aux antimicrobiens parmi les isolats humains de Salmonella.
Design de la surveillance
Au Canada, les laboratoires cliniques hospitaliers et privés effectuent la mise en culture des isolats humains de Salmonella. Bien que les cas de maladies à déclaration obligatoire doivent être rapportés, dans le cadre du Système national des maladies à déclaration obligatoire (SNMDO), l'acheminement des isolats de Salmonella au laboratoire de référence provincial est facultatif et de nature passive. Un pourcentage élevé (84 % en 2001)Note de bas de page 31 des isolats de Salmonella est acheminé aux laboratoires provinciaux de santé publique (LPSP), mais ce pourcentage peut varier selon les laboratoires. Le Yukon, les Territoires du Nord-Ouest et le Nunavut, qui n'ont pas de laboratoires équivalents aux LPSP, expédiaient leurs isolats à l'un des LPSP.
Avant 2002, les laboratoires provinciaux de santé publique acheminaient les isolats de Salmonella au Programme des maladies entériques du Laboratoire national de microbiologie (LNM)@Winnipeg de l'Agence de la santé publique du Canada (ASPC) à Winnipeg, au Manitoba, pour des tests de confirmation et de caractérisation des sous-types. Une lettre d'accord, en vertu de laquelle les provinces s'engageaient à acheminer tous leurs isolats de Salmonella (ou un sous‐groupe donné) au LNM@Winnipeg, a été signée en 2002 entre les LPSP et l'ASPC. C'est cet accord qui a officiellement lancé le programme de surveillance.
Afin d'assurer la validité statistique du programme d'échantillonnage, tous les isolats humains de Salmonella (liés ou non à une éclosion) reçus de manière passive de la part des laboratoires provinciaux de santé publique de la Saskatchewan, du Manitoba, du Nouveau- Brunswick, de la Nouvelle-Écosse, de l'Île-du-Prince-Édouard et de Terre-Neuve-et-Labrador ont été envoyés au Laboratoire national de microbiologie. Dans les provinces plus densément peuplées (Colombie-Britannique, Alberta, Ontario et Québec), les laboratoires provinciaux de santé publique envoyaient uniquement les isolats reçus entre le 1er et le 15e jour de chaque mois. Tous les isolats humains de S. Newport et de S. Typhi ont cependant été acheminés au Laboratoire national de microbiologie en raison des craintes de multirésistance dans le premier cas et de l'importance clinique de la bactérie dans le second cas.
On a également demandé aux laboratoires provinciaux de santé publique de chaque province de fournir des données précises sur chaque isolat envoyé, à savoir le nom du sérotype, la date de prélèvement, lieu d'origine de la détection, l'âge du patient, son sexe et sa province de résidence.
Surveillance de la viande vendue au détail
Objectif(s)
La composante Surveillance de la viande vendue au détail du PICRA a pour objectif de fournir des données sur la prévalence de la résistance aux antimicrobiens. Elle vise aussi à effectuer le suivi des variations temporelles de certaines bactéries observées dans la viande crue à l'échelle d'une province ou d'une région.
Design de la surveillance
La composante Surveillance de la viande vendue au détail fournit une mesure de l'exposition humaine à des bactéries résistantes aux antimicrobiens qui résulte de la consommation de viande insuffisamment cuite. L'échantillonnage de la viande vendue au détail représente un maillon logique de la surveillance de la résistance antimicrobienne puisqu'il s'agit de l'étape finale de la production animale. Grâce à la collecte et à l'analyse d'échantillons de viande, la composante de surveillance de la viande vendue au détail permet en outre de mesurer l'exposition humaine aux bactéries résistantes aux antimicrobiens, associée à la consommation de produits à base de viande proposés à la vente aux consommateurs canadiens. L'objet de la surveillance peut être modifié au besoin (p. ex., pour évaluer différents types d'aliments, de bactéries ou de zones géographiques) et la surveillance peut servir de plate-forme de recherche sur des questions précises liées à la résistance aux antimicrobiens dans le secteur agroalimentaire.
En 2017, l'objet de la Surveillance de la viande vendue au détail était l'isolat bactérien cultivé provenant d'une des denrées d'intérêt. Ces denrées étaient des produits de viande crue couramment consommés au Canada provenant de 3 espèces animales ayant fait l'objet d'échantillonnages dans le cadre de la composante Surveillance à l'abattoir de même que de la viande de dindon dont l'échantillonnage a commencé en 2012. Il s'agissait de la viande de poulet (cuisses ou ailes de poulet [avec la peau]), de dindon (viande hachée), de porc (côtelettes) et de bœuf (viande hachée).
Dans le cas du bœuf haché, nous avons procédé au prélèvement systématique d'échantillons de viande extra-maigre, maigre, mi‐maigre et ordinaire pour assurer une bonne représentation de l'hétérogénéité du bœuf haché en fonction de son origine (bœuf produit au pays ou bœuf importé, bovins de boucherie ou vaches laitières de réforme). Les coupes de viande (cuisses ou ailes avec la peau, dindon haché, côtelettes de porc et bœuf) ont été choisies en fonction de la prévalence élevée des espèces bactériennes cibles dans ces denrées et de leur faible coût d'achatNote de bas de page 32 ainsi que de leur facilité de comparaison avec d'autres programmes internationaux de surveillance des produits vendus au détail.
Dans la viande de poulet, les bactéries d'intérêt étaient Campylobacter, Salmonella et E. coli générique et dans le cas de la viande hachée de dindon, les bactéries d'intérêt étaient Salmonella et E. coli générique. La détection de Campylobacter à partir de dindon haché a été interrompue au milieu de l'année 2016 en raison de la faible prévalence observée; l'échantillonnage a donc cessé en 2017. Dans la viande de porc, des isolats de Salmonella et d'E. coli ont été mis en culture, mais seuls les isolats d'E. coli ont été soumis à des tests de sensibilité aux antimicrobiens, pour la surveillance de routine et les rapports annuels. L'isolement de Salmonella a été effectué pour les échantillons de viande de porc, mais surtout en vue d'obtenir une estimation de la prévalence de la bactérie dans ce type de viande pour d'autres programmes de l'Agence de la santé publique du Canada. Étant donné que la prévalence de Salmonella dans la viande de porc est faible, les résultats des tests de sensibilité aux antimicrobiens ne sont pas présentés sur une base annuelle, mais sont regroupés sur une période pluriannuelle aux fins de précision. Dans le cas de la viande de porc, l'isolement de Campylobacter n'a pas été effectué en raison de la faible prévalence observée au cours des premières phases de la Surveillance de la viande vendue au détail. Dans le cas du bœuf, seuls les isolats d'E. coli ont été mis en culture puis soumis à des tests de sensibilité aux antimicrobiens, compte tenu de la faible prévalence de Campylobacter et de Salmonella dans ce type de viande, au détail, observée au cours des premières phases du programme. Pour ce qui est de la viande de dindon, des bactéries Campylobacter, Salmonella et E. coli ont été isolées dans des échantillons de viande vendue au détail.
Méthodes d'échantillonnage
De manière générale, le protocole d'échantillonnage a été conçu pour évaluer la résistance aux antimicrobiens de certaines bactéries qui contaminent la viande vendue au détail et auxquelles les consommateurs canadiens pourraient donc être exposés. En 2017, des échantillons de viande vendue au détail ont été prélevés chaque semaine dans des régions (c.-à-d. des divisions de recensement définies par Statistique Canada) choisies selon un processus de sélection aléatoire et pondérées selon le poids démographique de la région dans chacune des provinces participantes.
En 2017, des échantillons de viande vendue au détail ont ainsi été prélevés en Colombie-Britannique, dans les Prairies (une région regroupant les provinces de la Saskatchewan, de l'Alberta et du ManitobaNote de bas de page 33), en Ontario et au Québec. Contrairement aux années antérieures (2013 et 2014), aucune donnée n'a été présentée au cours des dernières années (2015, 2016 et 2017) pour la région de l'Atlantique (une région comprenant le Nouveau‐Brunswick, la Nouvelle‐Écosse, l'Île‐du‐Prince‐Édouard et Terre-Neuve-et-LabradorNote de bas de page 34), étant donné que les activités d'échantillonnage des aliments vendus au détail dans cette région ont été suspendues en raison de contraintes budgétaires. De plus, en raison de la disponibilité limitée des techniciens effectuant l'échantillonnage, il a uniquement été possible de recueillir des données d'échantillonnage correspondant à une année partielle pour les produits vendus au détail pour l'Ontario en 2017. De ce fait, la cible d'échantillonnage et les objectifs en matière d'identification des isolats subséquents pour cette province n'ont pas été atteints. Par conséquent, toutes les données présentées pour la vente au détail en Ontario en 2017 doivent être interprétées avec prudence.
Les données de Statistique Canada ont été utilisées pour établir les strates. Des quartiles (ou tierciles) de la population totale de la province ont été créés à partir d'une liste des divisions de recensement par province, classifiées en ordre croissant de population. En général, entre 15 et 18 divisions de recensement par province/région ont ensuite été choisies par sélection stratifiée aléatoire et pondérées selon la population de chacune des strates. Le nombre de journées d'échantillonnage attribuées à chacune des strates a également été pondéré selon le poids démographique et le résultat est résumé ci-dessous :
Colombie-Britannique
- Strate 1 : 10 divisions choisies avec 1 journée d'échantillonnage par division et par an
- Strate 2 : 4 divisions choisies avec 3 journées d'échantillonnage par division et par an
- Strate 3 : 1 division choisie avec 20 journées d'échantillonnage par an
Prairies (Alberta seulement en 2017)
- Strate 1 : 9 divisions choisies avec 2 journées d'échantillonnage par division et par an
- Strate 2 : 5 divisions choisies avec 3 journées d'échantillonnage par division et par an
- Strate 3 : 2 divisions choisies avec 5 journées d'échantillonnage par division et par an
- Strate 4 : 1 division choisie avec 7 journées d'échantillonnage par an
Ontario et Québec
- Strate 1 : 10 divisions choisies avec 2 journées d'échantillonnage par division et par an
- Strate 2 : 4 divisions choisies avec 5 journées d'échantillonnage par division et par an
- Strate 3 : 2 divisions choisies avec 10 journées d'échantillonnage par division et par an
- Strate 4 : 1 division choisie avec 20 journées d'échantillonnage par an
De manière générale, en OntarioNote de bas de page 35 et au Québec, ceux qui s'occupaient des échantillonnages ont effectué des prélèvements chaque semaine, tandis qu'en Colombie‐Britannique et dans la région des Prairies, les prélèvements ont été effectués aux 2 semaines. L'échantillonnage a été moins fréquent en Colombie-Britannique et dans la région des Prairies en raison des contraintes budgétaires, de la capacité limitée des laboratoires et afin d'éviter le suréchantillonnage dans certains points de vente au détail. Les échantillons ont été prélevés le lundi ou le mardi et ont été acheminés au laboratoire au plus tard le mercredi. Les échantillons provenant de l'extérieur du Québec (à l'exception des échantillons provenant de la région de l'Atlantique) ont été envoyés au même laboratoire par messagerie, dans les 24 heures.
Dans la plupart des cas pour chaque province, les échantillons ont été prélevés dans 2 divisions de recensement chaque semaine d'échantillonnage. Dans chaque division de recensement, 4 magasins ont été choisis avant la journée d'échantillonnage en fonction de leur catégorie. En général, 3 magasins d'alimentation à succursales multiples et 1 épicerie indépendante ou 1 boucherie étaient choisis pour l'échantillonnage. Une exception a été cependant apportée au protocole. Ainsi, dans les divisions urbaines densément peuplées, comme Toronto et Montréal, on a prélevé des échantillons dans 2 magasins d'alimentation à succursales multiples et 2 épiceries indépendantes ou boucheries, afin de refléter les habitudes présumées d'approvisionnement de ces sous-populations. De manière générale, dans chaque type de magasin, on a tenté de prélever 1 échantillon de chaque type de viande étudiée, pour un total de 15 échantillons de viande (4 de poulet, 4 de dindon, 4 de porc et 3 de bœuf) par division et par journée d'échantillonnageNote de bas de page 36. Dans la mesure du possible, un magasin d'alimentation donné n'était échantillonné qu'une fois par année d'échantillonnage. Dans certains cas, nous n'avons pas pu prélever le nombre d'échantillons voulu, en raison de la disponibilité réduite de certains types de viande, de l'heure de fermeture de certains magasins, etc.
À l'aide d'estimations de la prévalence, les protocoles d'échantillonnage ont été optimisés de manière à obtenir 100 isolats par secteur de production animale, par province, par an (prévision), plus 20 % pour les échantillons perdus ou endommagés. Étant donné que les échantillonnages étaient moins fréquents en Colombie-Britannique et dans la région des Prairies qu'en OntarioNote de bas de page 37 et au Québec, il est possible que l'objectif de prélèvement de 100 isolats par an n'ait pas toujours été atteint dans ces provinces/régions.
Des ordinateurs de type Notebook, munis d'un formulaire de soumission électronique personnalisé, ont été utilisés pour consigner les données suivantes sur les magasins et les échantillons :
- type de magasin.
- nombre de caisses (mesure indirecte du volume de vente du magasin).
- date limite de vente ou date de l'emballage.
- étiquette « Peut contenir de la viande déjà congelée » : oui ou non.
- transformation finale en magasin : oui, non, ne sait pas.
- refroidi à l'air : oui, non, ne sait pas (pour les échantillons de poulet seulement).
- produit biologique : oui, non, ne sait pas.
- sans antibiotiques : oui, non, ne sait pas.
- prix au kilogramme.
Chaque échantillon a été emballé dans un sac doté d'une fermeture à glissière et placé dans une glacière de 16 litres pour le transport. Le nombre de blocs réfrigérants à placer dans chaque glacière a été déterminé en fonction de la température ambiante (c.-à-d. 1 bloc réfrigérant pour les températures inférieures à 20 °C et 2 blocs pour les températures supérieures ou égales à 20 °C). Chaque journée d'échantillonnage, des instruments de mesure de la températureNote de bas de page 38 ont été placés dans 1 ou 2 glacières pour que l'on puisse vérifier la température à laquelle les échantillons étaient exposés.
Surveillance à l'abattoir
Objectif(s)
La composante Surveillance à l'abattoir du PICRA a pour objectif de fournir des données annuelles représentatives à l'échelle nationale sur la résistance aux antimicrobiens des isolats bactériens détectés chez les animaux au moment où ils sont introduits dans la chaîne alimentaire. Ce volet vise également à effectuer le suivi des variations temporelles de la prévalence de la résistance aux antimicrobiens pour ces bactéries.
Design de la surveillance
La composante Surveillance à l'abattoir n'intègre que des animaux provenant d'installations situées au Canada. Mise en place en septembre 2002, cette composante ciblait initialement les bactéries Escherichia coli génériques et Salmonella provenant des animaux destinés à la consommation de viande les plus consommés par habitant : bovins de boucherie, poulets de chair et porcs. Elle a été modifiée en 2003 afin d'éliminer la recherche de Salmonella dans les échantillons provenant de bovins de boucherie, en raison de la faible prévalence de cette bactérie au sein de cette population. La surveillance de Campylobacter chez les bovins de boucherie a commencé à la fin de 2005 afin d'intégrer une surveillance de ce pathogène chez les bovins de boucherie et de fournir des données sur la résistance aux fluoroquinolones, par suite de l'autorisation de l'utilisation d'une fluoroquinolone chez les bovins. La surveillance de Campylobacter a également été lancée chez les poulets en 2010 et chez les porcs en 2012.
Dans le cadre de la Surveillance en abattoir, c'est l'isolat bactérien qui constitue l'objet de la surveillance. Les bactéries d'intérêt sont isolées à partir du contenu cæcal (et non des carcasses) des animaux abattus. On procède ainsi afin d'éviter les problèmes d'interprétation liés à la contamination croisée des carcasses et en vue d'obtenir des résultats plus représentatifs de la résistance aux antimicrobiens qu'on retrouve sur l'exploitation d'où proviennent les animaux.
Plus de 90 % de tous les animaux destinés à l'alimentation humaine au Canada sont abattus chaque année dans des abattoirs inspectés par le fédéralNote de bas de page 39. Le programme est basé sur la participation volontaire des abattoirs inspectés par les autorités fédérales situés dans tout le Canada. La méthode d'échantillonnage mise au point visait, pour l'ensemble du Canada, à obtenir 150 isolats de Salmonella et d'E. coli générique, ainsi que 100 isolats de Campylobacter pour chacune des 3 espèces animales au cours d'une période de 12 mois. Ces chiffres sont en fait un compromis entre une précision statistique acceptable et des coûts abordablesNote de bas de page 40. Le nombre réel d'échantillons à prélever dépend de chaque espèce animale, selon la prévalence prévue des bactéries cæcales pour le secteur de production animale visé. Ainsi, lorsque le nombre d'isolats visé était 150 et que la prévalence prévue d'une espèce bactérienne donnée était de 10 %, 1 500 échantillons devraient être recueillis pour isolement bactérien.
Le plan d'échantillonnage a été effectué en 2 étapes, chaque secteur de production animale ayant été traité séparément. La première étape consiste en une sélection aléatoire des abattoirs inspectés par le fédéral. La probabilité qu'un abattoir soit sélectionné est proportionnelle à son volume d'abattage annuel. La seconde étape consiste en une sélection systématique des animaux sur la chaîne d'abattage, le nombre annuel d'échantillons cæcaux recueillis dans chaque abattoir étant proportionnel au volume d'abattage.
Méthodes d'échantillonnage
Afin de réduire le plus possible les coûts d'expédition des échantillons et de permettre à chaque abattoir de demeurer efficace, le nombre annuel total d'échantillons à recueillir dans chaque abattoir est divisé par 5, ce qui donne le nombre de périodes d'échantillonnage. Pour chaque période d'échantillonnage, 5 à 7 échantillons cæcaux sont prélevés sur 5 jours, selon les disponibilités du personnel de l'abattoir, à la condition que les 5 animaux et les échantillons correspondants proviennent de groupes différents. L'échantillonnage de lots différents est important afin d'assurer la diversité des sources et d'éviter les résultats biaisés liés à une surreprésentation de certains éleveurs. Les périodes d'échantillonnage sont réparties uniformément durant l'année afin d'éviter tout biais qui pourrait être associé à la variation saisonnière de la prévalence bactérienne et aux résultats des tests de sensibilité aux antimicrobiens.
Quarante-quatre abattoirs sous inspection fédérale (5 abattoirs de bovins de boucherie, 26 abattoirs de volailles et 13 abattoirs de porcs) dans tout le Canada ont participé à la Surveillance à l'abattoir du PICRA en 2017. Ces abattoirs représentaient plus de 95 % des bovins, 70 % des poulets et 80 % des porcs en 2017. Les échantillons sont prélevés selon un protocole déjà établi, qui a été modifié de manière à tenir compte de la configuration spécifique de chaque chaîne d'abattage des divers abattoirs. Les protocoles ont été conçus de manière à éviter tout conflit avec les méthodes courantes d'inspection des carcasses, avec les pratiques de chaque établissement en lien avec le Programme d'amélioration de la salubrité des aliments et avec les exigences en matière de santé et de sécurité. Les protocoles permettent par ailleurs d'éviter les risques de contamination croisée. Les échantillons sont prélevés par le personnel de l'établissement, sous la supervision du vétérinaire responsable de l'Agence canadienne d'inspection des aliments.
Surveillance à la ferme
Objectif(s)
La composante Surveillance à la ferme du PICRA a notamment pour objectif de fournir des données sur l'utilisation d'antimicrobiens et sur la résistance à ces derniers. Elle vise également à effectuer le suivi des variations temporelles de la prévalence de la résistance aux antimicrobiens et à étudier les liens entre l'utilisation d'antimicrobiens et la résistance chez les bovins en parc d'engraissement, les poulets de chair, les porcs en croissance-finition et les dindons, ainsi qu'à évaluer les risques pour la santé humaine qui y sont associés.
Design de la surveillance
La Surveillance à la ferme est la troisième composante de surveillance active mise en oeuvre par le PICRA. Avec les composantes de la Surveillance à l'abattoir et de la Surveillance de la viande vendue au détail, ces données permettent de valider l'information recueillie à des étapes clés de la chaîne de production de la ferme à l'assiette. Cette initiative repose sur un cadre de fermes sentinelles. Elle utilise des questionnaires pour recueillir des données démographiques sur les fermes d'élevage, des renseignements sur la santé des animaux et l'utilisation d'antimicrobiens. Les échantillons composites de matières fécales provenant de plusieurs enclos sont prélevés et envoyés à des laboratoires qui isolent les espèces bactériennes présentes et effectuent des tests de sensibilité aux antimicrobiens. Chez les poulets de chair, les bovins en parc d'engraissement et les dindons, les bactéries d'intérêt étaient Campylobacter, Salmonella et E. coli générique alors que chez les porcs en croissance-finition la bactérie d'intérêt était E. coli générique.
Bovins en parc d'engraissement
La composante Surveillance à la ferme du PICRA chez les bovins en parc d'engraissement a été lancée en 2016. Actuellement, l'échantillonnage est uniquement effectué dans le site sentinelle de FoodNet Canada en Alberta mais, à plus long terme, l'objectif est d'en faire un programme à l'échelle nationale. Les bovins en parc d'engraissement sont échantillonnés lorsqu'ils ont presque atteint le poids du marché. Ce stade de production a été choisi en raison de sa proximité avec le consommateur.
Poulets de chair
La composante Surveillance à la ferme du PICRA chez les poulets de chair a été mise en œuvre en avril 2013 dans les 4 provinces productrices de volailles au Canada (Colombie‐Britannique, Alberta, Ontario et Québec). En 2014, grâce à un financement externe reçu de Saskatchewan Agriculture, la Saskatchewan a également participé au programme. La composante Surveillance à la ferme chez les poulets de chair consiste à échantillonner des troupeaux au moins 1 semaine avant leur chargement pour l'abattage (en fin de production). Cette phase de production a été choisie parmi les autres du fait de sa plus grande proximité avec le consommateur. La moitié des troupeaux échantillonnés en cours d'année l'ont également été au moment du placement des poussins afin d'établir le profil de résistance des poussins en début de production et de déterminer le transfert des organismes résistants depuis l'élevage précédent.
Porcs en croissance-finition
La composante porcine de la Surveillance à la ferme du PICRA a été lancée en 2006 dans les 5 principales provinces productrices de porcs au Canada (Alberta, Saskatchewan, Manitoba, Ontario et Québec). L'industrie porcine a été choisie pour piloter la mise en place de l'infrastructure de surveillance en raison, d'une part, de son programme de salubrité à la ferme, l'AQCMD (Assurance qualité canadienne), auquel adhère une très grande partie des éleveurs et, d'autre part, de l'absence en 2006 d'éclosions récentes de maladie animale exotique dans le cheptel porcin au pays, contrairement aux principaux autres produits du bétail. La composante Surveillance à la ferme est axée sur les porcs en croissance‐finition. Les porcs élevés durant cette phase de la production ont été choisis en raison de leur proximité avec le consommateur.
Dindons
En 2016, la composante de la surveillance à la ferme du PICRA en ce qui a trait aux dindons a été amorcée dans les 3 principales provinces de production de volaille au Canada (la Colombie-Britannique, l'Ontario et le Québec). La composante de la surveillance à la ferme des dindons procède à l'échantillonnage des troupeaux au moins une semaine avant qu'ils soient envoyés à l'abattage (c.-à-d., l'étape de fin de production). Cette étape de production a été choisie en raison de sa proximité avec le consommateur par rapport aux autres étapes de production à la ferme.
Méthodes d'échantillonnage
Bovins en parc d'engraissement
Les vétérinaires en médecine bovine, travaillant parmi les parcs d'engraissement dans le site sentinelle FoodNet Canada (FNC) de l'Alberta, ont été choisis à dessein à partir de la liste des vétérinaires en médecine bovine. Les vétérinaires inscrits ont ensuite recruté des troupeaux pour servir de sentinelles dans ce programme de surveillance volontaire. Les parcs d'engraissement inscrits devaient être représentatifs du profil de la clientèle du vétérinaire. Le nombre de troupeaux ciblés pour l'échantillonnage est de 30, ce qui constitue le nombre requis pour le site sentinelle de FNC. Pour préserver l'anonymat des éleveurs participants, le prélèvement des échantillons et la collecte de données ont été effectués par les vétérinaires travaillant en médecine bovine, qui ont ensuite transmis l'information à l'Agence de la santé publique du Canada sous forme codée.
Les parcs d'engraissement ont été visités une fois par année aux fins de collecte d'échantillons et de données. Des échantillons composites de matière fécale ont été prélevés dans 6 enclos de bovins dont le poids était près de celui du marché (idéalement étant mis plus de 120 jours à l'engraissement et pesant plus de 500 kg). Les vétérinaires ont également reçu la consigne de répartir leurs visites d'échantillonnage sur l'année afin de tenir compte des variations saisonnières de la prévalence des pathogènes et des maladies qui pourraient influencer l'utilisation d'antimicrobiens dans les fermes.
Une feuille de sondage d'une page a été jointe à chaque trousse d'échantillonnage afin de recueillir des renseignements pour FNC et le PICRA. Les données demandées pour chaque enclos de bovins échantillonnés comprenaient les jours minimum et maximum d'engraissement, les poids minimum et maximum des bovins dans l'enclos, la capacité moyenne de l'enclos, la capacité du parc d'engraissement et l'inventaire actuel. D'autres renseignements concernant la source d'eau et les traitements de l'eau ont été demandés pour FNC.
Poulets de chair
Suite à l'adhésion volontaire des éleveurs, les vétérinaires travaillant en médecine aviaire ont recruté des troupeaux devant servir de sentinelles dans ce programme national de surveillance. Le nombre de troupeaux sentinelles assigné à chacune des 4 provinces/régions participantes (Colombie-Britannique, région des Prairies [Alberta et Saskatchewan], Ontario et Québec) était proportionnel au nombre total d'éleveurs détenant une part du quota national, sauf pour les sites sentinelles de FoodNet Canada, parmi lesquels au moins 30 troupeaux ont été échantillonnés. En Alberta, les analyses de laboratoire de tous les élevages ont été effectuées par le Laboratoire d'agroalimentaire du ministère de l'agriculture et de la foresterie de l'Alberta. En Saskatchewan, le ministère de l'Agriculture de la Saskatchewan a pris en charge l'intégralité du financement pour 9 troupeaux.
Pour assurer l'anonymat des éleveurs participants, le prélèvement des échantillons et la collecte de données ont été effectués par les vétérinaires travaillant en médecine aviaire, qui ont transmis l'information à l'Agence de la santé publique du Canada (ASPC) sous forme codée. De plus, la Fédération canadienne des couvoirs (FCC) et le Conseil canadien des transformateurs d'œufs et de volailles ont garanti la confidentialité en conservant la clé des codes des couvoirs; seulement l'information codée était connue de l'ASPC.
Les vétérinaires étaient sélectionnés à dessein pour chaque province. Chaque vétérinaire choisissait un nombre préétabli de sites sentinelles proportionnel à son profil de pratique et aux choix disponibles selon les critères d'admissibilité et d'exclusion précis. Pour être admissible, l'exploitation inscrite devait être conforme au manuel Votre propre pouletMC du Programme d'assurance de la salubrité des aliments à la ferme (PASAF) pour les détenteurs de quota de production de poulets de chair (c.‐à‐d. que les poulets de chair sont la principale espèce animale élevée sur le site de production, mais que le producteur peut aussi élever d'autres espèces animales ou produire d'autres denrées). Les exploitations dont les animaux sont exempts d'antibiotiques ou dont les animaux sont élevés sans antibiotiques, de même que les élevages biologiques, étaient aussi sélectionnés selon la proportion qu'ils représentaient dans le profil de la clientèle du vétérinaire. De plus, les vétérinaires s'assuraient que les exploitations choisies étaient représentatives de tous les couvoirs membres de la FCC qui fournissent des poussins, et représentatives également des meuneries qui distribuent des aliments dans leur province d'exercice, et que ces exploitations étaient bien réparties au niveau géographique (c.‐à‐d. éviter les troupeaux voisins). De plus, ces fermes devaient aussi refléter, sur le plan démographique, la pratique vétérinaire et le profil d'ensemble de l'industrie du poulet de chair (p. ex. diversité de la gestion des élevages : troupeaux peu performants à très performants; diversité du volume de poussins placés : densité de population faible à élevée). L'application de ces critères a permis d'assurer la représentativité des troupeaux inscrits par rapport à l'ensemble des troupeaux de poulets de chair au Canada. Les vétérinaires ont également reçu la consigne de répartir leurs visites d'échantillonnage sur l'année afin de tenir compte des variations saisonnières de la prévalence des pathogènes et des maladies qui pourraient influencer l'utilisation d'antimicrobiens dans les couvoirs et dans les fermes.
Une fois par année, les troupeaux sentinelles de poulets de chair ont été visités pour la collecte d'échantillons et de données au cours de la dernière semaine de croissance (poulets âgés de plus de 30 jours). Quatre échantillons composites de matières fécales, représentant chacun un quart de la surface du plancher et constitués d'au moins 10 fientes, ont été ramassés dans des bâtiments et des niveaux (dans le cas des bâtiments à plusieurs étages/enclos) sélectionnés de manière aléatoire. À titre d'essai, une proportion des troupeaux a aussi été visitée à l'arrivée des poussins dans le bâtiment. Au moyen d'une éponge stérile, 2 échantillons de l'environnement (surfaces du bâtiment) et 3 échantillons de méconium ont été prélevés. Les échantillons de méconium ont été pris à partir des papiers absorbants mis au fond des caisses qui sont utilisées pour transporter les poussins du couvoir à la ferme.
Porcs en croissance-finition
Suite à l'adhésion volontaire des éleveurs, les vétérinaires travaillant en médecine porcine ont recruté des troupeaux devant servir de sentinelles dans ce programme national de surveillance. Le nombre de troupeaux sentinelles attribués à chacune des 5 provinces participantes était proportionnel au nombre total d'exploitations de porcs en croissance-finition à l'échelle nationale, sauf en Saskatchewan, où 3 troupeaux sentinelles supplémentaires ont été inclus. Le ministère de l'Agriculture de la Saskatchewan a financé les coûts associés aux 3 troupeaux additionnels.
Pour préserver l'anonymat des éleveurs participants, le prélèvement des échantillons et la collecte de données ont été effectués par les vétérinaires travaillant en production porcine, qui ont transmis l'information à l'ASPC sous forme codée. Dans le cas des troupeaux appartenant à des intégrateurs, des vétérinaires superviseurs de pratique privée ont veillé à la confidentialité des données en conservant la clé des codes de ces troupeaux. Cette disposition a été prise, car en ayant accès au nom du vétérinaire qui travaille pour un intégrateur, on peut savoir de quelle entreprise il s'agit, ce qui briserait l'anonymat.
Tous les vétérinaires travaillant en médecine porcine dans chacune des provinces sont éligibles au programme. Chaque vétérinaire choisit un nombre préétabli de sites sentinelles en se basant sur les critères d'admissibilité et d'exclusion du programme. Pour être admissible, l'exploitation inscrite doit être accréditée en vertu du programme AQCMD, produire plus de 2000 porcs de marché par année et être représentative des caractéristiques (c.-à-d. volumes de production et systèmes de production similaires) et de la répartition géographique des troupeaux clients du vétérinaire participant. Ne sont pas admissibles les troupeaux élevés selon des pratiques d'élevage considérées comme biologiques, les troupeaux qui consomment des matières résiduelles comestibles ou ceux qui sont élevés en pâturage. L'application de ces critères permet de s'assurer que les troupeaux inscrits sont représentatifs de l'ensemble des troupeaux de porcs en croissance-finition au Canada.
Les troupeaux sentinelles de porcs en croissance-finition ont été visités une fois par an aux fins de collecte d'échantillons et de données. Des échantillons composites de matière fécale ont été prélevés dans 6 enclos de porcs en fin de lot (c.-à-d. pesant plus de 80 kg [175 lb]). Les vétérinaires ont reçu la consigne de répartir leurs visites d'échantillonnage sur l'année afin de tenir compte des variations saisonnières de la prévalence des pathogènes et des maladies qui pourraient influencer l'utilisation d'antimicrobiens dans les fermes.
Dindons
Les vétérinaires travaillant en médecine aviaire ont recruté des troupeaux pour servir de sentinelles dans ce programme national de surveillance volontaire. Le nombre de troupeaux attribué à chacune des 3 provinces ou régions participantes (Colombie-Britannique, Ontario et Québec) était proportionnel au nombre total d'éleveurs détenant une part du quota national, sauf pour les sites sentinelles de FoodNet Canada, parmi lesquels au moins 30 troupeaux ont été échantillonnés.
Pour assurer l'anonymat des éleveurs participants, le prélèvement des échantillons et la collecte de données ont été effectués par les vétérinaires travaillant en médecine aviaire, qui ont transmis l'information à l'Agence de la santé publique du Canada (ASPC) sous forme codée. La Fédération canadienne des couvoirs (FCC) et le Conseil canadien des transformateurs d'œufs et de volailles (CCTOV) ont garanti la confidentialité en conservant la clé des codes des couvoirs; seulement l'information codée était connue de l'ASPC.
Les vétérinaires travaillant en médecine aviaire étaient sélectionnés à dessein pour chaque province. Chaque vétérinaire choisissait un nombre préétabli de sites sentinelles proportionnel à son profil de pratique et aux choix disponibles selon des critères d'admissibilité et d'exclusion précis. Pour être admissibles, les exploitations devaient être conformes aux exigences du Programme de salubrité des aliments à la ferme (PSAF) des éleveurs de dindons pour les détenteurs de quota de production de dindons de chair (c.‐à‐d. que les dindons de chair sont la principale espèce animale élevée sur le site de production, mais que le producteur peut aussi élever d'autres espèces animales ou produire d'autres denrées). Les exploitations dont les animaux sont exempts d'antibiotiques ou dont les animaux sont élevés sans antibiotiques, de même que les élevages biologiques, étaient aussi sélectionnés selon la proportion qu'ils représentaient dans le profil de la clientèle du vétérinaire. Les vétérinaires s'assuraient aussi que les exploitations choisies étaient représentatives de tous les couvoirs membres de la FCC qui fournissent des dindonneaux, et représentatives des meuneries qui distribuent des aliments dans leur province d'exercice, tout en s'assurant que ces exploitations étaient bien réparties au niveau géographique (c.‐à‐d. éviter les troupeaux voisins). De plus, ces fermes devaient aussi refléter, sur le plan démographique, la pratique vétérinaire et le profil d'ensemble de l'industrie du dindon de chair (p. ex. diversité de la gestion des élevages : troupeaux peu performants à très performants; diversité du volume de dindonneaux placés : densité de population faible à élevée). L'application de ces critères a permis d'assurer la représentativité des troupeaux inscrits par rapport à l'ensemble des troupeaux de dindons de chair au Canada. Les vétérinaires ont également reçu la consigne de répartir leurs visites d'échantillonnage sur l'année afin de tenir compte des variations saisonnières de la prévalence des pathogènes et des maladies qui pourraient influencer l'utilisation d'antimicrobiens dans les couvoirs et dans les fermes.
Une fois par année, les troupeaux sentinelles de dindons de chair ont été visités pour la collecte d'échantillons et de données au cours de la dernière semaine de croissance (pour les dindons, la visite a eu lieu au cours de la dernière semaine selon les catégories de poids du marché : dindons de chair, dindons femelles légères, dindons femelles lourdes, dindons mâles légers et dindons mâles lourds). Quatre échantillons composites de matières fécales, représentant chacun un quart de la surface du plancher et constitués d'au moins 10 fientes, ont été ramassés dans des bâtiments et des étages (dans le cas des bâtiments à plusieurs étages/enclos) sélectionnés de manière aléatoire.
Surveillance des isolats cliniques animaux
Objectif(s)
La composante Surveillance des isolats cliniques animaux a pour objectif de détecter les profils émergents de résistance aux antimicrobiens et de mettre en évidence de nouvelles associations sérotype/profil de résistance chez les isolats de Salmonella.
Design de la surveillance
Cette composante du PICRA est basée sur le prélèvement par les vétérinaires ou les éleveurs et l'envoi d'échantillons aux laboratoires de diagnostic vétérinaires. Les méthodes de prélèvement et d'envoi, ainsi que les techniques d'isolement de Salmonella, ont donc pu varier entre les laboratoires en cours d'année.
Les isolats de Salmonella ont été envoyés par les laboratoires provinciaux et privés de santé animale de tout le pays au Laboratoire de référence de Salmonella (LRS) du Laboratoire national de microbiologie (LNM)@Guelph. Par contre, les isolats du Québec ont été envoyés au Laboratoire d'épidémiosurveillance animale du Québec, du ministère de l'Agriculture, des Pêcheries et de l'Alimentation du Québec aux fins du sérotypage. Les isolats ainsi que les résultats du sérotypage de S. Enteritidis et de S. Typhimurium provenant du Québec ont ensuite été transmis au LNM@Guelph, afin d'effectuer des tests de lysotypage et de sensibilité aux antimicrobiens. Les isolats du Québec qui n'appartenaient pas aux espèces S. Enteritidis ou S. Typhimurium ont également été sérotypés au LNM@Guelph. Il est à noter que tous les isolats reçus par les laboratoires provinciaux de santé animale ne sont pas forcément envoyés au LNM@Guelph, à l'exception de ceux reçus par les laboratoires provinciaux de santé animale de la Colombie-Britannique, de l'Ontario, du Québec et de l'Île-du-Prince-Édouard. La répartition des isolats a donc pu varier considérablement entre les provinces.
Certains échantillons prélevés aux fins d'analyse peuvent avoir été recueillis sur un animal malade, dans des aliments pour animaux, dans l'environnement de l'animal ou sur des animaux non malades du même troupeau ou lot. Les résultats présentés concernent les poulets, les dindons, les bovins, les porcs et les chevaux. Les échantillons de bovins pouvaient provenir de bovins laitiers, de veaux de lait ou de grain ou encore de bovins de boucherie. Les isolats de poulets provenaient principalement de poules pondeuses ou de poulets de chair, mais pouvaient également provenir d'oiseaux reproducteurs pour des lignées de pondeuses ou de poulets de chair. Une partie des isolats provenant des dindons pouvaient provenir d'échantillons de l'environnement de ces oiseaux.
Aliments et ingrédients des aliments pour animaux
Design d'échantillonnage
Les données de la composante Aliments et ingrédients des aliments pour animaux du PICRA proviennent de sources variées, dont les programmes de surveillance de l'Agence canadienne d'inspection des aliments (ACIA), et quelques isolats viennent des autorités provinciales.
L'ACIA prélève des échantillons d'aliments pour le bétail dans le cadre de 2 programmes distincts : le programme 15A (Inspection de suivi : Salmonella) et le programme 15E (Inspection dirigée : Salmonella). En vertu du programme 15A, des échantillons d'aliments du bétail fabriqués dans des meuneries, des usines d'équarrissage, par des fabricants d'ingrédients et dans des exploitations d'élevage sont prélevés et mis en culture à la recherche de Salmonella. Bien que ce programme fasse appel à un processus d'échantillonnage aléatoire, une attention particulière est portée aux aliments pour le bétail qui sont susceptibles de présenter un niveau élevé de contamination par Salmonella, comme les produits animaux d'équarrissage, les tourteaux d'oléagineux, la farine de poisson, les céréales et les purées. Le programme 15E vise les aliments et les ingrédients pour le bétail provenant des établissements qui :
- fabriquent des produits animaux d'équarrissage, ou d'autres aliments contenant des ingrédients susceptibles d'être contaminés par Salmonella (p. ex. les tourteaux d'oléagineux ou la farine de poisson) ou un volume important d'aliments pour la volaille.
- sont réputés pour être fréquemment associés à des contaminations par Salmonella.
- sont associés à la présence d'un sérotype de Salmonella très pathogène (p. ex. Typhimurium, Enteritidis ou Newport).
Le programme 15E est un programme ciblé, c'est-à-dire que les échantillons ne sont pas choisis de manière aléatoire.
Méthodes d'isolement des bactéries
Tous les échantillons ont été mis en culture à l'aide des protocoles normalisés décrits ci-dessous. Dans tous les cas, l'isolement primaire des souches humaines de Salmonella a été effectué par un laboratoire hospitalier ou un laboratoire de clinique privée dans les provinces participantes. Dans la plupart des cas, l'isolement primaire d'Escherichia coli, de Salmonella et de Campylobacter provenant des échantillons du secteur agroalimentaire a été effectué par le Laboratoire national de microbiologie de Saint-Hyacinthe. Une partie de l'isolement primaire fait dans le cadre de la Surveillance à la ferme a été effectuée au Laboratoire d'agroalimentaire du ministère de l'agriculture et de la foresterie de l'Alberta. Les échantillons prélevés dans le cadre de la composante Surveillance des isolats cliniques animaux du PICRA ont été mis en culture dans divers laboratoires participants. La majorité de l'isolement primaire des échantillons recueillis dans le cadre de la composante Aliments et ingrédients des aliments pour animaux a été effectuée par la Division des services laboratoires de l'ACIA (à Calgary ou à Ottawa).
Salmonella
Surveillance des isolats cliniques humains
Les laboratoires d'hôpitaux et de cliniques privées ont isolé et identifié les Salmonella provenant d'échantillons prélevés chez des humains au moyen de méthodes approuvéesNote de bas de page 41,Note de bas de page 42,Note de bas de page 43,Note de bas de page 44.
Surveillance des isolats du secteur agroalimentaire (Surveillance de la viande vendue au détail, Surveillance à l'abattoir et Surveillance à la ferme)
Les bactéries Salmonella ont été isolées avec une modification de la méthode MFLP-75Note de bas de page 45. Cette méthode a permis d'isoler des Salmonella viables et mobiles à partir de matières fécales (Surveillance à la ferme), du contenu caecal (Surveillance à l'abattoir) et de viandes (Surveillance de la viande vendue au détail) d'échantillons du secteur agroalimentaire. Elle est basée sur la capacité de Salmonella à se multiplier et à être mobile dans un milieu semi-solide modifié de Rappaport Vassiliadis (MSRV), à une température de 42 °C.
Surveillance de la viande vendue au détail : Selon l'échantillon en cause, on a placé 1 cuisse de pouletNote de bas de page 46, 1 côtelette de porc ou 25 g de dindon haché dans 225 mL d'eau peptonée tamponnée (EPT). Cent millilitres de ce liquide de trempage ont été conservés pour l'isolement de Campylobacter ou d'E. coli. Les échantillons de poulet et de dindon ont été laissés dans le volume restant et incubés à 35 ± 1 °C, pendant 24 heures. Ensuite, 0,1 mL de la solution de rinçage a été inoculé sur une gélose MSRV qui a ensuite été incubée à 42 ± 1 °C pendant 24 à 72 heures. La migration égale ou supérieure à 20 mm a ensuite été étendue par strie sur gélose MacConkey. Les colonies isolées ont été purifiées, puis elles ont été inoculées sur une gélose inclinée aux trois sucres et au fer ainsi que sur une gélose en pente à l'urée. Les isolats présumés de Salmonella ont fait l'objet du test de l'indole, puis ont été vérifiés au moyen du test d'agglutination sur lame à l'aide d'antisérum Poly A-I et de Vi anti-Salmonella.
Surveillance à l'abattoir et Surveillance à la ferme : une quantité de 25 g d'échantillons de matière fécale ou de contenu caecal de porcs, combinés aux échantillons de matière fécale ou de contenu caecal de poulets de chair, ont été mélangés avec 225 mL d'EPT. Le contenu caecal/l'échantillon de matière fécale des poulets a été pesé et dilué au dixième dans de l'EPT. Les éponges provenant des échantillons environnementaux et du méconium des poussins ont été mélangées à 100 mL de l'EPT. Les échantillons ont été incubés à 35 ± 1 °C, pendant 24 heures. Ensuite, la méthode utilisée est celle décrite dans la section Salmonella, Surveillance de la viande vendue au détail.
Surveillance des isolats cliniques animaux
L'isolement de Salmonella a été effectué par les laboratoires participants à l'aide de leurs méthodes normalisées, lesquelles varient d'un laboratoire à l'autre. La plupart des méthodes utilisées pour la recherche de Salmonella dans les isolats cliniques animaux sont basées sur des principes similaires et utilisent un bouillon de pré-enrichissement, un bouillon d'enrichissement sélectif, des géloses différentielles et sélectives, l'isolement bactérien; l'identification des isolats est confirmée à l'aide de tests biochimiques et sérologiques.
Aliments et ingrédients des aliments pour animaux
Dans le cadre des 2 programmes de l'Agence canadienne d'inspection des aliments (15A et 15E), tous les échantillons ont été prélevés selon des méthodes aseptiques à des fins de culture et d'isolement bactériens. L'isolement de Salmonella a été fait sur gélose MSRV.
Escherichia coli
Surveillance de la viande vendue au détail
Cinquante millilitres de l'eau peptonée de trempage des échantillons préparée selon la méthode décrite dans la section Salmonella Surveillance de la viande vendue au détail ont été mélangés avec 50 mL de bouillon EC à double concentration, et le tout a été incubé pendant 24 heures, à 42 ± 1 °C. Une anse du mélange incubé a été étalée sur une gélose éosine-bleu de méthylène, laquelle a ensuite été incubée pendant 24 heures à 35 ± 1 °C. Les colonies isolées ont été purifiées, après quoi elles ont été repiquées sur des géloses trypticase soja contenant du sang de mouton à 5 %. Les colonies présumées d'E. coli ont été soumises aux tests de l'indole et du citrate de Simmons. Les isolats d'E. coli qui étaient négatifs au test de l'indole ont été identifiés à l'aide d'une trousse d'identification commercialeNote de bas de page 47.
Surveillance à l'abattoir et Surveillance à la ferme
Une goutte du mélange d'eau peptonée préparé selon les explications de la section Salmonella pour la Surveillance des isolats du secteur agroalimentaire en ce qui a trait à la Surveillance à l'abattoir et à la ferme a été étendue par stries sur gélose MacConkey, qui a été incubée pendant 18 à 24 heures à 35 ± 1 °C. Les colonies qui fermentaient le lactose ont été purifiées, puis elles ont été repiquées sur gélose Luria-Bertani. Les colonies présumées d'E. coli ont été identifiées par la méthode décrite dans la section E. coli pour la Surveillance de la viande vendue au détail.
Campylobacter
Surveillance de la viande vendue au détail
Cinquante millilitres de l'eau peptonée de trempage des échantillons, préparée selon la méthode décrite dans la section Salmonella pour la Surveillance de la viande vendue au détail, ont été mélangés avec 50 mL de bouillon Bolton à double concentration, et le tout a été incubé pendant 44 à 48 heures, à 42 ± 1 °C. À l'aide d'un écouvillon saturé de bouillon, on a ensemencé en stries une gélose modifiée au charbon, à la céfoperazone et au désoxycholate (mCCDA), laquelle a été incubée en microaérophilie, pendant 24 à 72 heures, à 42 ± 1 °C. Les colonies suspectes ont été étendues par stries une seconde fois sur une gélose mCCDA puis incubées. À partir de la seconde gélose mCCDA, une colonie a ensuite été étendue par stries sur une gélose Mueller Hinton contenant du sang de mouton citraté. Cette gélose a ensuite été incubée en microaérophilie à 42 ± 1 °C pendant 24 à 48 heures. L'identification des colonies présumées de Campylobacter a été confirmée au moyen des tests suivants : coloration de Gram, production d'oxydase et de catalase. L'espèce a ensuite été déterminée par PCR multiplexe (PCRm)Note de bas de page 48. Des cibles génomiques précises (gène codant l'hippuricase pour C. jejuni et celui codant l'aspartokinase pour C. coli) ont été amplifiées par PCRm des lysats bactériens. Les produits ont été visualisés sur un gel d'agarose et identifiés d'après leur taille moléculaire au moyen de la méthode QIAxcel®Note de bas de page 49. Un témoin universel interne (ARNr 16S) a été intégré à la PCR. Les oligonucléotides qui ont servi d'amorces dans la PCR étaient très spécifiques pour C. jejuni ou C. coli et n'amplifient pas l'ADN des autres espèces de Campylobacter ni celui des genres autres que Campylobacter. Dans le rapport du PICRA, lorsqu'il est question d'« autres espèces de Campylobacter », il s'agit d'organismes du genre Campylobacter dont l'espèce n'a pas été déterminée. Quand le terme « Campylobacter » est utilisé seul, toutefois, il renvoie à toutes les espèces du genre Campylobacter.
Surveillance à l'abattoir et Surveillance à la ferme
Un millilitre du mélange de l'EPT des échantillons préparés, selon la méthode décrite dans les sections relatives à Salmonella pour la Surveillance à l'abattoir et à la ferme, a été mélangé à 9 mL de bouillon d'enrichissement de Hunt, et le tout a été incubé pendant 4 heures, à 35 ± 1 °C. Après cette première incubation, 36 µL de céfopérazone stérile ont été ajoutés aux tubes de bouillon d'enrichissement de Hunt, qui ont été replacés en microaérophilie, à 42 ± 1 °C, pendant 20 à 24 heures. Un écouvillon saturé de bouillon d'enrichissement de Hunt a ensuite servi à ensemencer une gélose mCCDA, qui a été incubée en microaérophilie à 42 ± 1 °C, pendant 24 à 72 heures. Les colonies présumées de Campylobacter ont été identifiées comme on l'indique dans la section Campylobacter pour la Surveillance de la viande vendue au détail.
Méthodes de sérotypage et de lysotypage
Salmonella
Surveillance des isolats cliniques humains
De manière générale, les laboratoires cliniques envoient leurs isolats de Salmonella à leur laboratoire provincial de santé publique (LPSP) respectif pour l'identification et le sérotypage. Les LPSP acheminent à leur tour les isolats de Salmonella au Laboratoire national de microbiologie (LNM) de Winnipeg, selon un protocole d'analyse préétabli. Le Laboratoire national de microbiologie de Winnipeg confirme l'identification des isolats reçus si le sérotype n'a pas été déterminéNote de bas de page 50 ou si le lysotypage n'a pas donné de résultats concluants. Les antigènes O (somatiques) des isolats de Salmonella ont été mis en évidence par la méthode d'agglutination sur lameNote de bas de page 51. Au Laboratoire national de microbiologie de Winnipeg, les antigènes H (flagellaires) de Salmonella ont été déterminés par agglutination sur lame et confirmés par agglutination en tube. Les isolats de Salmonella ont été conservés à la température ambiante (de 25 à 35 °C) jusqu'au typage.
Le LNM de Winnipeg a procédé au lysotypage des isolats des sérotypes de Salmonella suivants : Enteritidis, Heidelberg, Typhimurium, Hadar, Newport, Typhi, Paratyphi BNote de bas de page 52, Paratyphi B var. L(+) tartrate (+), Infantis, Thompson, Oranienburg, Panama, 4,[5],12:b:- et 4,[5],12:i:-. La technique courante décrite par Anderson et WilliamsNote de bas de page 53 a été employée pour le lysotypage. Les isolats ont été étendus par stries sur des géloses nutritives, qui ont été incubées pendant 18 heures, à 37 °C. De 3 à 5 colonies lisses ont été choisies pour inoculer 4,5 mL de bouillon pour bactériophagesNote de bas de page 54, et le tout a été incubé de 1,5 à 2 heures dans un bain-marie agitateur à 37 °C, jusqu'à ce que la turbidité du bouillon soit équivalente à l'étalon 1 de McFarland. Environ 2 mL du milieu de culture ayant atteint la turbidité voulue ont été versés sur des gélosesNote de bas de page 55, puis l'excès de liquide a été aspiré avec une pipette Pasteur. On a laissé sécher les géloses, rincées pendant 15 minutes à la température ambiante, avant d'inoculer environ 10 µL de chaque bactériophage spécifique à chaque sérotype sur les tapis bactériens, à l'aide d'une méthode d'inoculation multiple par seringueNote de bas de page 56. Les géloses ont ensuite été incubées toute la nuit à 37 °C, après quoi les profils de lyse ont été examinésNote de bas de page 57.
Les souches de Salmonella Enteritidis ont été lysotypées avec des bactériophages obtenus de l'International Centre for Enteric Phage Typing (ICEPT), du Laboratoire central de santé publique de Colindale, au Royaume-UniNote de bas de page 58. Le protocole de lysotypage mis au point par CallowNote de bas de page 59, perfectionné par AndersonNote de bas de page 60, puis par Anderson et ses collaborateursNote de bas de page 61, ainsi que les bactériophages utilisés pour Salmonella Typhimurium ont été fournis par l'ICEPT. Le protocole de lysotypage de S. Heidelberg et les bactériophages ont été fournis par le Laboratoire national de microbiologie de WinnipegNote de bas de page 62. Les isolats qui ont réagi aux bactériophages sans toutefois correspondre à aucun des lysotypes reconnus ont été classés comme atypiques. Les souches qui n'ont réagi avec aucun des bactériophages ont été classées comme non typables.
L'unité du LNM@Winnipeg affectée à l'identification et au sérotypage et l'unité affectée au lysotypage sont certifiées à la norme ISO (Organisation internationale de normalisation) 17025 du Conseil canadien des normes. Ces unités d'identification et de sérotypage, de lysotypage et de détermination de la résistance aux antimicrobiens participent annuellement au réseau du Global Food-borne Infections (WHO-GFN), au système de contrôle externe de la qualité de l'Organisation mondiale de la Santé (External Quality Assurance System), au programme de contrôle de Salmonella d'Enter-Net (un réseau de surveillance européen des infections gastro-intestinales chez les humains) ainsi qu'à l'échange de souches avec le LNM@Guelph et LNM@Saint-Hyacinthe (pour Salmonella et Escherichia coli). Le LNM@Winnipeg et le Centre des maladies infectieuses d'origine alimentaire, environnementale et zoonotique participent depuis 2002 à titre de membres à la planification stratégique du programme du WHO-GFN.
Surveillance des isolats du secteur agroalimentaire, des isolats cliniques animaux et des isolats provenant des aliments pour animaux
Le sérotypage des isolats cliniques animaux de Salmonella provenant du Québec a été effectué au Laboratoire d'épidémiosurveillance animale du Québec, du ministère de l'Agriculture, des Pêcheries et de l'Alimentation du Québec (MAPAQ); les isolats ont ensuite été envoyés au Laboratoire de référence des Salmonelloses (LRS) du LNM@GuelphNote de bas de page 63 (anciennement connu sous le nom de Laboratoire des zoonoses d'origine alimentaire). Le sérotypage des isolats a été refait à Guelph par le Laboratoire de référence des Salmonelloses (LRS) de l'OIE à l'exception des isolats présentant certaines problématiques. Avant septembre 2017, le lysotypage a été effectué par le LRS sur les isolats d'Enteritidis, d'Heidelberg et de Typhimurium. Tous les échantillons soumis après le 28 septembre 2017 ont cessé d'être lysotypés. Tous les autres isolats de Salmonella testés dans le cadre du PICRA, en incluant les isolats cliniques provenant des autres provinces, ont été envoyés au LRS aux fins de sérotypage.
Le sérotypage des isolats du PICRA a été effectué à l'aide de la méthode conventionelle de sérotypage phénotypique et d'une méthode alternative basée sur les tests d'ADN appelée Salmonella GenoSerotyping Array (SGSA)Note de bas de page 64. De plus, les échantillons communs à FoodNet Canada et au PICRA, ont été séquencés en utilisant la plateforme MiSeq d'IlluminaMD; la prédiction du sérotype a été déterminée au moyen de la méthode SISTR (Salmonella in silico Typing Resource). La méthode de sérotypage phénotypique permet la détection des antigènes O, ou antigènes somatiques, des isolats de Salmonella au moyen de l'agglutination sur lameNote de bas de page 65. Les antigènes H ou, antigènes flagellaires, ont été identifiés par la technique de précipitation sur microplaquesNote de bas de page 66. Les formules antigéniques et les sérotypes des isolats de Salmonella ont été déterminés selon le système de White-Kauffmaan-Le Minor (WKL)Note de bas de page 67. Le SGSA détecte les gènes codant pour les antigènes O et H de surface et identifie le sérotype de Salmonella correspondant et ce, conformément au système de WKL existant.
La technique courante de Anderson et WilliamsNote de bas de page 68 susmentionnée a été employée pour le lysotypage. Avant septembre 2017, le lysotypage des isolats de Salmonella Enteritidis, Typhimurium et Heidelberg, les bactériophages utilisés pour ces 3 sérotypes étaient les mêmes que ceux utilisés pour la Surveillance des isolats cliniques humains.
À l'exception du séquençage du génome entier et de la prédiction des sérotypes in silico par la méthode SISTR, le LRS détient l'accréditation ISO 17025 par le Conseil canadien des normes. Le LRS participe à des échanges annuels interlaboratoires de panel de tests de sérotypage avec 2 autres laboratoires ainsi qu'au programme de contrôle External Quality Assurance System (système de contrôle externe de la qualité) de l'Organisation mondiale de la Santé.
Méthodologies des tests de sensibilité aux antimicrobiens
Le Laboratoire national de microbiologie (LNM)@Winnipeg a vérifié la sensibilité aux antimicrobiens de tous les isolats humains de Salmonella, et le LNM@Guelph, tous les isolats de Salmonella du secteur agroalimentaire et du secteur des aliments pour animaux. La majorité des isolats de Campylobacter et d'Escherichia coli du secteur agroalimentaire ont été analysés au LNM@Saint-Hyacinthe. Un seul isolat par échantillon positif a fait l'objet de tests de sensibilité aux antimicrobiens.
Les 3 laboratoires ont reçu l'accréditation ISO/IEC 17025 pour le test de sensibilité aux antimicrobiens. Le LNM@Winnipeg est un Centre collaborateur de l'Organisation mondiale de la Santé concernant la préparation et l'intervention, relativement aux pathogènes entériques et à leur résistance aux antimicrobiens. Le laboratoire du LNM@Guelph participe à des programmes de contrôle externes sur les tests de sensibilité aux antimicrobiens de Salmonella. Le laboratoire du LNM@Saint-Hyacinthe participe à des programmes inter-agence avec le Antimicrobial Resistance Monitoring System (NARMS) des États-Unis sur l'identification et les tests de sensibilité aux antimicrobiens de Salmonella, d'E. coli et de Campylobacter.
Salmonella et Escherichia coli
Les concentrations minimales inhibitrices (CMI) des antimicrobiens ont été déterminées pour Salmonella et E. coli à l'aide d'une méthode automatisée de microdilution en bouillon (Sensititre Complete Automated)Note de bas de page 69,Note de bas de page 70. Ce système d'incubation et de lecture automatisé utilise des microplaques dont les puits contiennent différentes concentrations d'antimicrobiens déshydratés. La plaque CMV4AGNFNote de bas de page 71 a été conçue par le NARMS et contient 14 antimicrobiens (voir le tableau 7, dans la section Valeurs seuils de la sensibilité aux antimicrobiens).
Les isolats ont été étendus par stries sur une gélose Mueller Hinton, qui ont été incubée pendant 18 à 20 heures à 35 ± 1 °C en vue d'obtenir des colonies isolées. Une colonie a été prélevée et étendue par stries sur des géloses de croissance Mueller Hinton (LNM@Guelph utilise des géloses MacConkey pour E. coli). Les géloses ont été incubées pendant 18 à 20 heures à 36 ± 1 °C, après quoi la croissance bactérienne obtenue a servi à préparer des suspensions à une densité de 0,5 McFarland dans 5,0 mL d'eau déminéralisée stérile. Dix microlitres de cette suspension ont été transférés dans des tubes contenant 10 mL de bouillon Mueller Hinton. Cinquante microlitres de cette suspension ont été distribués dans les puits des plaques CMV4AGNF à raison de 50 µL par puits, et les plaques, ont été scellées avec une pellicule de plastique adhésive. Après 18 heures à 35 ± 1 °C, les plaques ont été lues par un système de fluorométrie automatiséNote de bas de page 72. Suivant les normes établies par le CLSINote de bas de page 73, les souches de contrôle Staphylococcus aureus ATCC 29213, Escherichia coli ATCC 25922, Pseudomonas aeruginosa ATCC 27853 et Enterococcus faecalis ATCC 29212 ont été utilisées aux fins de l'assurance de la qualité pour assurer la validité des valeurs des CMI.
Campylobacter
Les valeurs de CMI pour Campylobacter ont été déterminées par la méthode de microdilution en bouillonNote de bas de page 74. Les plaques CAMPY conçues par le NARMS contenant 9 antimicrobiens déshydratés ont été utilisées (voir le tableau 8, dans la section Valeurs seuils de la sensibilité aux antimicrobiens). Des colonies ont été étendues en stries sur des géloses Mueller Hinton contenant 5 % de sang de mouton. Les géloses ont été incubées en microaérophilie, à 42 ± 1 °C pendant 24 heures. Des colonies isolées ont été prélevées et mises en suspension dans des tubes contenant 5 mL de bouillon Mueller Hinton à une densité de 0,5 McFarland. Ensuite, 100 µL de cette suspension ont été transférés dans 11 mL de bouillon Mueller Hinton contenant du sang de cheval hémolysé. Ce mélange a été distribué dans des plaques CAMPY, à raison de 100 µL par puits. Les plaques ont été scellées avec une pellicule de plastique adhésive perforée. Après 24 heures d'incubation en microaérophilie à 42 ± 1 °C, les plaques ont été lues par le système Sensititre VizionNote de bas de page 75. Campylobacter jejuni ATCC 33560 a été utilisé comme témoin de contrôle de la qualité. Les CMI obtenues ont été interprétées en fonction des standards établies par le CLSINote de bas de page 76.
Valeurs seuils de la sensibilité aux antimicrobiens
| aucune donnée disponible | Antimicrobien | Intervalle testé (μg/mL) | Valeurs seuils (μg/mL)Note de bas de page tableau 7 - a (μg/mL) | ||
|---|---|---|---|---|---|
| S | I | R | |||
| I | Amoxicilline-acide clavulanique | 1,0/0,5-32/16 | ≤ 8/4 | 16/8 | ≥ 32/16 |
| Ceftriaxone | 0,25-64 | ≤ 1 | 2 | ≥ 4 | |
| Ciprofloxacine | 0,015-4 | ≤ 0,06 | 0,12-0,5 | ≥ 1 | |
| Méropénème | 0,06-4 | ≤ 1 | 2 | ≥ 4 | |
| II | Ampicilline | 1-32 | ≤ 8 | 16 | ≥ 32 |
| AzithromycineNote de bas de page tableau 7 - b | 0,25-32 | ≤ 16 | S.O. | ≥ 32 | |
| Céfoxitine | 0,5-32 | ≤ 8 | 16 | ≥ 32 | |
| Gentamicine | 0,25-16 | ≤ 4 | 8 | ≥ 16 | |
| Acide nalidixique | 0,5-32 | ≤ 16 | S.O. | ≥ 32 | |
| StreptomycineNote de bas de page tableau 7 - b | 2-64 | ≤ 16 | S.O. | ≥ 32 | |
| Triméthoprime-sulfaméthoxazole | 0,12/2,38-4/76 | ≤ 2/38 | S.O. | ≥ 4/76 | |
| III | Chloramphénicol | 2-32 | ≤ 8 | 16 | ≥ 32 |
| Sulfisoxazole | 16-256 | ≤ 256 | S.O. | ≥ 512 | |
| Tétracycline | 4-32 | ≤ 4 | 8 | ≥ 16 | |
| IV | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible |
Les chiffres romains de I à IV correspondent aux catégories d'antimicrobiens selon leur importance en médecine humaine, telles que définies par la Direction des médicaments vétérinaires. S = Sensible. I = Sensibilité intermédiaire. R = Résistant. S.O. = sans objet. |
|||||
| aucune donnée disponible | Antimicrobien | Intervalle testé (μg/mL) | Valeurs seuilsNote de bas de tableau 8 - a (μg/mL) | ||
|---|---|---|---|---|---|
| S | I | R | |||
| I | Ciprofloxacin | 0,015-64 | ≤ 1 | 2 | ≥ 4 |
| TélithromycineNote de bas de tableau 8 - b | 0,015-8 | ≤ 4 | 8 | ≥ 16 | |
| II | Azithromycinetableau 8 - b | 0,015-64 | ≤ 2 | 4 | ≥ 8 |
| Clindamycinetableau 8 - b | 0,03-16 | ≤ 2 | 4 | ≥ 8 | |
| Érythromycine | 0,03-64 | ≤ 8 | 16 | ≥ 32 | |
| GentamicineNote de bas de tableau 8 - b | 0,12-32 | ≤ 2 | 4 | ≥ 8 | |
| Acide nalidixiqueNote de bas de tableau 8 - b | 4-64 | ≤ 16 | 32 | ≥ 64 | |
| III | FlorfénicolNote de bas de tableau 8 - b,Note de bas de tableau 8 - c | 0,03-64 | ≤ 4 | S.O. | S.O. |
| Tétracycline | 0,06-64 | ≤ 4 | 8 | ≥ 16 | |
| IV | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible | aucune donnée disponible |
Les chiffres romains de I à IV correspondent aux catégories d'antimicrobiens selon leur importance en médecine humaine, telles que définies par la Direction des médicaments vétérinaires. S = Sensible. I = Sensibilité intermédiaire. R = Résistant. S.O. = sans objet. |
|||||
Analyse des données
Surveillance des isolats provenant des humains et du secteur agroalimentaire
Gestion des données
Les données de la Surveillance des isolats humains et de la Surveillance des isolats du secteur agroalimentaire provenant d'un programme informatique propre à chacun des 2 laboratoires (LabWare du LNM@Winnipeg, et Labware de LNM@Guelph et LNM@Saint-Hyacinthe) ont été téléversées dans un référentiel de données à l'aide d'un logiciel intermédiaireNote de bas de page 77. Les données ont ensuite été transférées dans une base de données SAS®Note de bas de page 78 harmonisée au moyen de l'application Data Extraction and Analysis (DEXA). Les variables supplémentaires de la résistance aux antimicrobiens utilisées aux fins d'analyse provenaient de l'application DEXA, laquelle a également servi de point central d'accès aux données.
Taux de détection
Dans le cas de la Surveillance de la viande vendue au détail, de la Surveillance à l'abattoir et de la Surveillance à la ferme, le taux de détection correspondait au nombre de cultures positives divisé par le nombre total d'échantillons mis en culture.
Isolats résistants
Le pourcentage d'isolats résistants à au moins 1 antimicrobien correspondait au nombre d'isolats résistants à au moins 1 antimicrobien divisé par le nombre total d'isolats testés pour chacun des antimicrobiens, multiplié par 100.
Les valeurs seuils utilisées pour l'interprétation des résultats des tests de sensibilité aux antimicrobiens sont présentées au tableau 7 et au tableau 8 (voir la section précédente). Lorsque les valeurs de CMI étaient intermédiaires, les organismes ont été considérés comme sensibles aux antimicrobiens pour toutes les analyses. Une nouvelle valeur seuil pour la ceftriaxone a officiellement été adoptée par le CLSI en janvier 2010. Cette nouvelle valeur a été utilisée pour toutes les données du PICRA, y compris les données historiques. Une nouvelle plaque pour les Enterobacteriaceae, CMV4AGNF, a été utilisée à partir de janvier 2016. Les modifications notables de cette nouvelle plaque comprennent :
- Le retrait du ceftiofur (catégorie I)
- L'ajout de la méropénème (Catégorie I)
- L'ajustement de l'intervalle de sensibilité testé (0,25 à 32 μg/mL) pour déterminer la CMI de l'azithromycine
- Le changement du seuil de sensibilité de la streptomycine à plus grand ou égal à 32 μg/mL.
Profils de résistance
Pour chaque profil de résistance, le nombre total d'antimicrobiens correspondait au nombre d'antimicrobiens auxquels chacun des isolats était résistant. Le profil de résistance le plus fréquent peut comprendre 1 seul antimicrobien. Dans ce cas, comme pour les profils de résistance les plus fréquents comprenant 2 antimicrobiens ou plus, le nombre d'isolats indiqué ne comprend que ceux qui sont résistants au profil en question (c.-à-d. sans résistance à d'autres antimicrobiens).
Analyse statistique
Les données ont été analysées avec différents logiciels statistiquesNote de bas de page 79, et les résultats ont été exportés dans un tableur électroniqueNote de bas de page 80. Tous les tableaux et figures ont été créés avec le tableur.
En ce qui concerne la Surveillance à la ferme, les analyses statistiques ont été faites avec des équations d'estimation généraliséesNote de bas de page 81 pour tenir compte du regroupement des résistances aux antimicrobiens dans les troupeaux de bovins en parc d'engraissement, de porcs en croissance-finition, de poulets de chair et de dindons. Tous les modèles statistiques utilisés comprenaient des résultats binaires, une fonction de lien logit et une structure de corrélation échangeable. Des modèles de réponse binomiale par zéro ont été utilisés pour estimer la fréquence de la résistance à chacun des antimicrobiens. Pour chaque modèle, on a utilisé le point d'intersection (β0) et un intervalle de confiance à 95 % pour estimer la fréquence moyenne dans la population (équations d'estimation généralisées) avec l'équation [1 + exp(‐β0)]‐1. Lorsque la prévalence était de 0 %, un modèle a été exécuté avec un seul isolat positif afin de déterminer l'intervalle de confiance supérieur uniquement.
Analyse de la variation temporelle
La variation temporelle de la résistance à certains antimicrobiens a été analysée. Dans la mesure du possible, un seul antimicrobien par classe a été choisi parmi ceux qui sont couramment utilisés dans le secteur agroalimentaire ou chez les humains. Certains antimicrobiens étaient exclus des analyses de variations temporelles pour les raisons suivantes :
- La fréquence des isolats bactériens résistants à l'antimicrobien en question était faible ou nulle, ou encore la valeur seuil était discutable et d'autres antimicrobiens pouvaient être utilisés comme mesure indirecte de la résistance ou de la sensibilité intermédiaire (p. ex. l'acide nalidixique pour la ciprofloxacine).
- L'isolat présentait de la résistance croisée à un autre antimicrobien sélectionné (p. ex. l'amoxicilline-acide clavulanique et le ceftiofur).
- L'utilisation de l'antimicrobien en question a été interdite pour le secteur agroalimentaire et la résistance à ce dernier persiste en raison de l'utilisation d'un autre antimicrobien (p. ex. le chloramphénicol).
Dans les modèles de régression logistique (asymptotique ou exact selon la prévalence de la variable dépendante) développés, l'année a servi de variable nominale indépendante. Les données ont été analysées à l'aide d'un logiciel commercialNote de bas de page 82. Les données de la composante Surveillance à la ferme ont été pondérées pour tenir compte de l'effet de regroupement au niveau du troupeau chez les porcs en croissance‐finition et les poulets de chair. En ce qui concerne les composantes comportant une analyse de la variation temporelle à l'échelle régionale ou provinciale, la proportion d'isolats résistants à un antimicrobien spécifique de l'année en cours a été comparée aux proportions observées au cours de l'année de surveillance précédente et des 5 années précédentes. Dans le cas des poulets de chair, on a comparé les données de 2017 à celles de 2016 et 2013. En ce qui concerne les composantes comportant une analyse de la variation temporelle à l'échelle nationale, la proportion d'isolats résistants à un antimicrobien spécifique de l'année en cours a été comparée aux proportions observées au cours de l'année de surveillance précédente, des 5 années précédentes (aux fins de comparaison entre composantes) et des 10 années précédentes (ou de la première année de surveillance). Dans quelques cas bien précis, la première année de la surveillance peut être différente, compte tenu de la mise en œuvre de nouvelles composantes du PICRA (p. ex., 2006 pour la Surveillance à la ferme des porcs en croissance-finition, et l'ajout, en 2013, des poulets de chair à la composante Surveillance à la ferme). En ce qui concerne l'ampicilline et la ceftriaxone (antérieurement le ceftiofur), des analyses particulières de la variation temporelle de la résistance à ces antibiotiques ont été faites chez les isolats d'E. coli et de Salmonella de la viande de poulet vendue au détail ou de poulets à l'abattoir, afin de comparer les données de l'année en cours à celles de 2004 et de 2006, en raison du changement des pratiques d'utilisation du ceftiofur dans les couvoirs du Québec au début de 2005 et en 2007 (soit au début et à la fin, respectivement, de la période de retrait de cet antimicrobien). Ces analyses particulières ont aussi été effectuées pour des souches de Salmonella Heidelberg isolées d'humains, car on les soupçonnait de provenir de viande de poulet. Une valeur prédictive P inférieure ou égale à 0,05 a été considérée comme significative pour toutes ces analyses de la variation temporelle.
Classification des antimicrobiens
Classification des antimicrobiens selon leur importance en médecine humaine
La classification des antimicrobiens utilisée dans le présent rapport provient du document de classification des antimicrobiens selon leur importance en médecine humaineNote de bas de page 83 par la Direction des médicaments vétérinaires de Santé Canada (tableau 9). Les antimicrobiens sont considérés de « Très haute importance » en médecine humaine (Catégorie I) lorsqu'ils sont essentiels au traitement des infections bactériennes graves et qu'il y a rareté ou absence de médicaments efficaces. Ces antimicrobiens comprennent l'amoxicilline-acide clavulanique, la ceftriaxone (ceftiofurNote de bas de page 84), la ciprofloxacine et la télithromycine. Les antimicrobiens de « Haute importance » en médecine humaine (Catégorie II) sont ceux qui peuvent être utilisés dans le traitement de diverses infections, comme les infections graves pour lesquelles des antimicrobiens de remplacement sont généralement disponibles. Les bactéries résistantes aux antimicrobiens de cette catégorie sont généralement sensibles à ceux de la Catégorie I, lesquels peuvent servir d'antimicrobiens de remplacement. Les antimicrobiens d'« Importance moyenne » (Catégorie III) sont utilisés dans le traitement des infections bactériennes pour lesquelles des médicaments de remplacement sont généralement disponibles. Les infections causées par des bactéries résistantes à ces médicaments peuvent, en général, être traitées par des antimicrobiens de la Catégorie II ou de la Catégorie I. Les antimicrobiens de « Faible importance » en médecine humaine (Catégorie IV) ne sont pas utilisés actuellement en médecine humaine.
| aucune donnée disponible | Catégorie d'importance en médecine humaine | Classe d'antimicrobiens |
|---|---|---|
| I | Très haute importance | Carbapénèmes |
| Céphalosporines de troisième et de quatrième génération | ||
| Fluoroquinolones | ||
| Glycopeptides | ||
| Glycylcyclines | ||
| Kétolides | ||
| Lipopeptides | ||
| Monobactames | ||
| Nitroimidazoles (métronidazole) | ||
| Oxazolidinones | ||
| Associations de pénicilline-inhibiteur de β-lactamase | ||
| Polymyxines (colistine) | ||
| Agents thérapeutiques contre la tuberculose (ex. éthambutol, isoniazide, pyrazinamide et rifampine) | ||
| II | Haute importance | Aminoglycosides (sauf les agents topiques) |
| Céphalosporines - Première et deuxième générations (y compris les céphamycines) | ||
| Acide fusidique | ||
| Lincosamides | ||
| Macrolides | ||
| Pénicillines | ||
| Quinolones (sauf les fluoroquinolones) | ||
| Streptogramines | ||
| Triméthoprime-sulfaméthoxazole | ||
| III | Moyenne importance | Aminocyclitols |
| Aminoglycosides (agents topiques) | ||
| Bacitracines | ||
| Fosfomycine | ||
| Nitrofuranes | ||
| Phénicoles | ||
| Sulfamides | ||
| Tétracyclines | ||
| Triméthoprime | ||
| IV | Faible importance | Flavophospholipides |
| Ionophores | ||
Les chiffres romains de I à IV correspondent aux catégories d'antimicrobiens selon leur importance en médecine humaine, telles que définies par la Direction des médicaments vétérinaires. |
||
Liste des antimicrobiens tirée du questionnaire visant les fermes d'élevages de poulets de chair et de dindons
| aucune donnée disponible | Classe ATCvet | Antimicrobien |
|---|---|---|
| Antimicrobiens administrés par l'alimentation | ||
| II | Aminoglycosides, autre (QJ01GB) | Néomycine (QJ01GB05) |
| Apramycine (QJ01GB90) | ||
| Lincosamides (QJ01FF) | Lincomycine (AJ01FF02) | |
| Association de lincosamides-aminocyclitol (QJ01RA94) | Lincomycine-spectinomycine (pas de code ATCvet) | |
| Macrolides (QJ01FA) | Érythromycine (QJ01FA01) | |
| Tylosine (QJ01FA90) | ||
| Pénicillines (QJ01RA) | Pénicilline (QJ01RA01) | |
| Benzylpénicilline-procaïne (QJ01CE09) | ||
| Streptogramines (QJ01FG) | Virginiamycine (QJ01FG90) | |
| III | Bacitracines (QA07AA) | Bacitracine (QA07AA93) |
| Sulfamides, seul ou en association, voie intestinale (QP51AG) | Sulfaméthazine (pas de code ATCvet) | |
| Triméthoprime-sulfadiazine (pas de code ATCvet) | ||
| Tétracyclines (QJ01AA) | Chlortétracycline (QJ01AA03) | |
| Oxytétracycline (QJ01AA06) | ||
| Tétracycline (QJ01AA07) | ||
| IV | Flavophospholipides | Bambermycine (pas de code ATCvet) |
| Ionophores, agents contre les maladies à protozoaires (QP51A) | Lasalocide (QP51AH02) | |
| Maduramicine (QP51AX10) | ||
| Monensin (QP51AH03) | ||
| Narasin (QP51AH04) | ||
| Association de narasin-nicarbazine (QP51AH54) | ||
| Salinomycine (QP51AH01) | ||
| S.O. | Arsenicaux, agents contre les maladies à protozoaires (QP51AD) | Acide 4-nitrophenylarsonic (pas de code ATCvet) |
| Anticoccidiens de sythèse, autres protozoaires (QP51AX) | Amprolium (QP51AX09) | |
| Clopidol (pas de code ATCvet) | ||
| Décoquinate (QP51AX14) | ||
| Diclazuril (QP51AJ03) | ||
| Nicarbazine (QP51AE03) | ||
| Robénidine (QP51AX13) | ||
| Zoalène/Dinitolmide (QP51AX12) | ||
| Orthosomycine | Avilamycine (pas de code ATCvet) | |
| Antimicrobiens administrés par l'eau | ||
| I | Fluoroquinolones | Enrofloxacine (QJ01MA90) |
| II | Aminoglycosides, autres (QJ01GB) | Néomycine (QJ01GB05) |
| Apramycine (QJ01GB90) | ||
| Lincosamides, associations avec d'autres antimicrobiens | Lincomycine-spectinomycine (QJ01RA94) | |
| Macrolides (QJ01FA) | Érythromycine (QJ01FA01) | |
| Tylosine (QJ01FA90) | ||
| Pénicillines, à large spectre (QJ01CA) | Amoxicilline (QJ01CA04) | |
| Pénicillines (QJ01RA) | Pénicilline (QJ01RA90) | |
| Pénicillines, association avec d'autres antibactériens (QJ01RA) | Pénicilline-streptomycine (QJ01RA01) | |
| III | Amphénicols (QJ01BA) | Florfénicol (QJ01BA90) |
| Sulfamides, seul ou en association, intestinal (QP51AG) | Sulfaméthazine (pas de code ATCvet) | |
| Sulfaquinoxaline (QP51AG03) | ||
| Sulfaquinoxaline-pyriméthamine (pas de code ATCvet) | ||
| Tétracyclines (QJ01AA) | Chlortétracycline (QJ01AA03) | |
| Oxytétracycline (QJ01AA06) | ||
| Tétracycline (QJ01AA07) | ||
| Tétracyclines et associations (QJ01RA90) | Oxytétracycline-néomycine (pas de code ATCvet) | |
| Tétracycline-néomycine (pas de code ATCvet) | ||
| Antimicrobiens administrés par injection sous-cutanée ou in ovo | ||
| I | Céphalosporines de troisième-génération (QJ01DD) | Ceftiofur (QJ01DD90) |
| II | Aminoglycosides, autres (QJ01GB) | Gentamicine (QJ01GB03) |
| Associations de lincosamides-aminocyclitol (QJ01RA94) | Lincomycine-spectinomycine (pas de code ATCvet) | |
ATC = anatomique, thérapeutique et chimique. Les chiffres romains de I à IV correspondent aux catégories d'antimicrobiens selon leur importance en médecine humaine, telles que définies par la Direction des médicaments vétérinaires. S.O. = sans objet (aucune classification disponible au moment de la rédaction du présent rapport). Le système ATC pour la classification des médicaments vétérinaires est fondé sur les mêmes principes de base que le système ATC pour les substances utilisées en médecine humaine. Ce système est un outil permettant l'échange et la comparaison de données sur l'utilisation de médicaments en médecine vétérinaire à l'échelle internationale, nationale ou localeNote de bas de page 85. |
||
Liste des antimicrobiens tirée du questionnaire visant les fermes d'élevages de porcs en croissance-finition
| aucune donnée disponible | Classe ATCvet | Antimicrobien |
|---|---|---|
| I | Céphalosporines de troisième génération (QJ01DD) | Ceftiofur (QJ01DD90) |
| Fluoroquinolones (QJ01MA) | Enrofloxacine (QJ01MA90) | |
| II | Amphénicols (QJ01BA) | Florfénicol (QJ01BA90) |
| Pénicillines à large spectre (QJ01CA) | Ampicilline (QJ01CA01) | |
| Amoxicilline (QJ01CA04) | ||
| Pénicillines sensibles aux β-lactamases (QJ01CE) | Pénicilline (QJ01CE01) | |
| Association de sulfadoxine et de triméthoprime (QJ01EW) | Triméthoprime-sulfadoxine (QJ01EW13) | |
| Macrolides (QJ01FA) | Érythromycine (QJ01FA01) | |
| Tylosine (QJ01FA90) | ||
| Tilmicosine (QJ01FA91) | ||
| Tulathromycine (QJ01FA94) | ||
| Lincosamides (QJ01FF) | Lincomycine (QJ01FF02) | |
| Streptogramines (QJ01FG) | Virginiamycine (QJ01FG90) | |
| Autres aminoglycosides (QJ01GB) | Néomycine (QJ01GB05) | |
| Association d'antibactériens (QJ01RA) | Pénicilline-streptomycine (QJ01RA01) | |
| Chlortétracycline-sulfaméthazine-pénicilline (QJ01RA90) | ||
| Oxytétracycline-néomycine (QJ01RA90) | ||
| Tétracycline-néomycine (QJ01RA90) | ||
| Lincomycine-spectinomycine (QJ01RA94) | ||
| Autres antibactériens (QJ01XX) | Spectinomycine (QJ01XX04) | |
| III | Tétracyclines (QJ01AA) | Chlortétracycline (QJ01AA03) |
| Oxytétracycline (QJ01AA06) | ||
| Tétracycline (QJ01AA07) | ||
| Chlortétracycline, combinations (QJ01AA53) | ||
| Sulfonamides (QJ01EQ) | Association de sulfonamides (QJ01EQ30) | |
| Pleuromutilins (QJ01XQ) | Tiamuline (QJ01XQ01) | |
| Autres antibactériens (QJ01XX) | Bacitracine (QJO1XX10) | |
| IV | Pas de code ATCvet | Bambermycine (pas de code ATCvet) |
| Pyranes et hydropyranes (QP51AH) | Salinomycine (QP51AH01) | |
ATC = Système de classification anatomique, thérapeutique et chimique. Les chiffres romains de I à IV correspondent aux catégories d'antimicrobiens selon leur importance en médecine humaine, telles que définies par la Direction des médicaments vétérinaires. Le système ATC pour la classification des médicaments vétérinaires est fondé sur les mêmes principes de base que le système ATC pour les substances utilisées en médecine humaine. Ce système est un outil permettant l'échange et la comparaison de données sur l'utilisation de médicaments en médecine vétérinaire à l'échelle internationale, nationale ou localeNote de bas de page 87. |
||
Annexe
Abréviations
Provinces canadiennes, territoires et régions
Provinces
- BC
- Colombie-Britannique
- AB
- Alberta
- SK
- Saskatchewan
- MB
- Manitoba
- ON
- Ontario
- QC
- Québec
- NB
- New Brunswick
- NS
- Nouvelle-Écosse
- PE
- Île-du-Prince-Édouard
- NL
- Terre-Neuve-et-Labrador
Territoires
- YT
- Yukon
- NT
- Territoires du Nord-Ouest
- NU
- Nunavut
RégionsNote de bas de page 88
Prairies : AB, SK, MB
Maritimes : NB, NS, PE
Atlantique : NB, NS, PE, NL
Antimicrobiens
- AMC
- amoxicilline-acide clavulanique
- AMP
- ampicilline
- AZM
- azithromycine
- CHL
- chloramphénicol
- CIP
- ciprofloxacine
- CLI
- clindamycine
- CRO
- ceftriaxone
- ERY
- érythromycine
- FLR
- florfénicol
- FOX
- céfoxitine
- GEN
- gentamicine
- MEM
- méropénème
- NAL
- acide nalidixique
- SSS
- sulfisoxazole
- STR
- streptomycine
- SXT
- triméthoprime-sulfaméthoxazole
- TEL
- télithromycine
- TET
- tétracycline
- TIO
- ceftiofur
Profils de résistance importants
- A2C-AMP
- amoxicilline-acide clavulanique, céfoxitine, ceftiofur et ampicilline
- ACSSuT
- ampicilline, chloramphénicol, streptomycine, sulfisoxazole et tétracycline
Autres abréviations
- APP
- Actinobacillus pleuropneumoniae
- DEP
- Diarrhée épidémique porcine
- DMV
- Direction des médicaments vétérinaires, Santé Canada
- ECAP
- Escherichia coli aviaire pathogène
- FNC
- FoodNet Canada
- GET
- Gastroentérite transmissible
- MACVP
- Maladie associée au circovirus porcin
- PJAR
- Porcs-jours à risque
- SRRP
- Syndrome reproducteur et respiratoire porcin
- VBI
- Virus de la bronchite infectieuse
Données additionnelles
| Voie d'administration | Voie d'administration européenne | Antimicrobiens | Utilisation de base du dosage moyen | Dosage moyen | DDDvetCA (mgmédicament/kganimal/jour) |
|---|---|---|---|---|---|
| Aliments | Orale | Avilamycine | TP | 22,5 | 2,9 |
| Bacitracine | TP | 77,9 | 10,1 | ||
| Chlortétracycline | TP | 128,3 | 16,7 | ||
| Érythromycine | TP | 220,0 | 28,6 | ||
| Oxytétracycline | TP | 128,3 | 16,7 | ||
| Pénicilline G procaïne | TP | 41,3 | 5,4 | ||
| Sulfadiazine-triméthoprime (UMDDE)Note de bas de tableau A-1 - a (ELDU) | TP | 83,3 | 10,8 | ||
| Triméthoprime-sulfadiazine (UMDDE)Note de bas de tableau A-1 - a (ELDU) | TP | 16,8 | 2,2 | ||
| Tylosine | TP | 200,0 | 26,0 | ||
| Virginiamycine | TP | 22,0 | 2,9 | ||
| Injection | Parentérale | Ceftiofur (UMDDE) | TP | 2,6 | 2,6 |
| Gentamicine | TP | 10,8 | 10,8 | ||
| Lincomycine-spectinomycine (UMDDE)Note de bas de tableau A-1 - a (ELDU) | TP | 6,0 | 6,0 | ||
| Spectinomycine-lincomycinea (UMDDE)Note de bas de tableau A-1 - a (ELDU) | TP | 12,0 | 12,0 | ||
| Eau | Orale | Amoxicilline | TP | 52,0 | 12,0 |
| Apramycine (UMDDE) | TP | 100,0 | 23,0 | ||
| Érythromycine | TP | 86,7 | 19,9 | ||
| Lincomycine | TP | 16,0 | 3,7 | ||
| Lincomycine-spectinomycineNote de bas de tableau A-1 - a | TP | 277,5 | 63,8 | ||
| Néomycine | TP | 94,8 | 21,8 | ||
| Oxytétracycline | TP | 81,9 | 18,8 | ||
| Pénicilline G | TP | 178,3 | 41,0 | ||
| Pénicilline G (supp) | TP | 16,5 | 3,8 | ||
| Spectinomycine-lincomycineNote de bas de tableau A-1 - a | TP | 555,0 | 127,7 | ||
| Streptomycine (supp) | TP | 85,2 | 19,6 | ||
| Sulfaméthazine | TP | 1027,8 | 236,4 | ||
| Sulfaquinoxaline | TP | 317,2 | 72,9 | ||
| Tétracycline | TP | 93,1 | 21,4 | ||
| Tylosine | TP | 312,5 | 71,9 | ||
| Sulfaquinoxaline-pyriméthamineNote de bas de tableau A-1 - a | TP | 48,8 | 11,2 | ||
L'utilisation de médicaments en dérogation des directives de l'étiquette (UMDDE) chez le poulet de chair, la dose ou les dose(s) proviennent de l'opinion d'experts ou de consultations auprès de vétérinairesNote de bas de page 89. TP = traitement et prévention. SC = stimulation de la croissance. Supp = supplément ou produit ayant une faible teneur d'antimicrobien. Dose moyenne = moyenne de toutes les doses indiquées dans le Recueil des notices sur les substances médicatricesNote de bas de page 90 et le Compendium des produits vétérinairesNote de bas de page 91; les valeurs ont été multipliées aux valeurs standards de consommation d'aliments ou d'eau (voir le tableau A. 3) pour obtenir les valeurs standards de DDDvetCA pour les volailles. DDDvetCA = doses définies journalières (Defined Daily Doses) canadiennes pour les animaux (dose moyenne selon les directives de l'étiquette) en milligrammes par kilogramme de poulet de chair ou de dindon par jour (mgmédicament/kganimal/Jour). Les standards des DDDvetCA pour les produits dont le dosage est beaucoup plus faible que les produits utilisés à des fins préventives et thérapeutiques, tels que les ionophores, les anticoccidiens de synthèse et les produits destinés principalement à la stimulation de la croissance (les flavophospolipides et la pénicilline G par la voie alimentaire), ont été développés et sont disponibles dans des rapports antérieurs ou sur demande. Le nombre total de DDDvetCA pour ces produits n'est pas inclus dans ce rapport. |
|||||
| Voie d'administration | Antimicrobiens | Utilisation de base du dosage moyen | Dosage moyen | DDDvetCA (mgmédicament/kganimal/jour) |
|---|---|---|---|---|
| Aliments | Avilamycine | TP | 80,0 | 3,2 |
| Bacitracine | TP | 113,4 | 4,5 | |
| Bambermycine | SC | 3,0 | 0,1 | |
| Chlortétracycline | TP | 260,3 | 10,4 | |
| Lincomycine | TP | 124,7 | 5,0 | |
| Lincomycine-spectinomycineNote de bas de tableau A-2 - a | TP | 22,0 | 0,9 | |
| Narasin | SC | 15,0 | 0,6 | |
| Oxytétracycline | TP | 189,4 | 7,6 | |
| TP | 32,1 | 1,3 | ||
| Salinomycine | SC | 25,0 | 1,0 | |
| Spectinomycine-lincomycineNote de bas de tableau A-2 - a | TP | 22,0 | 0,9 | |
| Sulfaméthazine | TP | 110,0 | 4,4 | |
| Tiamuline | TP | 116,0 | 4,6 | |
| Tilmicosine | TP | 300,0 | 12,0 | |
| Tylosine | TP | 77,0 | 3,1 | |
| Tylvalosine | TP | 42,5 | 1,7 | |
| Virginiamycine | TP | 82,5 | 3,3 | |
| Injection | Ampicilline | TP | 6,0 | 6,0 |
| Benzathine pénicilline G (association)Note de bas de tableau A-2 - a | TP | 1,2 | 1,2 | |
| Ceftiofur | TP | 3,0 | 3,0 | |
| Ceftiofur-longue action | TP | 1,0 | 1,0 | |
| Enrofloxacin | TP | 7,5 | 7,5 | |
| Florfénicol | TP | 7,5 | 7,5 | |
| Gentamicine | TP | 1,3 | 1,3 | |
| Lincomycine | TP | 10,0 | 10,0 | |
| Oxytétracycline | TP | 5,9 | 5,9 | |
| Pénicilline G procaïne | TP | 13,5 | 13,5 | |
| Pénicilline G procaïne (longue-action) | TP | 6,7 | 6,7 | |
| Pénicilline G procaïne (association)Note de bas de tableau A-2 - a | TP | 1,5 | 1,5 | |
| Sulfadoxine-triméthoprimeNote de bas de tableau A-2 - a | TP | 13,3 | 13,3 | |
| Tiamuline | TP | 11,0 | 11,0 | |
| Triméthoprime-sulfadoxineNote de bas de tableau A-2 - a | TP | 2,4 | 2,4 | |
| Tulathromycine | TP | 0,3 | 0,3 | |
| Tylosine | TP | 5,5 | 5,5 | |
| Eau | Amoxicilline | TP | 200,0 | 20,0 |
| Apramycine | TP | 100,0 | 10,0 | |
| Lincomycine | TP | 33,3 | 3,3 | |
| Lincomycine-spectinomycineNote de bas de tableau A-2 - a | TP | 22,2 | 2,2 | |
| Néomycine | TP | 115,9 | 11,6 | |
| Oxytétracycline | TP | 146,4 | 14,6 | |
| Pénicilline G | TP | 178,0 | 17,8 | |
| Spectinomycine-lincomycineNote de bas de tableau A-2 - a | TP | 44,4 | 4,4 | |
| Sulfamérazine (supp) | TP | 32,9 | 3,3 | |
| Sulfaméthazine | TP | 789,7 | 79,0 | |
| Sulfaméthazine (supp) | TP | 62,8 | 6,3 | |
| Sulfapyridine | TP | 333,3 | 33,3 | |
| Sulfathiazole | TP | 462,1 | 46,2 | |
| Sulfathiazole (supp) | TP | 103,0 | 10,3 | |
| Tétracycline | TP | 85,9 | 8,6 | |
| Tiamuline | TP | 49,0 | 4,9 | |
| Tylosine | TP | 166,5 | 16,7 | |
| Tylvalosine | TP | 50,0 | 5,0 | |
| Bolus | Néomycine (supp) | TP | 7,5 | 7,5 |
| Néomycine | TP | 19,7 | 19,7 | |
| Oxytétracycline | TP | 29,3 | 29,3 | |
| Spectinomycine-lincomycineNote de bas de tableau A-2 - a | TP | 18,8 | 18,8 | |
| Succinylsulfathiazole (supp) | TP | 36,0 | 36,0 | |
| Sulfaguanidine | TP | 83,8 | 83,8 | |
| Sulfaméthazine | TP | 118,1 | 118,1 | |
| Sulfanilamide | TP | 73,1 | 73,1 | |
| Sulfathiazole | TP | 57,4 | 57,4 | |
| Tétracycline | TP | 15,3 | 15,3 | |
| Toltrazuril | TP | 20,0 | 20,0 | |
TP = traitement et prévention. SC = stimulation de la croissance. Supp = supplément ou produit ayant une faible teneur d'antimicrobien. Dose moyenne = moyenne de toutes les doses indiquées dans le Recueil des notices sur les substances médicatricesNote de bas de page 92 et le Compendium des produits vétérinairesNote de bas de page 93; les valeurs ont été multipliées aux valeurs standards de consommation d'aliments ou d'eau (voir le tableau A-4) pour obtenir les valeurs standards de DDDvetCA pour les porcs en croissance-finition. DDDvetCA = doses définies journalières (Defined Daily Doses) canadiennes pour les animaux (dose moyenne selon les directives de l'étiquette) en milligrammes par kilogramme de porcs en croissance-finition par jour (mgmédicament/kganimal/Jour). |
||||
| Valeurs standards relatives à la consommation d'aliments et d'eau | Volaille |
|---|---|
| Poids standard canadien du dindonneau (kg au temps de l'éclosion)Note de bas de page tableau A-3 - a | 0,06 |
| Poids standard canadien du poussin (kg au temps de l'éclosion)Note de bas de page tableau A-3 - a | 0,042 |
| Poids standard canadien du poulet de chair (kg)Note de bas de page tableau A-3 - a | 1,0 |
| Standard canadien de l'indice de conversion alimentaire | 0,13 |
| Standard canadien de l'indice de conversion hydrique | 0,23 |
| Indice de conversion alimentaire de l'ESVAC (kg d'aliment/kg d'animal)Note de bas de page tableau A-3 - b | 0,13 |
| Indice de conversion hydrique de l'ESVAC (L d'eau/kg d'animal)Note de bas de page tableau A-3 - b | 0,23 |
ESVAC = European Surveillance of Veterinary Antimicrobial Consumption (Surveillance européenne de la consommation d'antibiotiques à usage vétérinaire). DDDA = Dose définie journalière pour les animaux. |
|
| Valeurs standards relatives à la consommation d'aliments et d'eau | Porcin |
|---|---|
| Poids standard canadien du porcelet (kg) | 4,00 |
| Poids standard canadien de porc en croissance-finition (kg) | 65,00 |
| Standard canadien de la consommation d'eau (pour un porc de 65 kg) (L)Note de bas de tableau A-4 - a | 6,50 |
| Standard canadien de la consommation d'aliments (pour un porc de 65 kg) (kg) | 2,18 |
| Standard canadien de l'indice de conversion alimentaire | 0,04 |
| Standard canadien de l'indice de conversion hydrique | 0,10 |
| Indice de conversion alimentaire de l'ESVAC (kg d'aliment/kg d'animal)Note de bas de tableau A-4 - b | 0,04 |
| Indice de conversion hydrique de l'ESVAC (L d'eau/kg d'animal)Note de bas de tableau A-4 - b | 0,10 |
ESVAC = European Surveillance of Veterinary Antimicrobial Consumption (Surveillance européenne de la consommation d'antibiotiques à usage vétérinaire). |
|
Résumé du flot des données de RAM et d'UAM
Figure A-1 Résumé du flux des échantillons et des données du PICRA, 2017
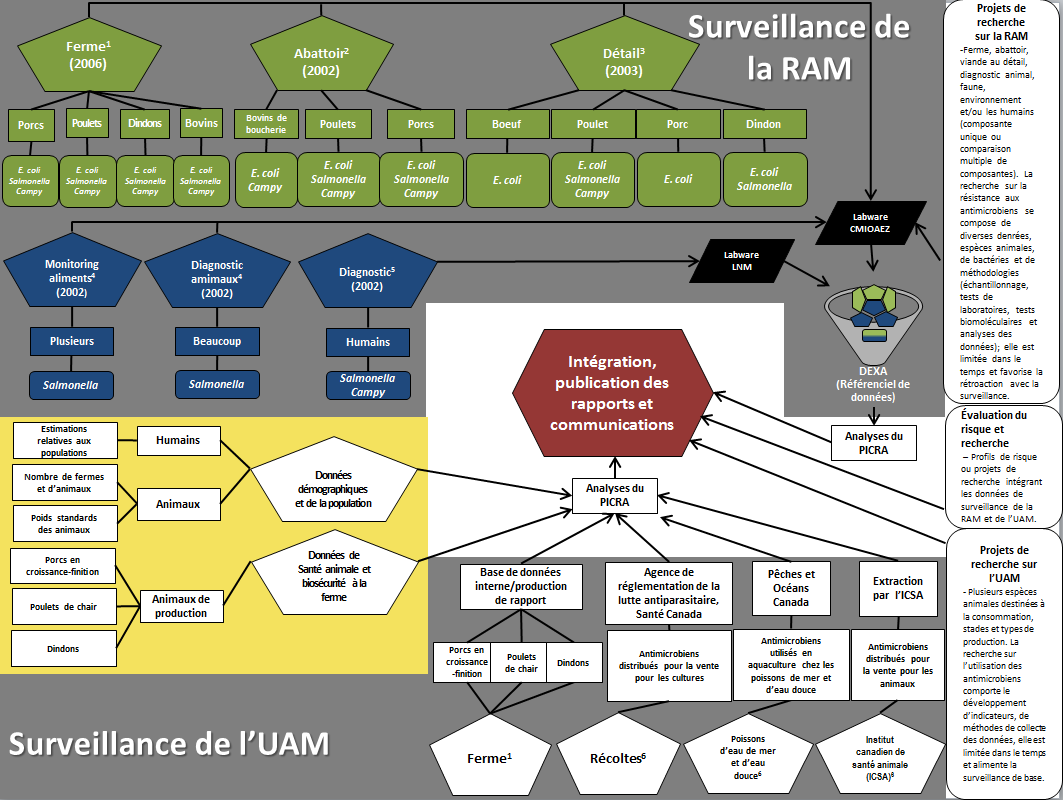
Forme en vert = Surveillance active; données primaires, principalement pour estimer la prévalence. Forme en bleu = Surveillance passive; données secondaires, principalement pour la détection de la RAM.
CMIOAEZ = Centre des maladies infectieuses d'origine alimentaire, environnementale et zoonotique. LNM = Laboratoire national de microbiologie
1-7 Responsables des composantes du PICRA : 1 David Léger (david.leger@canada.ca), Sheryl Gow (sheryl.gow@canada.ca) et Agnes Agunos (agnes.agunos@canada.ca); 2 Anne Deckert (anne.deckert@canada.ca); 3,4,5 Brent Avery (brent.avery@canada.ca); 4,5 Colleen Murphy (colleen.murphy@canada.ca); 5 Amrita Bharat (amrita.bharat@canada.ca) et Michael Mulvey (michael.mulvey@canada.ca); 6 Carolee Carson (carolee.carson@canada.ca).
Coordonnateurs du programme du PICRA : Rebecca Irwin (rebecca.irwin@canada.ca), Richard Reid-Smith (richard.reid-smith@canada.ca), et Michael Mulvey (michael.mulvey@canada.ca).
Figure A-1 - Équivalent textuel
Ce schéma donne une vue d'ensemble de toutes les composantes de surveillance du Programme intégré canadien de surveillance de la résistance aux antimicrobiens (PICRA) ainsi que toutes les sources de données colligées aux fins d'analyse et de publication de rapports.
La moitié supérieure de la figure (zone grise) inclut toutes les composantes de la résistance aux antimicrobiens (RAM) du PICRA. Les composantes de l'utilisation des antimicrobiens (UAM) sont présentées dans le bas de la figure (zone grise). D'autres sources de données sont présentées (zone en jaune et les zones à la droite de la figure): données démographiques, données concernant les populations, données de santé animale et de biosécurité à la ferme. Toutes ces données vont être analysées, intégrées, publiées dans des rapports et être communiquées (zone blanche au centre de la figure).
Section de la RAM (moitié supérieure/zone grise)
Composantes de surveillance active (formes en vert)
Les trois composantes de surveillance active de la RAM sont représentées (à la ferme, à l'abattoir et au détail) :
La composante de surveillance à la ferme a débuté en 2006 et comporte des échantillons provenant de porcs en croissance-finition, de poulets de chair, de dindons et de bovins à la ferme. Les bactéries Escherichia coli, Salmonella et Campylobacter sont isolées à partir des échantillons provenant de porcs, de poulets, de dindons et de bovins.
La composante de surveillance à l'abattoir a débuté en 2002 et repose sur des échantillons provenant de bovins de boucherie, de poulets et de porcs. Les bactéries Escherichia coli, Salmonella et Campylobacter sont isolées à partir d'échantillons provenant de poulets et de porcs alors que les bactéries E. coli et Campylobacter sont isolées à partir d'échantillons de bovins de boucherie.
La composante de surveillance de la viande vendue au détail a débuté en 2003 et repose sur la collecte de viande de bœuf, de poulet, de porc et de dindon. L'isolement d'E. coli est effectué pour tous ces types de viande, l'isolement pour Salmonella est effectué à partir d'échantillons de viande de poulet, de porc et de dindon alors que, l'isolement pour Campylobacter est effectué seulement à partir d'échantillons de viande de poulet.
Composantes de surveillance passive (formes en bleu)
En complément à la surveillance active de la RAM, il y a trois composantes de surveillance passive au PICRA : le monitoring des aliments (pour les animaux), le diagnostic chez les animaux et le diagnostic chez les humains.
La composante de monitoring des aliments comporte plusieurs types d'échantillons alors que la surveillance effectuée à partir d'échantillons pour le diagnostic chez les animaux peut inclure beaucoup d'espèces animales. Toutes les composantes de surveillance passive incluent la recherche d'isolats de Salmonella mais la recherche de Campylobacter est aussi effectuée pour les isolats humains.
Projets de recherche sur la RAM
De plus, les projets de recherche sur la RAM peuvent inclurent plusieurs types de viandes, d'espèces animales, de bactéries, de méthodes (méthode d'échantillonnage, tests de laboratoire, tests biomoléculaires et analyses de données). Ces projets d'une durée limitée apportent un complément aux données obtenues par la surveillance régulière de la RAM.
Analyses des données de RAM
Enfin, toutes les données de RAM d'origine humaines sont compilées dans le système de gestion de données Labware-LNM (forme en noir) du Laboratoire national de microbiologie (LNM). Toutes les données de RAM du secteur agroalimentaire sont compilées dans un second système de gestion de données Labware-CMIOAEZ (forme en noir), cette fois géré par le Centre des maladies infectieuses d'origine alimentaire, environnementale et zoonotique (CMIOAEZ). Les données de RAM provenant des projets de recherche sont aussi entrées dans le Labware du CMIOAEZ. Toutes les données sont alors acheminées dans DEXA, un référenciel centralisé, qui rend les données accessibles par les analystes du PICRA.
Section de l'UAM (moitié inférieure/zone grise)
Les quatre composantes de surveillance de l'UAM (à la ferme, les récoltes et l'Institut canadien de santé animale) sont présentées :
Les données d'UAM à la ferme proviennent de porcs en croissance-finition, de poulets de chair, de dindons et sont compilées dans une base de données interne et un système de production de rapports. Les données sur les cultures et l'aquaculture des poissons marins et d'eau douce résument la quantité d'antimicrobiens distribués pour la vente pour utilisation; ces données sont fournies directement au PICRA par l'Agence de réglementation de la lutte antiparasitaire de Santé Canada et par Pêches et Océans Canada. Les données de l'Institut canadien de santé animale (ICSA) résument les quantités d'antimicrobiens distribués pour la vente aux fins d'utilisation chez les animaux. Ces données sont acheminées directement au PICRA par l'ICSA.
En complément de la surveillance de l'UAM s'ajoute les résultats des projets de recherche sur l'UAM. Ces projets reposent sur plusieurs espèces animales destinées à la consommation, stades de production et types de production. La recherche, sur l'utilisation des antimicrobiens, comporte le développement d'indicateurs, de méthodes de collecte des données, elle est limitée dans le temps et alimente la surveillance de base.
Autres informations (zone en jaune)
Les données démographiques sont requises pour interpréter les données d'UAM obtenues. Pour les données d'origine humaine, les données démographiques utilisées sont des estimés de la population; pour les données concernant les populations d'animaux de production, ces données correspondent aux nombres de fermes, au nombre d'animaux et à la consommation d'aliments par tête. Les données relatives à la santé animale et à la biosécurité recueillies à la ferme sont également importantes pour interpréter les données d'UAM provenant d'animaux de production (porcs, poulets et dindons).
Integration, rapport et communication (forme en rouge)
Toutes les données d'UAM d'origine animale sont compilées ensemble par les analystes du PICRA alors que les données d'UAM d'origine humaine sont analysées par une autre équipe d'analystes. Toutes les analyses du PICRA (RAM et UAM) de même que celles issues des projets de recherche sur l'AMR ou l'UAM ou encore, des projets sur l'évaluation du risque, sont intégrées ensemble pour être publiées sous forme de rapports et d'autres moyens de communication.
Notes de bas de page
- Note de bas de page 1
-
Sommaire Exécutif accessible à l'adresse : https://www.canada.ca/fr/sante-publique/services/publications/medicaments-et-produits-sante/systeme-canadien-surveillance-resistance-antimicrobiens-2018-rapport-resume.html. Le rapport complet est disponible sur demande.
- Note de bas de page 2
-
Canadian Animal Health Institute - About Us. Accessible à l'adresse : //cahi-icsa.ca/about/. Consultée en octobre 2017.
- Note de bas de page 3
-
European Medicines Agency. European Surveillance of Veterinary Antimicrobial Consumption, 2017-Sales of veterinary antimicrobial agents in 30 European countries in 2015. (EMA/184855/2017). Accessible à l'adresse : http://www.ema.europa.eu/docs/en_GB/document_library/Report/2017/10/WC500236750.pdf. Consultée en octobre 2017.
- Note de bas de page 4
-
European Medicines Agency. European Surveillance of Veterinary Antimicrobial Consumption, 2017-Sales of veterinary antimicrobial agents in 30 European countries in 2015. (EMA/184855/2017). Accessible à l'adresse : http://www.ema.europa.eu/docs/en_GB/document_library/Report/2017/10/WC500236750.pdf. Consultée en octobre 2017.
- Note de bas de page 5
-
Trends in the sales of veterinary antimicrobial agents in nine European countries-Reporting period: 2005-2009. European Medicines Agency. European Surveillance of Veterinary Antimicrobial Consumption (ESVAC). Accessible à l'adresse : www.ema.europa.eu/docs/en_GB/document_library/Report/2011/09/WC500112309.pdf. Consultée en octobre 2017.
- Note de bas de page 6
-
Accessible à l'adresse : http://www.dfo-mpo.gc.ca/aquaculture/management-gestion/apr-rpa-reporting-fra.htm. Consulté en juin 2019.
- Note de bas de page 7
-
Gouvernement du Canada. Biosécurité animale : Norme nationale de biosécurité pour les fermes avicoles. Accessible à l'adresse : www.inspection.gc.ca/DAM/DAM-animals-animaux/STAGING/text-texte/terr_biosec_avian_standard_1375192173847_fra.pdf. Consultée en septembre 2014.
- Note de bas de page 8
-
Government of Canada. Animal biosecurity: National avian on-farm biosecurity standard. Accessible à l'adresse : www.inspection.gc.ca/DAM/DAM-animals-animaux/STAGING/text-texte/terr_biosec_avian_standard_1375192173847_fra.pdf. Consultée en septembre 2014.
- Note de bas de page 9
-
En ce qui concerne le calcul de la quantité d'antimicrobiens utilisés chez les porcs en croissance-finition et les dindons, veuillez consulter la section « Quantité d'antimicrobiens utilisés chez les poulets de chair ».
- Note de bas de page 10
-
Microsoft Excel®2003 et Microsoft Access®2003, Microsoft Corp., Redmond, WA, USA; SAS®9.1, SAS Institute Inc., Cary, NC, USA.
- Note de bas de page 11
-
Cobb-Vantress, Inc. Products: Cobb 500™. Broiler Performance and Nutrition Supplement. Révisé en décembre 2012. Accessible à l'adresse: https://cobb-guides.s3.amazonaws.com/a71b8bc0-bbd4-11e6-bd5d-55bb08833e29.pdf. Consultée en octobre 2017.
- Note de bas de page 12
-
Cobb-Vantress, Inc. Products: Cobb 700™. Broiler Performance and Nutrition Supplement. Révisé en juillet 2015. Accessible à l'adresse: //www.cobb-vantress.com/docs/default-source/cobb-700-guides/cobb700_broiler_performance_nutrition_supplement_english9294AABB12037B70EE475E39.pdf. Consultée en septembre 2016.
- Note de bas de page 13
-
Aviagen. Ross 308. Accessible à l'adresse: http://en.aviagen.com/assets/Tech_Center/Ross_Broiler/Ross-308-Broiler-PO-2014-EN.pdf. Consultée en octobre 2017.
- Note de bas de page 14
-
Aviagen. Ross 708. Accessible à l'adresse: http://en.aviagen.com/assets/Tech_Center/Ross_Broiler/Ross-708-Broiler-PO-2014-EN.pdf. Consultée en octobre 2017.
- Note de bas de page 15
-
Wallenstein Feeds (révisé en mars 2016) et Trouw Nutrition, anciennement connue sous le nom de Nutreco Canada Inc. (version reçue en octobre 2016).
- Note de bas de page 16
-
Information obtenue par Trouw Nutrition, anciennement connue sous le nom de Nutreco Canada Inc. (version reçue en octobre, 2016).
- Note de bas de page 17
-
European Medicines Agency, 2016: Defined daily doses for animals (DDDvet) and defined course doses for animals (DCDvet). European Surveillance of Veterinary Antimicrobial Consumption (ESVAC). Consultée en janvier 2017.
- Note de bas de page 18
-
CFIA, 2016b: Compendium of Medicating Ingredient Brochure. Accessible à l'adresse : http://www.inspection.gc.ca/animals/feeds/medicating-ingredients/eng/1300212600464/1320602461227. Consultée en janvier 2017.
- Note de bas de page 19
-
Canadian Animal Health Institute, 2016: Compendium of Veterinary Products. Accessible à l'adresse : https://bam.naccvp.com/?u=country&p=msds. Consultée en janvier 2017.
- Note de bas de page 20
-
Canadian Association of Poultry Veterinarians. Accessible à l'adresse : http://www.capv-acva.ca/BroilerChicken.htm. Consultée en janvier 2017.
- Note de bas de page 21
-
European Medicines Agency, 2016. European Surveillance of Veterinary Antimicrobial Consumption. Defined daily doses for animals (DDDvet) and defined course doses for animals (DCDvet) (ESVAC)http://www.ema.europa.eu/docs/en_GB/document_library/Other/2016/04/WC500205410.pdf.
- Note de bas de page 22
-
Persoons D, Dewulf J, Smet A, Herman L, Heyndrickx M, Martel A, et al. Antimicrobial use in Belgian broiler production. Prev Vet Med. 2012.
- Note de bas de page 23
-
Timmerman T, Dewulf J, Catry B, Feyen B, Opsomer G, de Kruif A, Maes D. 2006. Quantification and evaluation of antimicrobial drug use in group treatments for fattening pigs in Belgium. Prev. et Med. 74:251-263.
- Note de bas de page 24
-
Collineau L, Belloc C, Stärk KD, Hémonic A, Postma M, Dewulf J, Chauvin C. 2017. Guidance on the Selection of Appropriate Indicators for Quantification of Antimicrobial Usage in Humans and Animals. Zoonoses Public Health. 64:165-184.
- Note de bas de page 25
-
The AACTING-network. Guidelines for collection, analysis and reporting of farm-level antimicrobial use, in the scope of antimicrobial stewardship. http://www.aacting.org/guidelines/. Consultée en mars 2018.
- Note de bas de page 26
-
European Centre for Disease Prevention and Control (ECDC), European Food Safety Authority (EFSA) and European Medicines Agency (EMA). Second joint report on the integrated analysis of the consumption of antimicrobial agents and occurrence of antimicrobial resistance in bacteria from humans and food-producing animals-Joint Interagency Antimicrobial Consumption and Resistance Analysis (JIACRA) Report. Accessible à l'adresse : http://www.ema.europa.eu/docs/en_GB/document_library/Report/2017/07/WC500232336.pdf. Consultée en octobre 2017.
- Note de bas de page 27
-
Version d'avril 2009. Accessible à l'adresse : www.hc-sc.gc.ca/dhp-mps/vet/antimicrob/amr_ram_hum-med-rev-fra.php. Consultée en février 2017.
- Note de bas de page 28
-
Objectifs de performance de Nicolas. http://www.aviagenturkeys.us/uploads/2015/12/21/nicholas_comm_perf_obj_select_2015.pdf. Consultée en octobre, 2017.
- Note de bas de page 29
-
Hybrid turkeys performance goals. http://resources.hybridturkeys.com/commercial/birds. Consultée en octobre, 2017.
- Note de bas de page 30
-
Accessible à l'adresse : http://www.aviagenturkeys.us/uploads/2015/12/21/Aviagen%20Breeder%20Guide%202015.pdf. Consultée en octobre, 2017.
- Note de bas de page 31
-
Rapport de l'enquête nationale sur les laboratoires - 2001, Études nationales sur les maladies gastro-intestinales aiguës, Division des entéropathies et des maladies d'origine hydrique et alimentaire, 2002.
- Note de bas de page 32
-
Ravel, A. Antimicrobial Surveillance in food at retail - Proposal for a pilot project. 2002, 13 p.
- Note de bas de page 33
-
Aucun échantillonnage de viande vendue au détail n'a été effectué au Manitoba à ce jour ou en Saskatchewan en 2017.
- Note de bas de page 34
-
Aucun échantillonnage de viande vendue au détail n'a été effectué à Terre-Neuve-et-Labrador.
- Note de bas de page 35
-
En 2017, en raison du manque de disponibilité des techniciens pour effectuer les échantillonnages, une année partielle d'échantillonnage a été effectuée en Ontario et dans les Prairies. Les objectifs d'échantillonnage et de rendement en matière de détection bactérienne n'ont donc pas été atteints. L'ensemble des données d'Ontario et des Prairies de 2017 doivent être interprétées avec prudence. De plus les activités d'échantillonnage de la région de l'Atlantique n'ont pas eu lieu en 2017 en raison de contraintes budgétaires.
- Note de bas de page 36
-
Dans 1 magasin de chacune des divisions (à l'exception de la région de l'Atlantique), on a volontairement omis de prélever l'échantillon de viande de bœuf afin de minimiser les risques de suréchantillonnage de ce type de viande.
- Note de bas de page 37
-
En 2017, en raison du manque de disponibilité des techniciens pour effectuer les échantillonnages, une année partielle d'échantillonnage a été effectuée en Ontario et dans les Prairies. Les objectifs d'échantillonnage et de rendement en matière de détection bactérienne n'ont donc pas été atteints. L'ensemble des données d'Ontario et des Prairies de 2017 doivent être interprétées avec prudence. De plus les activités d'échantillonnage de la région de l'Atlantique n'ont pas eu lieu en 2017 en raison de contraintes budgétaires.
- Note de bas de page 38
-
Ertco Data LoggerTM, West Patterson, New Jersey, États-Unis.
- Note de bas de page 39
-
Agriculture et Agroalimentaire Canada. Information sur le marché des viandes rouges. Disponible au : agriculture.canada.ca/redmeat-vianderouge/index_fra.htm. Consultée en septembre 2014.
- Note de bas de page 40
-
Ravel, A. Élaboration d'un système national de surveillance de l'antimicrobiorésistance dans le secteur agroalimentaire - Options de conception de l'échantillonnage. Présenté au Comité national directeur sur la résistance chez les microorganismes entériques. Canada, 2001. 79 p.
- Note de bas de page 41
-
Kauffman F. The Bacteriology of Enterobacteriaceae. Baltimore: Williams and Wilkins Co, 1966.
- Note de bas de page 42
-
Ewing WH. Edwards and Ewing's Identification of Enterobacteriaceae. 4e éd. New York. Elsevier Science Publishing Co, 1986.
- Note de bas de page 43
-
Le Minor L. Guide pour la préparation des sérums anti‐Salmonella. Paris, France. Centre Collaborateur de l'OMS de Référence et de Recherche pour les Salmonella, Institut Pasteur, 2001.
- Note de bas de page 44
-
Murray PR, Baron EJ, Pfaller MA et al., éditeurs. Manual of Clinical Microbiology. 8e éd. Washington DC, ASM Press, 2005.
- Note de bas de page 45
-
Compendium de méthodes, Direction générale de la protection de la santé, Méthodes officielles pour l'analyse microbiologique des aliments, gouvernement du Canada.
- Note de bas de page 46
-
Lorsque des cuisses avec la peau n'étaient pas disponibles, des ailes avec la peau ou d'autres coupes de poulet ont été achetées.
- Note de bas de page 47
-
API®20E system.
- Note de bas de page 48
-
The multiplex PCR speciation of Campylobacter jejuni and Campylobacter coli was based on the following published method. Person S, KE Olsen. Multiplex PCR for identification of Campylobacter coli and Campylobacter jejuni from pure cultures and directly on stool samples. J Med Microbiol 2005; 54:1043-1047.
- Note de bas de page 49
-
Qiagen®. QIAxcel®DNA Handbook, 5th Edition November 2014. Disponible au : https://www.qiagen.com/ca/resources/resourcedetail?id=f6158498-a857-4a2f-b40b-569fba3793e2&lang=en. Consultée en octobre 2016.
- Note de bas de page 50
-
Grimont PAD, Weill F-X. Antigenic formulae of the Salmonella serovars. 9e éd., Paris, France. Centre collaborateur de l'OMS de référence et de recherche pour les Salmonella, Institut Pasteur, 2007.
- Note de bas de page 51
-
Ewing WH. Edwards and Ewing's Identification of Enterobacteriaceae. 4e éd. New York: Elsevier Science Publishing Co, 1986.
- Note de bas de page 52
-
Le sérotype de Salmonella Paratyphi B n'inclut pas le S. Paratyphi B var. L (+) tartrate (+), anciennement nommé S. Paratyphi var. Java. Le biotype de S. Paratyphi B inclus ici est tartrate (‐) et il est associé à une fièvre pseudotyphoïde plus grave. Salmonella Paratyphi B var. L (+) tartrate (+) est souvent associé aux maladies gastro‐intestinales.
- Note de bas de page 53
-
Anderson E, Williams R. Bacteriophage typing of enteric pathogens and staphylococci and its use in epidemiology. J Clin Pathol 1956; 9: 94-127.
- Note de bas de page 54
-
Difcoä phage broth, Difco Laboratories, Baltimore, MD; pH 6,8.
- Note de bas de page 55
-
Difcoä phage agar, Difco Laboratories.
- Note de bas de page 56
-
Farmer J, Hickman F, Sikes J. Automation of Salmonella Typhi phage-typing. Lancet 1975; 2(7939): 787-790.
- Note de bas de page 57
-
Anderson E, Williams R. Bacteriophage typing of enteric pathogens and staphylococci and its use in epidemiology. J Clin Pathol 1956; 9: 94-127.
- Note de bas de page 58
-
Ward L, de Sa J, Rowe B. A phage-typing scheme for Salmonella Enteritidis. Epidemiol Infect 1987; 99: 291-294.
- Note de bas de page 59
-
Callow B. A new phage typing scheme for Salmonella Typhimurium. J Hyg (Lond) 1959; 57: 346-359.
- Note de bas de page 60
-
Anderson E. The phagetyping of Salmonella other than S. Typhi. In: Van Oye E, ed. The World Problem of Salmonellosis. The Hague, The Netherlands: Dr W. Junk Publishers, 1964; 89-100.
- Note de bas de page 61
-
Anderson E, Ward L, de Saxe M, et al. Bacteriophage-typing designations of Salmonella Typhimurium. J Hyg (Lond) 1977; 78: 297-300.
- Note de bas de page 62
-
Demczuk W, Soule G, Clark C, et al. Phage-based typing scheme for Salmonella enterica serovar Heidelberg, a causative agent of food poisonings in Canada. J Clin Microbiol 2003; 41: 4279-4284.
- Note de bas de page 63
-
Office Internationale des Épizooties (OIÉ); Organisation mondiale pour la santé animale, Laboratoire de référence pour les Salmonelloses, Guelph, Ontario.
- Note de bas de page 64
-
Yoshida C., et al. Multi-laboratory evaluation of the rapid genoserotyping array (SGSA) for the identification of Salmonella serovars. Diag Microbiol & Infect Dis 2014; 80:185-190.
- Note de bas de page 65
-
Ewing WH. Edwards and Ewing's Identification of Enterobacteriaceae. 4th ed. New York: Elsevier Science Publishing Co, 1986.
- Note de bas de page 66
-
Shipp C, Rowe B. A mechanised microtechnique for Salmonella serotyping. J Clin Pathol 1980; 33: 595-597.
- Note de bas de page 67
-
Grimont PAD, Weill F-X. Antigenic Formulae of the Salmonella Serovars. 9th ed. Cedex, France: Collaborating Center for Reference and Research on Salmonella, Institut Pasteur, 2007.
- Note de bas de page 68
-
Anderson E, Williams R. Bacteriophage typing of enteric pathogens and staphylococci and its use in epidemiology. J Clin Pathol 1956; 9: 94-127.
- Note de bas de page 69
-
Clinical and Laboratory Standards Institute (CLSI) M7‐A10.
- Note de bas de page 70
-
SensititreMC Trek Diagnostic Systems Ltd, West Sussex, England.
- Note de bas de page 71
-
SensititreMC Trek Diagnostic Systems Ltd, West Sussex, England.
- Note de bas de page 72
-
ARISMC Trek Diagnostic Systems Ltd, West Sussex, England.
- Note de bas de page 73
-
CLSI M100‐S27.
- Note de bas de page 74
-
CLSI M45‐A3.
- Note de bas de page 75
-
SensititreMC, Trek Diagnostic Systems Ltd, West Sussex, England.
- Note de bas de page 76
-
CLSI M45‐A3.
- Note de bas de page 77
-
Oracle®, Oracle Corp., Redwood Shores, CA, USA.
- Note de bas de page 78
-
SAS®9.3, SAS Institute Inc., Cary, NC, USA.
- Note de bas de page 79
-
SAS®9.3; and Stata®13 SE, Stata Corp., College Station, TX, USA.
- Note de bas de page 80
-
Microsoft®Excel 2010, Microsoft Corp.
- Note de bas de page 81
-
PROC GENMOD, SAS®9.3.
- Note de bas de page 82
-
Stata®13 SE.
- Note de bas de page 83
-
Santé Canada. 2009. Catégorisation des médicaments antimicrobiens basée sur leur importance en médecine humaine. Version avril 2009. Accessible à l'adresse : https://www.canada.ca/fr/santecanada/categorisation‐medicaments‐antimicrobiens‐basee‐leur‐importance‐medecine‐humaine.html. Consultée en juillet 2017.
- Note de bas de page 84
-
Le ceftiofur est homologué pour une utilisation chez les animaux seulement. La résistance au ceftiofur est généralement observée en combinaison avec de la résistance à l'amoxicilline‐acide clavulanique, à la céfoxitine, à l'ampicilline et à la ceftriaxone (profil de résistance A2C‐AMP‐CRO).
- Note de bas de page 85
-
Centre collaborateur de l'Organisation mondiale de la Santé pour la méthodologie sur l'établissement des statistiques concernant les produits médicamenteux. ATCvet. Accessible à l'adresse : www.whocc.no/atcddd. Consultée en mai 2017.
- Note de bas de page 86
-
Centre collaborateur de l'Organisation mondiale de la Santé pour la méthodologie sur l'établissement des statistiques concernant les produits médicamenteux. ATCvet. Accessible à l'adresse : www.whocc.no/atcddd. Consultée en mai 2017.
- Note de bas de page 87
-
Centre collaborateur de l'Organisation mondiale de la Santé pour la méthodologie sur l'établissement des statistiques concernant les produits médicamenteux. Accessible à l'adresse : www.whocc.no/atcddd. Consultée en mai 2017.
- Note de bas de page 88
-
En 2017, toutes les provinces ne sont pas représentées pour les composantes faisant l'objet d'une surveillance dans les Prairies et la région des provinces de l'Atlantique.
- Note de bas de page 89
-
Association canadienne des vétérinaires aviaires. Accessible à l'adresse : http://www.capv-acva.ca/BroilerChicken.htm. Consultée en janvier 2017.
- Note de bas de page 90
-
Inspection canadienne d'inspection des aliments, 2016b: Recueil des notices sur les substances médicatrices. Accessible à l'adresse : //www.inspection.gc.ca/animals/feeds/medicating-ingredients/fra/1300212600464/1320602461227. Consultée en janvier 2017.
- Note de bas de page 91
-
Institut canadien de la santé animale, 2016: Compendium of Veterinary Products. Accessible à l'adresse : https://bam.naccvp.com/?u=country&p=msds. Consultée en janvier 2017.
- Note de bas de page 92
-
Inspection canadienne d'inspection des aliments, 2016b: Recueil des notices sur les substances médicatrices. Accessible à l'adresse : //www.inspection.gc.ca/animals/feeds/medicating-ingredients/fra/1300212600464/1320602461227. Consultée en janvier 2017.
- Note de bas de page 93
-
Institut canadien de la santé animale, 2016: Compendium of Veterinary Products. Accessible à l'adresse : https://bam.naccvp.com/?u=country&p=msds. Consultée en janvier 2017.
- Note de bas de page 94
-
Accessible à l'adresse : http://www.ema.europa.eu/ema/index.jsp?curl=pages/includes/document/document_detail.jsp?webContentId=WC500184369&mid=WC0b01ac058009a3dc. Consulté en janvier 2017.
- Note de bas de page 95
-
Accessible à l'adresse : http://www.sites.ext.vt.edu/newsletter-archive/livestock/aps-06_07/aps-349.html. Accessed on January 2017. Disponible au : http://www.sites.ext.vt.edu/newsletter-archive/livestock/aps-06_07/aps-349.html. Consulté en janvier 2017.
- Note de bas de page 96
-
Accessible à l'adresse : http://www.ema.europa.eu/ema/index.jsp?curl=pages/includes/document/document_detail.jsp?webContentId=WC500184369&mid=WC0b01ac058009a3dc. Consultée en janvier 2017.