Programme National de Surveillance des Maladies Entériques (PNSME) Sommaire Annuel 2020

Téléchargez le format alternative
(Format PDF, 6 Mo, 44 pages)
Organization : Agence de la santé publique du Canada
Publiée : 2024-XX-XX
Incluant des tableaux sur les sérotypes pour l’année 2020, PNSME et le Laboratoire national de microbiologie (LNM)
Préparé par :
Le Laboratoire national de microbiologie (LNM), le Centre des maladies infectieuses d’origine alimentaire, environnementale et zoonotique (CMIOAEZ), Agence de la santé publique du Canada et les laboratoires provinciaux de santé publique
Remerciements
Programme national de surveillance des maladies entériques (PNSME) équipe de coordination :
- Céline Nadon, chef, Surveillance, Section de la détection des éclosions et de l’intervention, Division des maladies entériques, Laboratoire national de microbiologie (LNM)
- Sara Christianson, chef, Section des services de référence et de diagnostic, Division des maladies entériques, LNM
- Lori Lozinski, commis à la surveillance, Division des maladies entériques, LNM
- Kate Thomas, gestionnaire, Division de la surveillance des maladies d’origine alimentaire et de la résistance aux antimicrobiens (DSMOARA), Centre des maladies infectieuses d’origine alimentaire, environnementale et zoonotique (CMIOAEZ)
- Symron Bansal, épidémiologiste, DSMOARA, CMIOAEZ
- Lauren Sherk, épidémiologiste, DSMOARA, CMIOAEZ
Laboratoires partenaires provinciaux :
- Laboratoire de santé publique du centre de lutte contre la maladie de Colombie-Britannique
- Alberta Precision Laboratories
- Laboratoire de lutte contre les maladies de la Saskatchewan
- Laboratoire provincial de microbiologie de Cadham (Manitoba)
- Santé publique Ontario
- Laboratoire de santé publique du Québec (LSPQ)
- Laboratoires de santé publique du Nouveau-Brunswick
- Laboratoires de santé publique de la Nouvelle-Écosse
- Laboratoires de santé publique de l’Île-du-Prince-Édouard
- Laboratoire de santé publique de Terre-Neuve-et-Labrador
Partenaires provinciaux et territoriaux en épidémiologie :
- Centre de contrôle des maladies de la Colombie-Britannique
- Ministère de la Santé de l’Alberta
- Ministère de la Santé de la Saskatchewan
- Santé Manitoba
- Santé publique Ontario
- Ministère de la Santé et des Services sociaux du Québec
- Ministère de la Santé du Nouveau-Brunswick
- Ministère de la Santé et du Mieux-être de la Nouvelle-Écosse
- Ministère de la Santé et du Mieux-être de l’Île-du-Prince-Édouard
- Ministère de la Santé et des Services communautaires de Terre-Neuve-et-Labrador
- Ministère de la Santé et des Services sociaux du Yukon
- Ministère de la Santé et des Services sociaux des Territoires du Nord-Ouest
- Ministère de la Santé et des Services sociaux du Nunavut
Aperçu
Le Programme national de surveillance des maladies entériques (PNSME) est axé sur la collaboration entre l'Agence de la santé publique du Canada (ASPC) et les laboratoires provinciaux de santé publique. Au moyen du PNSME, des analyses sont effectuées et des rapports sont produits toutes les semaines pour 14 microorganismes différents causant des maladies entériques, dont 10 doivent être obligatoirement déclarés à l'échelle nationale. Les données obtenues de ce système de surveillance soutiennent la détection des grappes et des éclosions multiprovinciales, orientent les interventions en matière de santé publique et sont conçues pour s'intégrer aux efforts nationaux et internationaux visant à limiter la transmission des maladies entériques.
En 2020, 10 000 résultats d'analyses d'isolats ont été rapportés, ce qui représente une baisse de 35 % par rapport au nombre moyen de déclarations reçues au cours des cinq années précédentes (15 340). Cette diminution s'explique probablement, en partie, par les effets de la pandémie de COVID-19. Salmonella spp. continue d'être l'organisme le plus fréquemment identifié, 4 919 déclarations ayant été envoyés en 2020, ce qui représente 49 % de tous les isolats déclarés. Comme pour les années précédentes, Salmonella Enteritidis (29 %) et S. Typhimurium (10 %) comptent parmi les trois principaux sérotypes de l'ensemble des sérotypes de Salmonella déclarés en 2020. S. Newport (14 %) était l'un des trois sérotypes les plus fréquemment détectés en 2020 en raison d'une éclosion multijuridictionnelle survenue à l'été 2020. Ensemble, ces trois sérotypes représentent 53 % de tous les sérotypes de Salmonella identifiés.
Le taux d'incidence d'Escherichia coli producteurs de Shigatoxines (ECST) O157 a diminué par rapport à un taux relativement stable entre 2010 et 2019, alors que 0,62 cas pour 100 000 personnes a été signalé en 2020. Une diminution a également été observée dans le taux d'incidence des isolats d'ECST autres que O157 déclarés en 2020 (0,84 cas pour 100 000 personnes) par rapport à un taux élevé de 1,58 cas pour 100 000 personnes en 2019. Il s'agit de la quatrième année consécutive où l'on a rapporté moins d'isolats d'E. coli O157 que d'isolats d'ECST autres que O157.
Le taux d'incidence de la listériose invasive en 2020 (0,42 cas pour 100 000 personnes) est similaire à celui observé au cours des deux dernières années. Au cours de la période de huit ans pendant laquelle l'hépatite A a fait l'objet d'une surveillance nationale, c'est en 2019 que l'incidence la plus élevée a été signalée au programme (1,55 cas pour 100 000 personnes) alors que le taux d'incidence en 2020 était de 0,67 cas pour 100 000 personnes. Contrairement aux années précédentes au cours desquelles Shigella sonnei était l'espèce la plus souvent signalée parmi les espèces de Shigella, en 2020, Shigella flexneri a représenté 57 % de toutes les espèces de Shigella signalées, et le taux d'incidence de Shigella flexneri (0,59 cas pour 100 000 personnes) était également supérieur à celui de Shigella sonnei (0,40 cas pour 100 000 personnes). Les tendances relatives à toutes les autres espèces de Shigella étaient à la baisse en 2020 par rapport aux années précédentes.
Table des matiéres
- Remerciements
- Aperçu
- À propos du Programme national de surveillance des maladies entériques (PNSME)
- Nombre d’isolats confirmés en laboratoire et taux d’incidence
- Salmonella
- Escherichia coli
- Listeria monocytogenes
- Shigella
- Hépatite A
Tableaux
- Tableau 1. Enquêtes sur les éclosions multijuridictionnelles survenues en 2020
- Tableau 2. Nombre d'isolats déclarés au PNSME par principaux groupes d'organismes, par province ou territoire, 2020
- Tableau 3. Totaux nationaux annuels et taux (pour 100 000 personnes) des groupes d'entéropathogènes et de microorganismes déclarés au PNSME de 2015 à 2020
- Tableau 4. Taux annuels (pour 100 000 personnes) d'infection par certains groupes d'agents pathogènes régulièrement déclarés au PNSME en 2020, par province/territoire
- Tableau 5. Nombre d'isolats des dix principaux sérotypes de Salmonella déclarés au PNSME en 2020, par province/territoire
- Tableau 6. Nombre total de cas au Canada (classement général) pour les dix sérotypes de Salmonella les plus fréquemment déclarés au PNSME de 2015 à 2020
Figures
- Figure 1. Proportion des sérotypes de Salmonella responsables de maladies humaines déclarés au PNSME, 2020 (n = 4 919)
- Figure 2. Nombre annuel d'isolats (2011 à 2020) des cinq principaux sérotypes de Salmonella déclarés au PNSME en 2020
- Figure 3. Taux d'incidence relative (pour 100 000 personnes) de S. Enteritidis, de S. Newport, de S. Typhimurium et d'autres sérotypes de Salmonella déclarés au PNSME, par année, de 2016 à 2020, comparativement à la période de référence de 2011 à 2015
- Figure 4. Taux d'incidence (pour 100 000 personnes) des sérotypes d'E. coli O157, d'ECST autres que O157 et autre d'E. coli non typés qui ont été déclarés au PNSME, de 1997 à 2020
- Figure 5. Répartition des sérotypes d'ECST autres que O157 déclarés au PNSME en 2020 (n = 513)
- Figure 6. Taux d'incidence (pour 100 000 personnes) des cinq sérotypes d'ECST autres que O157 les plus déclarés au PNSME, de 2011 à 2020
- Figure 7. Taux d'incidence (pour 100 000 personnes) de la listériose invasive déclarés au PNSME par province, de 2011 à 2020
- Figure 8. Taux d'incidence (pour 100 000 personnes) des espèces de Shigella déclarées au PNSME, de 1997 à 2020
- Figure 9. Taux d'incidence (pour 100 000 personnes) nationaux et provinciaux de l'hépatite A déclarés au PNSME, de 2013 à 2020
Annexes
- Annexe A. Comparaison des nombres totaux, des taux d'incidence (pour 100 000 personnes) et des pourcentages de cas de maladies entériques enregistrés au Canada, en 2019, par le Système de surveillance des maladies à déclaration obligatoire du Canada (SSMDOC) et le PNSME
- Annexe B. Données sur les espèces et les sérotypes déclarés au PNSME par province et territoire, 2020
- Annexe C : Effets de la pandémie de COVID-19 – Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 en ce qui concerne certains agents pathogènes
- Figure 10. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, tous les sérotypes de Salmonella, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 11. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, tous les sérotypes de Salmonella, à l'exception de S. Enteritidis, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 12. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, S. Enteritidis, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 13. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, E. coli O157, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 14. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, ECST autres que O157, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 15. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, Listeria monocytogenes, combinés au nombre de cas de COVID-19, au Canada, en 2020
- Figure 16. Comparaison du nombre d'isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019, Shigella, combinés au nombre de cas de COVID-19, au Canada, en 2020
À propos du Programme national de surveillance des maladies entériques (PNSME)
Au Canada, la surveillance des maladies entériques est effectuée par le PNSME et le Système de surveillance des maladies à déclaration obligatoire du Canada (SSMDOC)Note de bas de page 1. Le PNSME est administré conjointement par le Laboratoire national de microbiologie (LNM) et le Centre des maladies infectieuses d’origine alimentaire, environnementale et zoonotique (CMIOAEZ) de l’ASPC. Depuis 1997, les analyses et les rapports hebdomadaires sur les cas de maladies entériques confirmés en laboratoire par les laboratoires provinciaux de santé publique sont effectués par le PNSME.
Le PNSME identifie le premier niveau de caractérisation (l’espèce et le sérotype) de données qui sont essentielles et intégrées aux autres programmes de surveillance. Le suivi des données agrégées permet une évaluation et une réponse rapide aux éclosions de maladie entériques. De plus, ces données permettent de décrire les tendances des sous-types des agents pathogènes et l’incidence des entéropathogènes à déclaration obligatoire au Canada. Le SSMDOC reçoit les données recueillies par les services de santé locaux, acheminées aux autorités sanitaires provinciales ou territoriales et agrégées par le Centre de la lutte contre les maladies transmissibles et les infections (CLMTI) de l’ASPC. Ces données peuvent être plus représentatives du nombre total de maladies annuelles, cependant le SSMDOC n’est pas conçu pour fournir rapidement les informations nécessaires à la détection des agrégats ou des éclosions. Ces deux systèmes de surveillance (le SSMDOC et le PNSME) se complètent l’un l’autre pour fournir des résultats épidémiologiques et de laboratoire, cependant des différences existent entre eux. En raison de ses protocoles et de ses normes en matière de déclaration, le SSMDOC est plus fiable pour ce qui est du nombre total de maladies, tandis que le PNSME est plus à jour et suit les tendances de plus près. Une comparaison des nombres de cas et des taux d’incidence à l’échelle nationale pour les maladies entériques est incluse à l’annexe A.
Le PNSME est également hautement complémentaire d’un autre système de surveillance en laboratoire : PulseNet CanadaNote de bas de page 2. Aussi administré par l’ASPC, PulseNet Canada recueille en temps réel des données à haute résolution sur les cas de maladies entériques afin de détecter les éclosions et d’intervenir. En raison des tests supplémentaires que PulseNet Canada effectue (le typage moléculaire ou génomique), il existe des différences dans le délai d’exécution par rapport aux données de PNSME hebdomadaires. De plus, PulseNet ne surveille qu’une partie des organismes suivis par le PNSME.
Collecte des données
Les isolats (ou les spécimens) sont soumis aux laboratoires provinciaux de santé publique aux fins d'identification ou de confirmation de l'entéropathogène. Une fois par semaine, chaque laboratoire provincial de santé publique consigne le nombre de microorganismes entériques isolés chez des patients humains. Les renseignements précisent le genre, l'espèce et le sérotype (le cas échéant). La « semaine de rapports » du PNSME couvre la période du dimanche au samedi en fonction de la date à laquelle l'analyse de laboratoire a été réalisée, à l'exception de l'Alberta où la semaine de rapports est établie en fonction de la date à laquelle l'analyse a été reçue. Les données sont transmises directement au LNM, par télécopieur ou courriel, ou par leur saisie au moyen de l'application Web (PNSME-Web) hébergée sur le Réseau canadien de renseignements sur la santé publique (RCRSP). Les informations sont acheminées le plus rapidement possible, au plus tard, le deuxième jour suivant une fin de semaine ou un jour férié. Une exception à ce système de déclaration se produit lorsque l'isolat doit être envoyé à un autre laboratoire pour terminer l'identification. Dans ce cas, l'isolat est signalé au niveau de typage ou d'identification atteint (p. ex., Salmonella sp.) pour la semaine au cours de laquelle il a été envoyé au laboratoire de référence. L'enregistrement du PNSME est mis à jour lorsque le laboratoire de référence achemine les dernières données sur l'identification (p. ex., le rapport de la semaine 35 indique qu'un cas de « Salmonella sp. » signalé à la semaine 33 a été confirmé comme étant S. Banana). Cette mise à jour figure dans le rapport hebdomadaire du PNSME subséquent.
Toutes les données soumises sont agrégées par province et par agent pathogène et ne contiennent aucune information permettant d'identifier un patient, ni aucun renseignement de nature confidentielle. Les partenaires du PNSME tentent de n'inclure que les nouveaux isolats décelés au laboratoire dans la semaine ou les mises à jour des chiffres précédemment transmis. Pour éviter les doublons, le laboratoire provincial de santé publique recherche les échantillons multiples, les échantillons répétés ou les échantillons provenant d'une même personne faisant l'objet d'un suivi, et considère comme un seul cas tous les isolats identiques provenant d'un même patient et recueillis sur une période de trois mois.
Les données recueillies aux fins de surveillance sont de plus en plus souvent obtenues par séquençage du génome entier (SGE) plutôt que par des méthodes microbiologiques traditionnelles. Cela dit, la plupart des données du PNSME peuvent provenir des données de la séquence du génome entier in silicio (identification de l'espèce, sérotype). Ainsi, plus de deux décennies de données servant aux analyses du PNSME demeureront compatibles avec la surveillance à l'ère de la génomique. Dès 2018, une partie des données recueillies et analysées par le PNSME auraient été générées par SGE.
Analyse et diffusion des données
L'analyse des données est réalisée chaque semaine à l'aide d'un algorithme afin de déterminer si les nombres de cas hebdomadaires actuels sont significativement plus élevés que les nombres attendus. La signification statistique repose sur la probabilité cumulative de la loi de Poisson entre le nombre de cas déclarés et le nombre médian rétrospectif sur cinq ans.
Les résultats de l'analyse hebdomadaire inclus dans le « rapport hebdomadaire du PNSME » sont envoyés à tous les laboratoires provinciaux de santé publique, à, au moins, un épidémiologiste ou un médecin-hygiéniste de chaque province ou territoire et à plusieurs intervenants du gouvernement fédéral. Bien que le protocole permette la transmission des rapports à d'autres professionnels de la santé publique qui ont besoin de cette information dans un but opérationnel, les rapports hebdomadaires ne sont pas destinés à une diffusion publique. Les professionnels de la santé publique ne sont pas tenus d'intervenir à la suite des hausses statistiques indiquées dans les rapports. Ces rapports visent à fournir en temps opportun des renseignements utiles aux responsables des interventions en santé publique.
En plus des rapports hebdomadaires du PNSME, les partenaires peuvent analyser les données en temps réel, évaluer les tendances et afficher les données pour leur territoire de compétence respectif dans le PNSME-Web. PulseNet Canada se sert de ces données, en plus des données de laboratoire sur les empreintes génétiques (électrophorèse en champ pulsé ou ECP) et d'autres données moléculaires ou génomiques, pour déceler les grappes de cas et les éclosions. Les analyses des données obtenues sont également publiées sur le RCRSP où peuvent les consulter les laboratoires provinciaux de santé publique, l'Agence canadienne d'inspection des aliments (ACIA), Santé Canada, l'ASPC et les épidémiologistes des provinces et des territoires. L'évaluation coordonnée des résultats de laboratoire obtenus par ces réseaux de surveillance en laboratoire permet d'interpréter les données microbiologiques cliniques pendant les enquêtes épidémiologiques multijuridictionnelles conformément aux Modalités canadiennes d'intervention lors de toxi-infection d'origine alimentaire (MITIOA)Note de bas de page 3.
Limites
Il convient de noter que ces données comportent des limites inhérentes. Pour certains microorganismes, le nombre d'isolats rapportés ne représente qu'un sous-ensemble des isolements réalisés en laboratoire et peut ne pas refléter l'incidence de la maladie à l'échelle provinciale ou nationale. Par exemple, les isolats de Campylobacter ne sont pas systématiquement acheminés au laboratoire provincial de santé publique ou au laboratoire de référence central pour des analyses supplémentaires au-delà de la caractérisation du genre ou de l'espèce. Ces isolats sont par conséquent largement sous-représentés dans le PNSME. En revanche, le nombre d'isolats de Salmonella et d'E. coli O157 enregistrés par le PNSME est plus représentatif de l'incidence réelle des la maladies au Canada, puisque le nombre de cas signalés au SSMDOC et le nombre d'isolats déclarés au PNSME présentent un degré élevé de concordance pour les deux maladies. Certains organismes peuvent être surreprésentés dans le PNSME si plusieurs échantillons provenant d'un même patient sont signalés, mais des mesures sont prises pour réduire ce phénomène. Les données relatives aux sites d'isolement extra-intestinaux et aux voyages à l'étranger ne sont pas transmises de façon systématique au PNSME par tous les laboratoires provinciaux de santé publique, et toute interprétation doit donc être considérée avec prudence.
En mars 2020, la pandémie de COVID-19 a été déclaréeNote de bas de page 4 et des mesures de santé publique ont été prises à l'échelle mondiale pour y faire face. Partout au Canada et dans certains territoires, provinces et régions, diverses mesures de santé publique ont été mises en place. Ces mesures de santé publique et les mesures d'adaptation mises en œuvre par les canadiens dans le contexte de la pandémie de COVID-19 ont non seulement permis de réduire la transmission du virus responsable de la maladie, mais ont également eu un impact sur d'autres maladies infectieuses signalées à divers degrés. Les données du PNSME pour 2020 doivent être interprétées en tenant compte des mesures de santé publique et des mesures d'adaptation adoptées dans le contexte de la pandémie de COVID-19 (annexe C).
Toute question ou correspondance peut être transmise par courriel aux adresses suivantes :
| Enquêtes sur les éclosions multijuridictionnelles | Nombre de cas – dernières données (Canada seulement) | Date de survenue du premier cas | Date de survenue du dernier cas | Provinces et territoires ayant signalé des cas |
|---|---|---|---|---|
| [2020-058] [Comité de coordination de l'enquête sur l'éclosion (CCEE) : grappe de cas d'hépatite A en C.-B., en Alb., en Ont. et au N.-B.] [févr. à juin] | 13 | 2019-07-28 | 2020-03-23 | C.-B. 2 Alb. 1 Ont. 9 N.-B. 1 |
| [2020-116] [CCEE : S. Typhimurium en C.-B., en Alb., en Sask., en Ont., au Qc et au N.-B.] [avr. à déc.] | 31 | 2017-06-01 | 2020-10-15 | C.-B. 3 Alb. 6 Sask. 1 Ont. 4 Qc 16 N.-B. 1 |
| [2020-133] [CCEE : E. coli O157 en Alb., en C.-B. et au Man. (anciennement CCEE 2019-212)] [mai à août] | 5 | 2020-03-08 | 2020-08-02 | C.-B. 2 Alb. 2 Man. 1 |
| [2020-137] [CCEE : S. Typhimurium en Alb., en C.-B. et au Yn] [juin à déc.] | 10 | 2020-02-28 | 2020-09-30 | C.-B. 5 Alb. 4 Yn 1 |
| [2020-148] [CCEE : cyclosporose contractée localement en Ont., au Qc, à T.-N.-L., au N.-B., en C.-B. et au Nt] [juin à oct.] | 399 | 2020-05-15 | 2020-10-08 (date de prélèvement de l'échantillon) | C.-B. 1 Ont. 283 Qc 105 N.-B. 2 T.-N.-L. 6 Nt 2 |
| [2020-151] [CCEE : S. Newport en Alb., en C.-B., au Man., en Ont., en Sask., à l'Î.-P.-É., au Qc et aux États-Unis] [juill. à sept.] | 515 | 2020-06-15 | 2020-08-29 | C.-B. 121 Alb. 293 Sask. 35 Man. 26 Ont. 14 Qc 25 Î.-P.-É. 1 |
| [2020-175] [CCEE : S. Enteritidis en Ont., au Qc et aux États-Unis] [août à oct.] | 57 | 2020-06-30 (date de prélèvement de l'échantillon) | 2020-09-03 (date d'isolement) | Ont. 41 Qc 16 |
| [2020-216] [CCEE : S. Oranienburg en Ont., au Qc et au N.-B.] [sept. à nov.] | 10 | 2020-08-11 | 2020-09-22 | Ont. 4 Qc 5 N.-B. 1 |
| [2020-217] [CCEE : Vibrio parahaemolyticus au N.-B., à l'Î.-P.-É., au Qc et en Sask.] [sept. à déc.] | 23 | 2020-07-03 | 2020-11-02 (date d'isolement) | Sask. 1 Qc 7 N.-B. 10 Î.-P.-É. 5 |
| [2020-239] [CCEE : E. coli O157 en C.-B., en Alb. et aux États-Unis] [oct. à déc.] | 5 | 2020-07-08 | 2020-09-21 | C.-B. 4 Alb. 1 |
| [2020-255] [CCEE : E. coli O157:H7 en C.-B., au Man. et aux États-Unis] [nov. à janv.] | 4 | 2020-10-10 | 2020-11-12 | C.-B. 1 Man. 3 |
Nombre d’isolats confirmés en laboratoire et taux d’incidence
En 2020, les laboratoires provinciaux de santé publique ont transmis les résultats de 10 000 isolats d'entéropathogènes au PNSME, ce qui représente une diminution par rapport au nombre moyen de déclarations au cours des cinq années précédentes (15 340). Le groupe d'entéropathogènes le plus souvent signalé était Salmonella, suivi des virus entériques (norovirus, hépatite A, rotavirus et adénovirus) et de Campylobacter (tableau 2). L'annexe B présente le nombre d'isolats d'organismes déclarés par province et par territoire en 2020.
| Groupetableau 2 note 4 | C.-B. | Alb. | Sask | Man. | Ont. | Qc | N.-B. | N.-É. | Î.-P.-É. | T.-N.-L. | Yn | T. N.-O. | Nt | Total | % du total des isolats déclarés |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Salmonella | 760 | 741 | 148 | 180 | 1 631 | 968 | 122 | 146 | 11 | 209 | 2 | 1 | 0 | 4 919 | 49,19 |
| Campylobactertableau 2 note 1 | 25 | 252 | 169 | 48 | 87 | 208 | 252 | 90 | 48 | 105 | 4 | 1 | 0 | 1 289 | 12,89 |
| Virustableau 2 note 1 | 112 | 78 | 40 | 113 | 382 | 34 | 56 | 53 | 19 | 178 | 2 | 0 | 0 | 1 067 | 10,67 |
| Parasitestableau 2 note 1 | 144 | 10 | 42 | 68 | 467 | NStableau 2 note 3 | 83 | 95 | 14 | 83 | 10 | 1 | 0 | 1 017 | 10,17 |
| E. colitableau 2 note 2 | 97 | 201 | 42 | 40 | 124 | 107 | 1 | 1 | 1 | 216 | 0 | 0 | 0 | 830 | 8,30 |
| Shigella | 56 | 37 | 7 | 10 | 156 | 121 | 0 | 2 | 0 | 3 | 0 | 1 | 0 | 393 | 3,93 |
| Yersinia | 101 | 27 | 10 | 4 | 114 | 18 | 2 | 3 | 0 | 2 | 2 | 0 | 0 | 283 | 2,83 |
| Listeria | 12 | 9 | 6 | 7 | 66 | 44 | 6 | 5 | 1 | 2 | 0 | 0 | 0 | 158 | 1,58 |
| Vibrio | 11 | 4 | 3 | 1 | 3 | 1 | 14 | 1 | 6 | 0 | 0 | 0 | 0 | 44 | 0,44 |
| Total | 1 318 | 1 359 | 467 | 471 | 3 030 | 1 501 | 536 | 396 | 100 | 798 | 20 | 4 | 0 | 10 000 | 100,00 |
Table 2 Notes
|
|||||||||||||||
Les taux d'incidence nationaux annuels des groupes d'entéropathogènes déclarés au PNSME entre 2015 et 2020 sont illustrés au tableau 3 et à l'annexe A. Les isolats d'E. coli O157, d'ECST autres que O157, de Listeria monocytogenes, de Salmonella et de Shigella sont couramment transmis aux laboratoires provinciaux de santé publique, tandis que les isolats de Campylobacter, de Yersinia, de parasites entériques (Giardia, Cryptosporidium, Entamoeba histolytica/dispar et Cyclospora) et de virus entériques (norovirus, rotavirus et adénovirus) ne sont pas déclarés de façon régulière aux laboratoires provinciaux de santé publique ou au laboratoire de référence central. Ainsi, on considère que les taux d'incidence signalés au PNSME reflètent les véritables taux d'incidence de ces agents pathogènes couramment déclarés, permettant ainsi de calculer les taux d'incidence provinciaux et territoriaux, tel qu'il est illustré au tableau 4.
| Groupe | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Total | Tauxtableau 3 note 1 | Total | Tauxtableau 3 note 1 | Total | Tauxtableau 3 note 1 | Total | Tauxtableau 3 note 1 | Total | Tauxtableau 3 note 1 | Total | Tauxtableau 3 note 1 | |
| E. coli O157 | 379 | 1,06 | 415 | 1,15 | 348 | 0,95 | 426 | 1,15 | 397 | 1,06 | 237 | 0,62 |
| ECST autres que O157tableau 3 note 2 | 229 | 0,64 | 205 | 0,57 | 361 | 0,99 | 525 | 1,42 | 595 | 1,58 | 320 | 0,84 |
| Listeria | 125 | 0,35 | 191 | 0,53 | 109 | 0,30 | 150 | 0,40 | 174 | 0,46 | 158 | 0,42 |
| Salmonella | 7 717 | 21,61 | 7 816 | 21,65 | 7 313 | 20,01 | 7 300 | 19,70 | 6 350 | 16,89 | 4 919 | 12,94 |
| Shigella | 739 | 2,07 | 807 | 2,23 | 699 | 1,91 | 784 | 2,12 | 828 | 2,20 | 393 | 1,03 |
| Campylobacter | 1 514 | 4,24 | 1 378 | 3,82 | 1 287 | 3,52 | 1 333 | 3,60 | 1 664 | 4,43 | 1 289 | 3,39 |
| Vibrio | 85 | 0,24 | 44 | 0,12 | 54 | 0,15 | 67 | 0,18 | 52 | 0,14 | 44 | 0,12 |
| Yersinia | 383 | 1,07 | 353 | 0,98 | 387 | 1,06 | 404 | 1,09 | 318 | 0,85 | 283 | 0,74 |
| Parasites | 1 845 | 5,17 | 1 921 | 5,32 | 1 679 | 4,59 | 1 675 | 4,52 | 1 639 | 4,36 | 1 017 | 2,68 |
| Virus | 3 075 | 8,61 | 2 295 | 6,36 | 2 600 | 7,11 | 2 303 | 6,21 | 2 656 | 7,07 | 1 067 | 2,81 |
Table 3 Notes
|
||||||||||||
| Groupetableau 4 note 2 | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.-É. | T.-N.-L. | Yn | T.-N.-O. | Nt |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| E. coli O157 | 0,72 | 1,00 | 0,34 | 1,60 | 0,58 | 0,51 | 0,13 | 0 | 0 | 0 | 0 | 0 | 0 |
| ECST autres que O157 | 0,85 | 3,55 | 3,22 | 1,31 | 0,26 | 0,26 | 0 | 0,10 | 0,63 | 0 | 0 | 0 | 0 |
| Listeria | 0,23 | 0,20 | 0,51 | 0,51 | 0,45 | 0,51 | 0,77 | 0,51 | 0,63 | 0,38 | 0 | 0 | 0 |
| Salmonella | 14,76 | 16,76 | 12,56 | 13,05 | 11,07 | 11,29 | 15,61 | 14,91 | 6,89 | 40,03 | 4,76 | 2,21 | 0 |
| Shigella | 1,09 | 0,84 | 0,59 | 0,73 | 1,06 | 1,41 | 0 | 0,20 | 0 | 0,57 | 0 | 2,21 | 0 |
Table 4 Notes
|
|||||||||||||
Salmonella
Au total, 4 919 isolats de Salmonella représentant 196 sérotypes ont été déclarés en 2020. Salmonella Enteritidis représentait 29 % de tous les cas de salmonellose humaine et, une fois combiné aux huit autres sérotypes les plus fréquents (figure 1), ils représentaient 73 % de tous les cas d'infection de Salmonella déclarés. Les nombres de cas nationaux, provinciaux et territoriaux causés par Salmonella qui ont été déclarés en 2020 sont indiqués au tableau 5 et à l'annexe B.
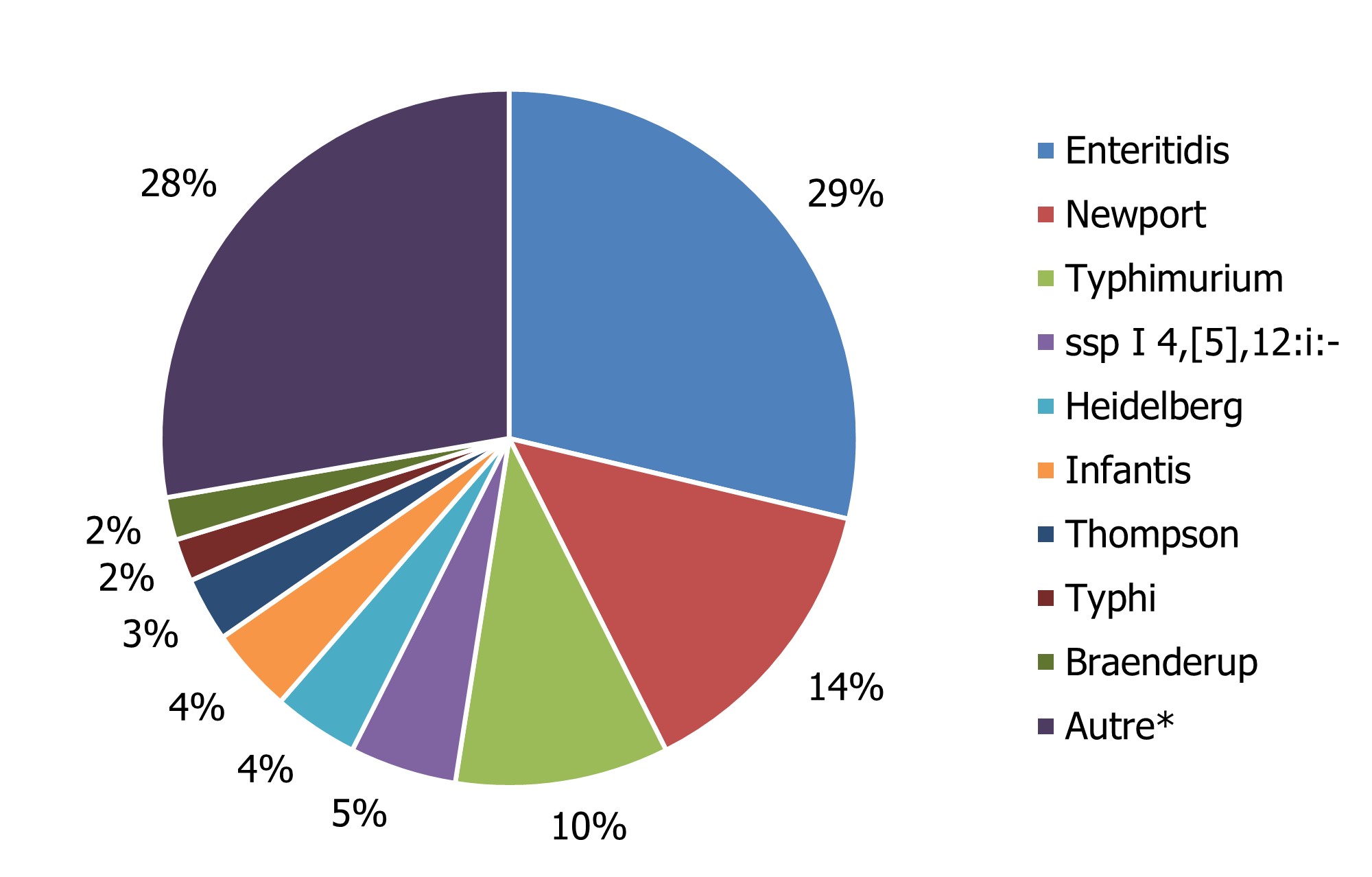
Figure 1 - Équivalent textuel
| Sérotypes de Salmonella | Proportion de Salmonelles totales |
|---|---|
| Enteritidis | 29 % |
| Newport | 14 % |
| Typhimurium | 10 % |
| ssp I 4,[5],12:i:- | 5 % |
| Heidelberg | 4 % |
| Infantis | 4 % |
| Thompson | 3 % |
| Typhi | 2 % |
| Braenderup | 2 % |
| Autrefigure 1 note * | 28 % |
Remarques sur la figure 1
|
|
*Les autres isolats (1 355 isolats) ont été répartis entre 187 sérotypes ou profils antigéniques incomplets, et 58 isolats ont été déclarés en tant qu'espèce de Salmonella indéterminée.
| Groupe | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.-É. | T.-N.-L. | Yn | T.-N.-O. | Nt | Total | % du total d'isolats de Salmonella (n=4 919) |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Enteritidis | 303 | 138 | 43 | 42 | 357 | 232 | 70 | 104 | 5 | 127 | 1 | 0 | 0 | 1 422 | 28,91 |
| Newport | 153 | 322 | 38 | 34 | 91 | 50 | 0 | 3 | 1 | 1 | 0 | 0 | 0 | 693 | 14,09 |
| Typhimurium | 49 | 55 | 10 | 18 | 208 | 116 | 2 | 3 | 3 | 4 | 0 | 0 | 0 | 468 | 9,51 |
| ssp I 4,[5],12:i:- | 14 | 24 | 4 | 2 | 76 | 113 | 11 | 8 | 0 | 4 | 0 | 0 | 0 | 256 | 5,20 |
| Heidelberg | 9 | 13 | 2 | 5 | 113 | 50 | 6 | 3 | 0 | 6 | 0 | 0 | 0 | 207 | 4,21 |
| Infantis | 11 | 18 | 4 | 10 | 72 | 67 | 6 | 5 | 1 | 4 | 0 | 0 | 0 | 198 | 4,03 |
| Thompson | 7 | 2 | 0 | 2 | 69 | 40 | 2 | 4 | 0 | 0 | 0 | 0 | 0 | 126 | 2,56 |
| Typhi | 20 | 11 | 7 | 5 | 59 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 113 | 2,30 |
| Braenderup | 11 | 13 | 2 | 4 | 35 | 11 | 3 | 1 | 0 | 0 | 0 | 1 | 0 | 81 | 1,65 |
| Montevideo | 6 | 3 | 0 | 1 | 8 | 52 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 71 | 1,44 |
| Total | 583 | 599 | 110 | 123 | 1 088 | 742 | 101 | 131 | 10 | 146 | 1 | 1 | 0 | 3 635 | 73,90 |
Comparé au nombre moyen de déclarations pour Salmonella reçues entre 2015 et 2019 (7 299 cas), une diminution de 32,6 % est observée en 2020 (4 919 cas), laquelle s'explique probablement et en partie par les effets de la pandémie de COVID-19 et les répercussions durables de la réglementation mise en œuvre par l'ACIA, en avril 2019, pour réduire la présence de Salmonella dans les produits surgelés de poulet cru pané (figure 2). Bien que S. Enteritidis soit demeuré le sérotype le plus fréquent au cours de cette période, on a observé des variations chez les autres sérotypes de Salmonella les plus fréquents (tableau 6).
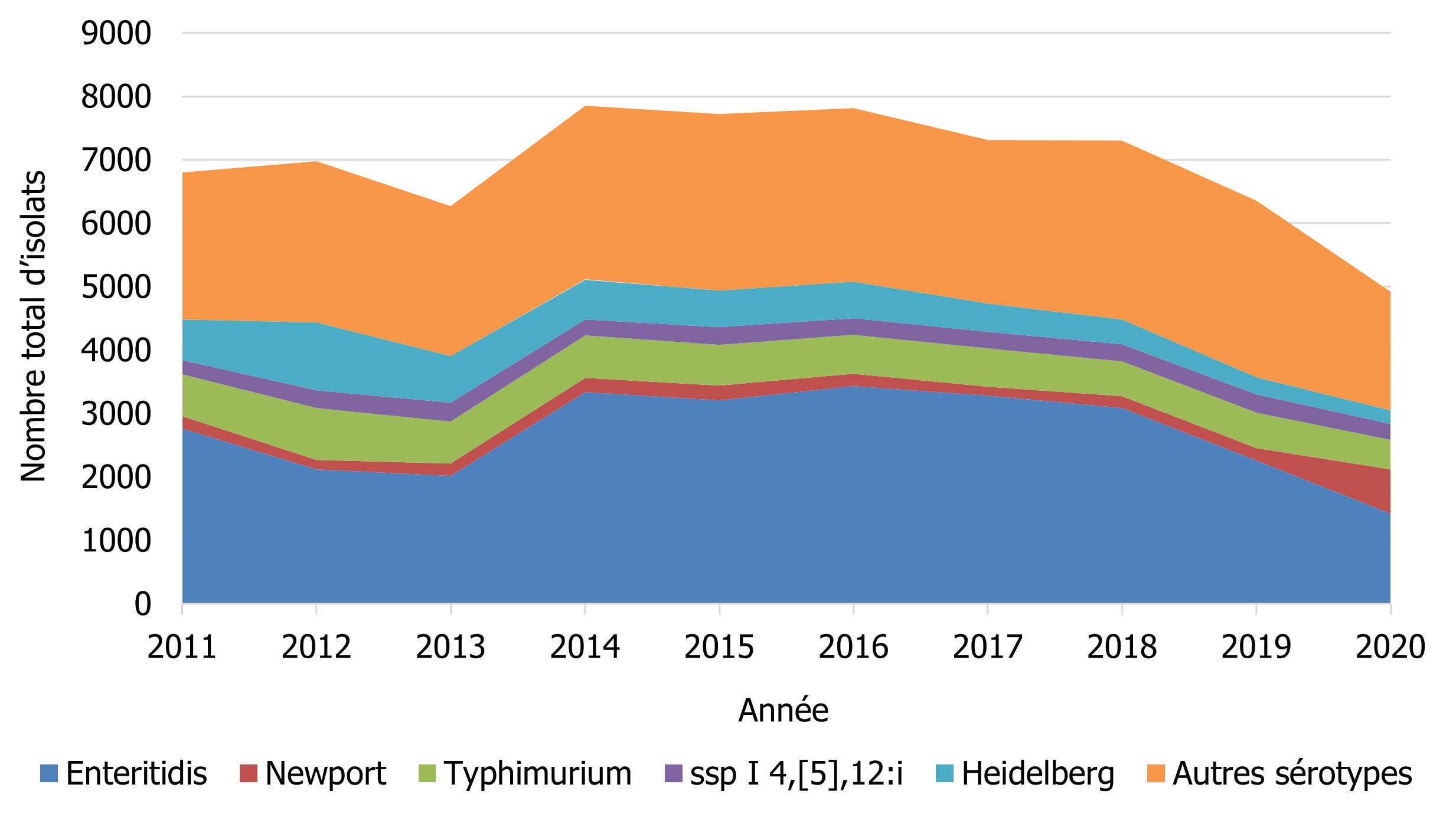
Figure 2 - Équivalent textuel
| Sérotypes de Salmonella | Année | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 2011 | 2012 | 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| Enteritidis | 2763 | 2117 | 2019 | 3337 | 3209 | 3433 | 3278 | 3083 | 2254 | 1422 |
| Newport | 195 | 153 | 189 | 224 | 235 | 198 | 143 | 192 | 200 | 693 |
| Typhimurium | 661 | 814 | 668 | 671 | 642 | 607 | 602 | 551 | 557 | 468 |
| ssp I 4,[5],12:i | 219 | 281 | 299 | 251 | 280 | 259 | 265 | 263 | 294 | 256 |
| Heidelberg | 641 | 1071 | 733 | 628 | 571 | 580 | 444 | 390 | 267 | 207 |
| Autres sérotypes | 2325 | 2544 | 2362 | 2739 | 2780 | 2739 | 2581 | 2821 | 2778 | 1873 |
| Sérotypes | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | Nbre moyen d'isolats (2015 - 2019) |
|---|---|---|---|---|---|---|---|
| Enteritidis | 3 209 | 3 433 | 3 278 | 3 083 | 2 254 | 1 422 | 3 051 |
| Newport | 235 | 198 | 143 | 192 | 200 | 693 | 194 |
| Typhimurium | 642 | 607 | 602 | 551 | 557 | 468 | 592 |
| ssp I 4,[5],12:i:- | 280 | 259 | 265 | 263 | 294 | 256 | 272 |
| Heidelberg | 571 | 580 | 444 | 390 | 267 | 207 | 450 |
| Infantis | 279 | 378 | 244 | 313 | 264 | 198 | 296 |
| Thompson | 311 | 290 | 135 | 148 | 98 | 126 | 196 |
| Typhi | 121 | 136 | 181 | 198 | 232 | 113 | 174 |
| Braenderup | 123 | 81 | 145 | 127 | 102 | 81 | 116 |
| Montevideo | 26 | 30 | 32 | 45 | 36 | 71 | 34 |
| Oranienburg | 68 | 61 | 53 | 113 | 104 | 71 | 80 |
| Paratyphi A | 77 | 62 | 62 | 56 | 116 | 59 | 75 |
| Javiana | 136 | 114 | 111 | 118 | 143 | 50 | 124 |
| Agona | 63 | 120 | 103 | 125 | 101 | 35 | 102 |
En mai 2017, PulseNet Canada s'est mis à effectuer le séquençage génomique pour tous les isolats de Salmonella faisant l'objet d'une surveillance régulière en laboratoire, fournissant ainsi des données très précises sur les sous-types de génomes qui permettent de détecter les agrégats et d'intervenir en conséquence.
Salmonella Enteritidis
En 2020, on a signalé au PNSME 1 422 isolats de S. Enteritidis au Canada, ce qui représentait 28,9 % de tous les isolats de Salmonella soumis. Le taux d'incidence observé en 2020 était 51,9 % moins élevé (3,7 cas pour 100 000 personnes) qu'au cours de la période de référence de 2011 à 2015 (7,7 cas pour 100 000 personnes). On observe également une diminution générale de l'incidence entre 2016 et 2019, ce qui semble indiquer que le taux observé en 2020 suit une tendance continue accentué par sans lien avec les effets de la pandémie de COVID-19 (figure 3).
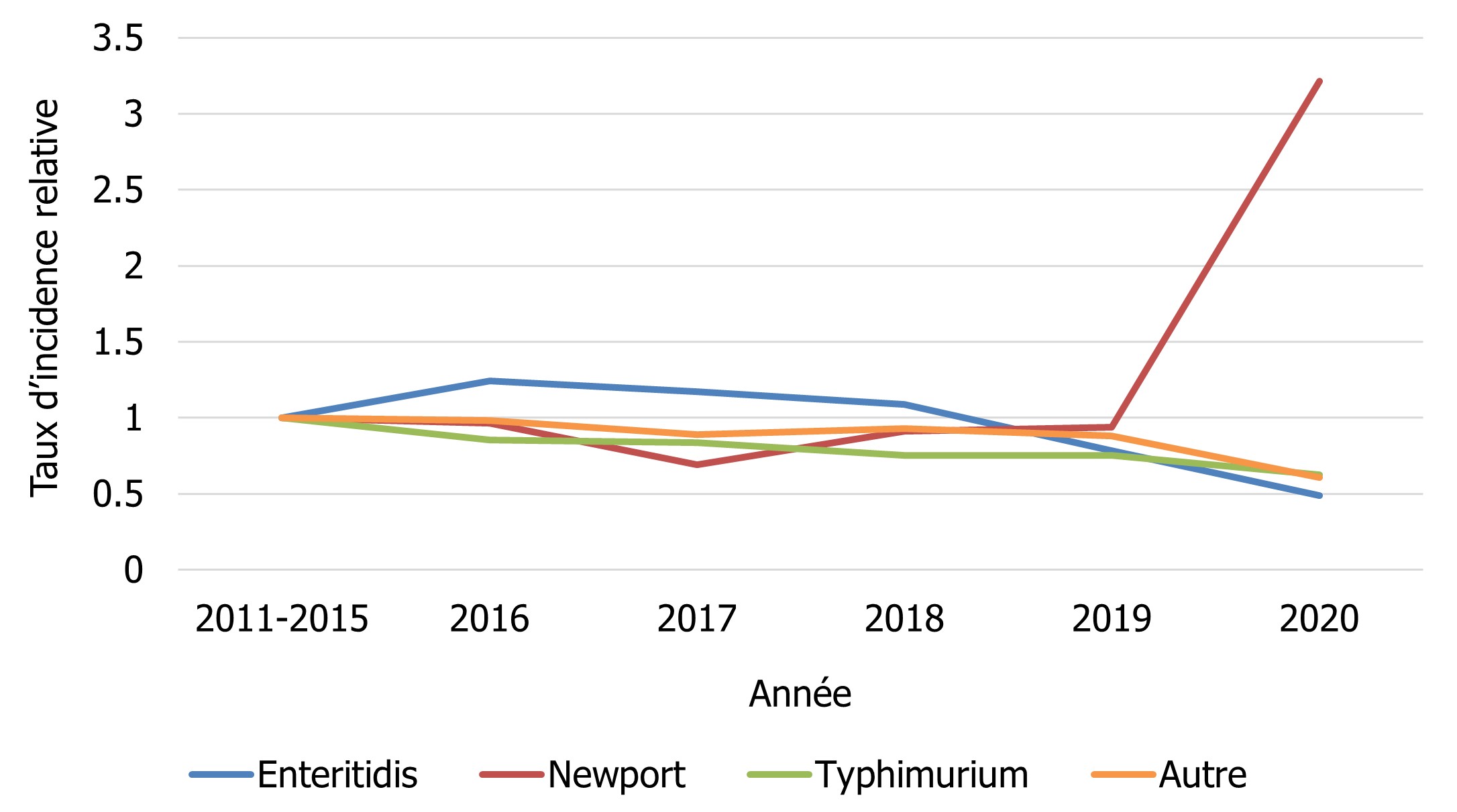
Figure 3 - Équivalent textuel
| Sérotypes de Salmonella | Année | |||||
|---|---|---|---|---|---|---|
| 2011-2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| Enteritidis | 1 | 1,24 | 1,17 | 1,09 | 0,78 | 0,49 |
| Newport | 1 | 0,97 | 0,69 | 0,91 | 0,94 | 3,21 |
| Typhimurium | 1 | 0,85 | 0,83 | 0,75 | 0,75 | 0,62 |
| Autre | 1 | 0,98 | 0,89 | 0,93 | 0,88 | 0,61 |
- Figure 3 Remarque *
-
Les taux sont comparés à ceux de la période de référence de 2011 à 2015.
Salmonella Newport
Le taux d'incidence de S. Newport en 2020 (1,82 cas pour 100 000 personnes) était beaucoup plus élevé qu'en 2019 (0,53 cas pour 100 000 personnes) et qu'au cours de la période de référence (0,57 cas pour 100 000 personnes) en raison d'une éclosion multijuridictionnelle de 515 cas, survenue entre le 15 juin et le 29 août 2020 (tableau 1).
Salmonella Typhimurium
Comparativement à la période de référence de 2011 à 2015, on a observé une baisse de 26 % de l'incidence de S. Typhimurium en 2020 (1,97 cas comparativement à 1,46 cas pour 100 000 personnes, respectivement). De 2015 à 2019, on a constaté une légère tendance à la diminution de l'incidence de S. Typhimurium (figure 3). Bien que S. Typhimurium demeure l'un des trois principaux sérotypes responsables de la salmonellose humaine au Canada, il ne représente que 10 % de tous les isolats de Salmonella signalés au PNSME en 2020 (figure 1 et tableau 6).
Escherichia coli
Sauf indication contraire, on suppose que tous les échantillons signalés au PNSME par les provinces et les territoires sont des échantillons d'Escherichia coli producteur de shigatoxines (ECST). En 2020, le taux du sérotype O157 (0,62 cas pour 100 000 personnes) est inférieur aux taux relativement stables observés entre 2010 et 2019, probablement en raison des répercussions de la pandémie de COVID-19 (figure 4). En 2020, trois provinces ont rapporté des taux d'incidence d'E. coli O157 supérieurs au taux d'incidence national déclaré, soit l'Alberta (1,00 cas pour 100 000 personnes), le Manitoba (1,60 cas pour 100 000 personnes) et la Colombie-Britannique (0,72 cas pour 100 000 personnes) [tableau 4]. Le taux d'incidence d'ECST autres que O157 a diminué en 2020 (0,84 cas pour 100 000 personnes) par rapport au taux déclaré en 2019 (1,58 cas pour 100 000 personnes), probablement en raison des répercussions de la pandémie (figure 4). Il s'agit de la quatrième année consécutive où la proportion d'isolats d'ECST autres que O157 a dépassé la proportion d'isolats d'E. coli O157. Il convient de faire remarquer que les sérotypes d'ECST autres que O157 ne sont pas signalés aussi systématiquement au PNSME qu'E. coli O157 et que, par conséquent, tout changement observé au fil du temps reflète les pratiques d'analyse de certains laboratoires provinciaux de santé publique. En outre, 6,6 % des isolats avaient été identifiés à l'aide de test de diagnostic sans culture (TDSC) qui sont des analyses basées sur une amplification en chaîne par polymérase et servent à identifier des organismes sans isolat cultivé. La culture réflexe d'un échantillon positif confirmé par TDSC pourrait générer un isolat pour un sous-typage additionnel, ce qui se traduirait par une mise à jour dans le PNSME.
En 2020, parmi les isolats d'ECST autres que O157 soumis à des tests de sérotypage, 54 % d'entre eux étaient représentés par cinq sérotypes : E. coli O26, E. coli O111, E. coli O121, E. coli O103 et E. coli O118 (figure 5). En 2020, on ignorait le sérotype de 20 % d'ECST autres que O157. En 2017, le LNM a demandé aux laboratoires provinciaux de santé publique de déclarer la méthode d'analyse servant à identifier les organismes, étant donné que le recours aux TDSC devient de plus en plus répandu au Canada.
Par rapport au taux déclaré en 2019, une diminution du taux d'incidence pour 100 000 personnes a été observée, en 2020, pour les cinq principaux sérotypes d'E. coli, à l'exception d'E. coli O121, probablement en raison des répercussions de la pandémie de COVID-19 (figure 6). L'annexe B dresse la liste de tous les sérotypes d'E. coli, y compris d'ECST autres que O157, ayant été confirmés, et tout autre pathotype signalé.
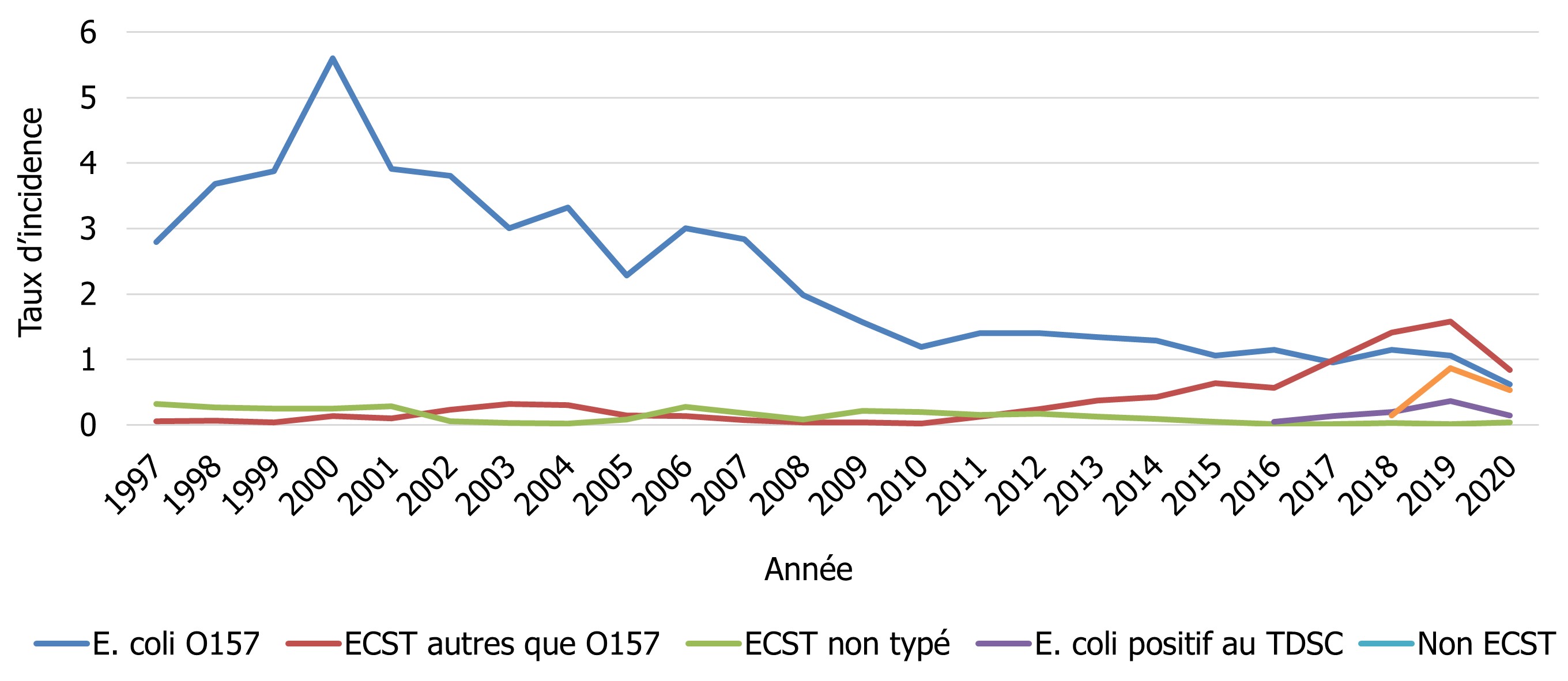
Figure 4 - Équivalent textuel
| Groupe de E. coli | Année | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1997 | 1998 | 1999 | 2000 | 2001 | 2002 | 2003 | 2004 | 2005 | 2006 | 2007 | 2008 | |
| E. coli O157 | 2,79 | 3,68 | 3,88 | 5,60 | 3,91 | 3,81 | 3,01 | 3,32 | 2,28 | 3,00 | 2,84 | 1,99 |
| ECST autres que O157 | 0,06 | 0,07 | 0,04 | 0,13 | 0,10 | 0,23 | 0,32 | 0,30 | 0,14 | 0,14 | 0,08 | 0,04 |
| ECST non typé | 0,32 | 0,27 | 0,25 | 0,25 | 0,28 | 0,05 | 0,03 | 0,03 | 0,08 | 0,28 | 0,18 | 0,09 |
| E. coli positif au TDSC | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Non ECST | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Groupe de E. coli | Année | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2009 | 2010 | 2011 | 2012 | 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| E. coli O157 | 1,57 | 1,19 | 1,40 | 1,40 | 1,35 | 1,29 | 1,06 | 1,15 | 0,95 | 1,15 | 1,06 | 0,62 |
| ECST autres que O157 | 0,04 | 0,02 | 0,13 | 0,24 | 0,37 | 0,43 | 0,64 | 0,57 | 0,99 | 1,42 | 1,58 | 0,84 |
| ECST non typé | 0,21 | 0,19 | 0,15 | 0,18 | 0,13 | 0,10 | 0,05 | 0,01 | 0,01 | 0,04 | 0,01 | 0,04 |
| E. coli positif au TDSC | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0,05 | 0,14 | 0,19 | 0,37 | 0,14 |
| Non ECST | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0,15 | 0,87 | 0,53 |
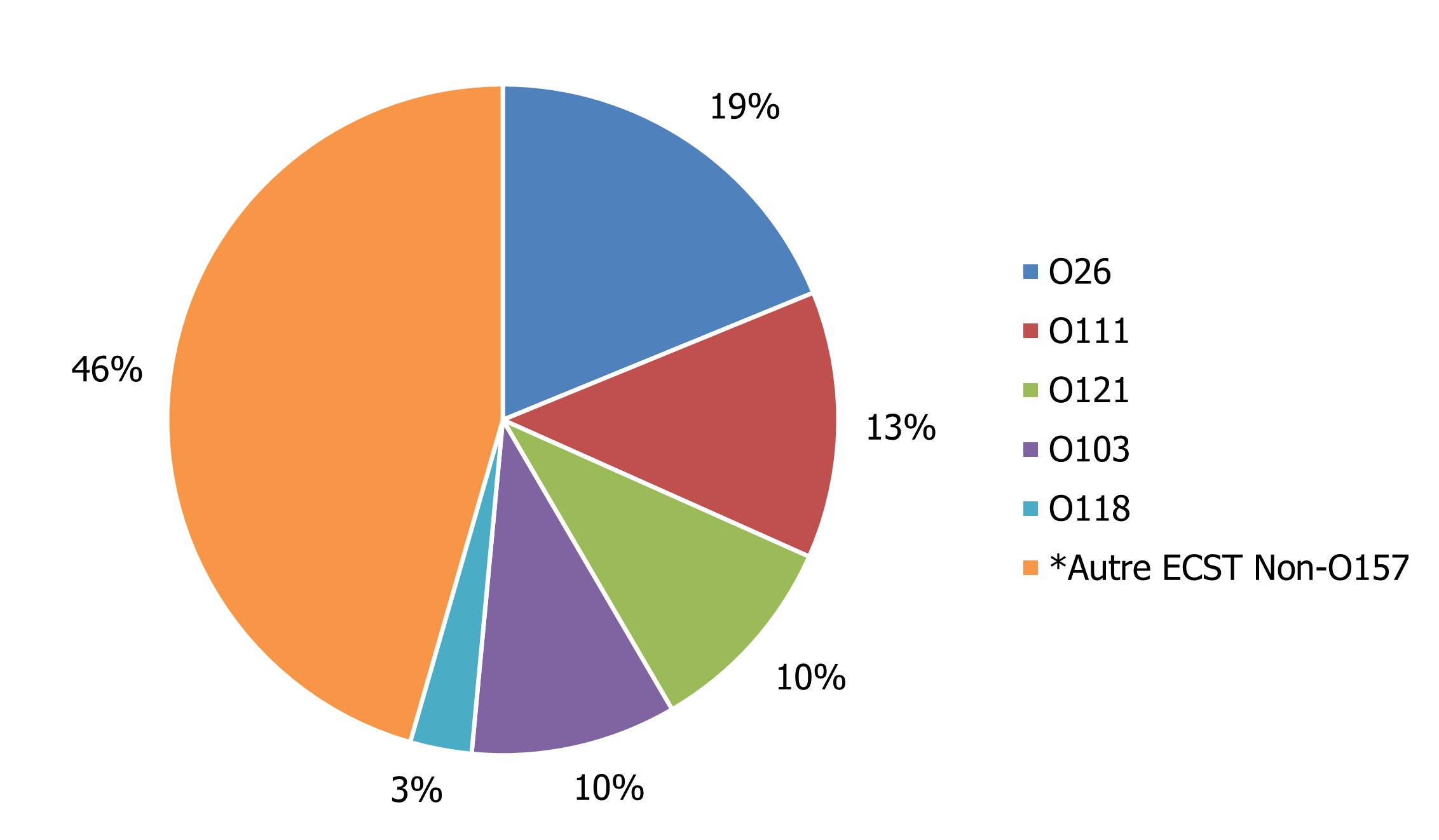
Figure 5 - Équivalent textuel
| Sérotypes d'ECST autres que O157 | Total pourcentage d'ECST autres que O157 |
|---|---|
| O26 | 19 % |
| O111 | 13 % |
| O121 | 10 % |
| O103 | 10 % |
| O118 | 3 % |
| Autre ECST Non-O157figure 5 note * | 46 % |
Remarques sur la figure 5
|
|
*Les autres sérotypes (146 isolats) ont été répartis entre 36 sérotypes, et 63 isolats ont été déclarés en tant qu’ECST non O157 indéterminés.
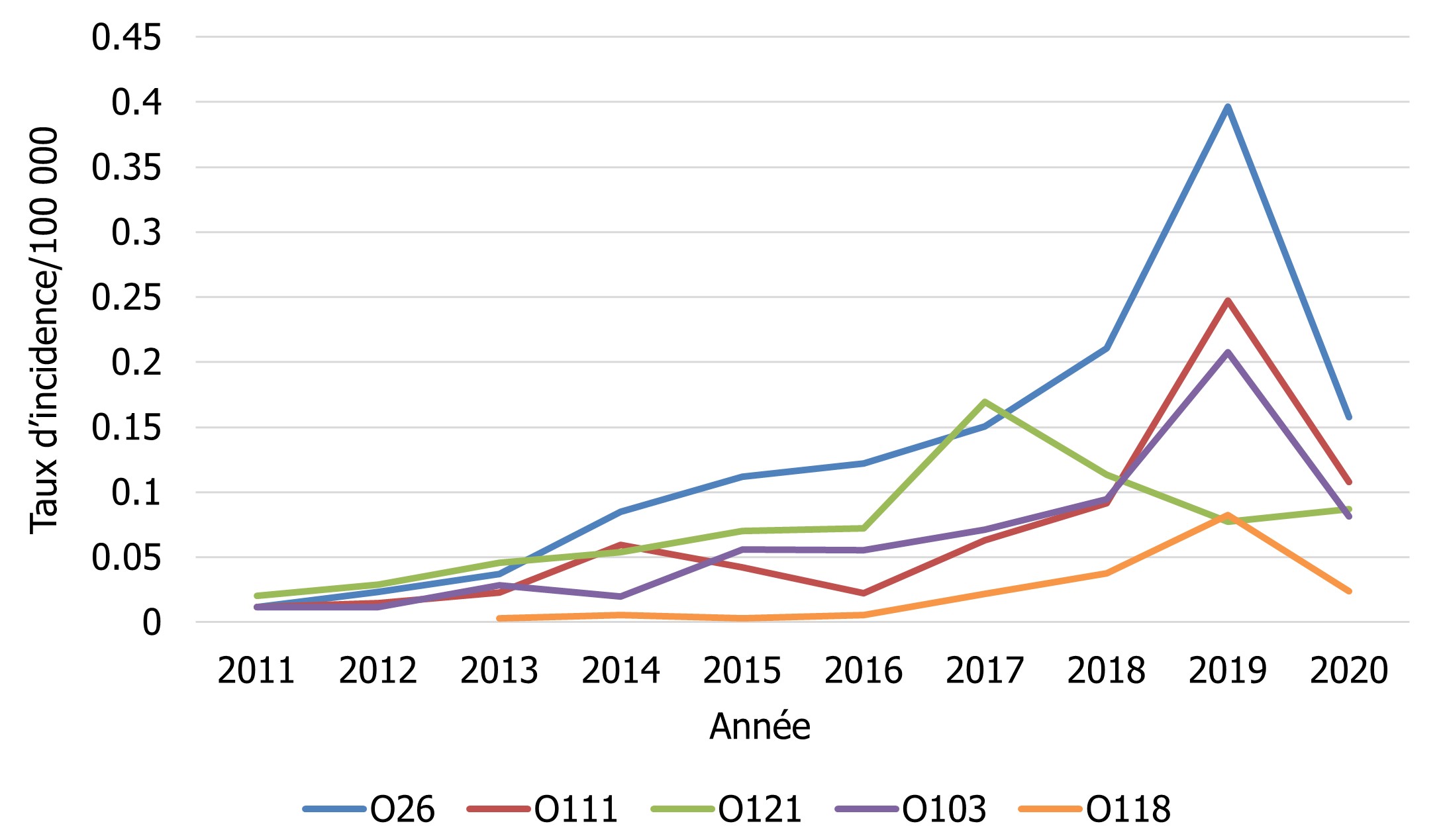
Figure 6 - Équivalent textuel
| Sérotypes d'ECST autres que O157 | Année | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 2011 | 2012 | 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| O26 | 0,01 | 0,02 | 0,04 | 0,08 | 0,11 | 0,12 | 0,15 | 0,21 | 0,40 | 0,16 |
| O111 | 0,01 | 0,01 | 0,02 | 0,06 | 0,04 | 0,02 | 0,06 | 0,09 | 0,25 | 0,11 |
| O121 | 0,02 | 0,03 | 0,05 | 0,05 | 0,07 | 0,07 | 0,17 | 0,11 | 0,08 | 0,09 |
| O103 | 0,01 | 0,01 | 0,03 | 0,02 | 0,06 | 0,06 | 0,07 | 0,09 | 0,21 | 0,08 |
| O118 | 0 | 0 | 0 | 0,01 | 0 | 0,01 | 0,02 | 0,04 | 0,08 | 0,02 |
Listeria monocytogenes
Conformément à la définition de cas de la listériose invasive, seuls les isolats prélevés d'un site normalement stérile ou de tissus placentaires ou fœtaux devraient être déclarés. En 2020, on a déclaré un nombre moins important d'isolats responsables de la listériose invasive (158) comparativement à 2019 (174). Puisque le nombre de cas de listériose invasive est faible dans la plupart des provinces et des territoires, l'ampleur du changement en est largement affectée, même avec une différence d'un seul cas (figure 7).
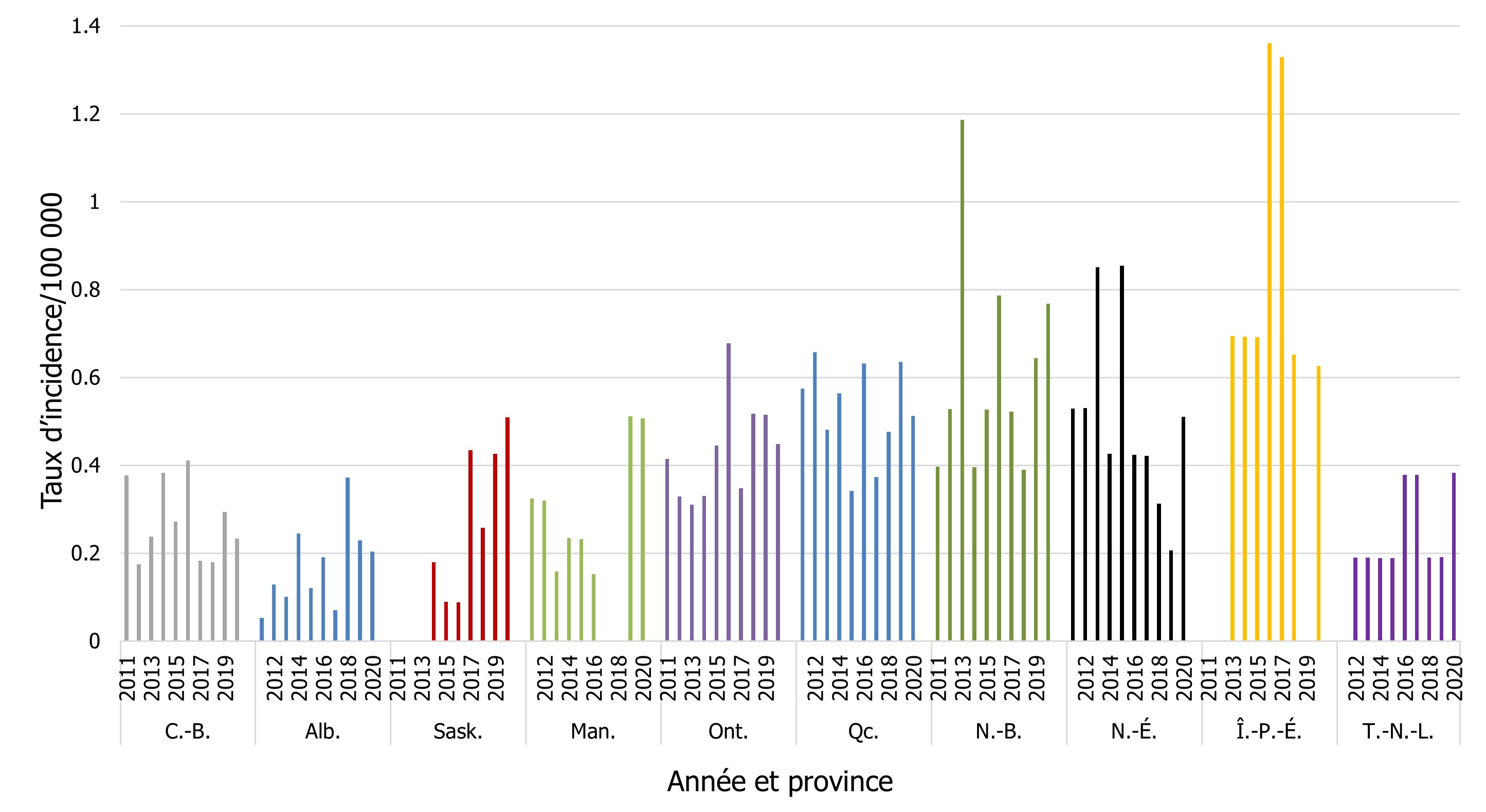
Figure 7 - Équivalent textuel
| Province | Année | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 2011 | 2012 | 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| C.-B. | 0,38 | 0,18 | 0,24 | 0,38 | 0,27 | 0,41 | 0,18 | 0,18 | 0,29 | 0,23 |
| Alb. | 0,05 | 0,13 | 0,10 | 0,24 | 0,12 | 0,19 | 0,07 | 0,37 | 0,23 | 0,20 |
| Sask. | 0 | 0 | 0 | 0,18 | 0,09 | 0,09 | 0,43 | 0,26 | 0,43 | 0,51 |
| Man. | 0,32 | 0,32 | 0,16 | 0,23 | 0,23 | 0,15 | 0 | 0 | 0,51 | 0,51 |
| Ont. | 0,41 | 0,33 | 0,31 | 0,33 | 0,45 | 0,68 | 0,35 | 0,52 | 0,52 | 0,45 |
| Qc. | 0,57 | 0,66 | 0,48 | 0,56 | 0,34 | 0,63 | 0,37 | 0,48 | 0,64 | 0,51 |
| N.-B. | 0,40 | 0,53 | 1,19 | 0,40 | 0,53 | 0,79 | 0,52 | 0,39 | 0,64 | 0,77 |
| N.-É. | 0,53 | 0,53 | 0,85 | 0,43 | 0,85 | 0,42 | 0,42 | 0,31 | 0,21 | 0,51 |
| Î.-P.- É. | 0 | 0 | 0,69 | 0,69 | 0,69 | 1,36 | 1,33 | 0,65 | 0 | 0,63 |
| T.-N.-L. | 0 | 0,19 | 0,19 | 0,19 | 0,19 | 0,38 | 0,38 | 0,19 | 0,19 | 0,38 |
- Figure 1 Remarque *
-
Aucun cas de listériose invasive n'a été déclaré en 2020 par le Yukon, les Territoires du Nord-Ouest et le Nunavut.
Shigella
Un total de 393 isolats de Shigella a été déclaré en 2020, ce qui représente un taux d'incidence de 1,03 cas pour 100 000 personnes comparativement à une moyenne de 2,11 cas pour 100 000 personnes entre 2015 et 2019 (figure 8). Les isolats de Shigella sonnei et de S. flexneri représentaient respectivement 38 % et 57 % de toutes les déclarations. Les tendances globales pour Shigella ont, depuis longtemps, été attribuables à l'incidence de S. sonnei (0,40 cas pour 100 000 personnes). Toutefois, le taux de S. flexneri (0,59 cas pour 100 000 personnes) a dépassé celui de S. sonnei en 2020 (figure 8). Parmi les autres espèces de Shigella, les tendances de l'incidence au fil du temps se sont relativement maintenues, les taux d'incidence de Shigella boydii et de Shigella dysenteriae étant respectivement de 0,03 cas et de 0,01 cas pour 100 000 personnes en 2020 (figure 8). Les taux d'incidence de toutes les espèces de Shigella indiquées dans la figure 8 étaient plus faibles en 2020 qu'en 2019, ce qui est probablement attribuable aux effets de la pandémie de COVID-19.
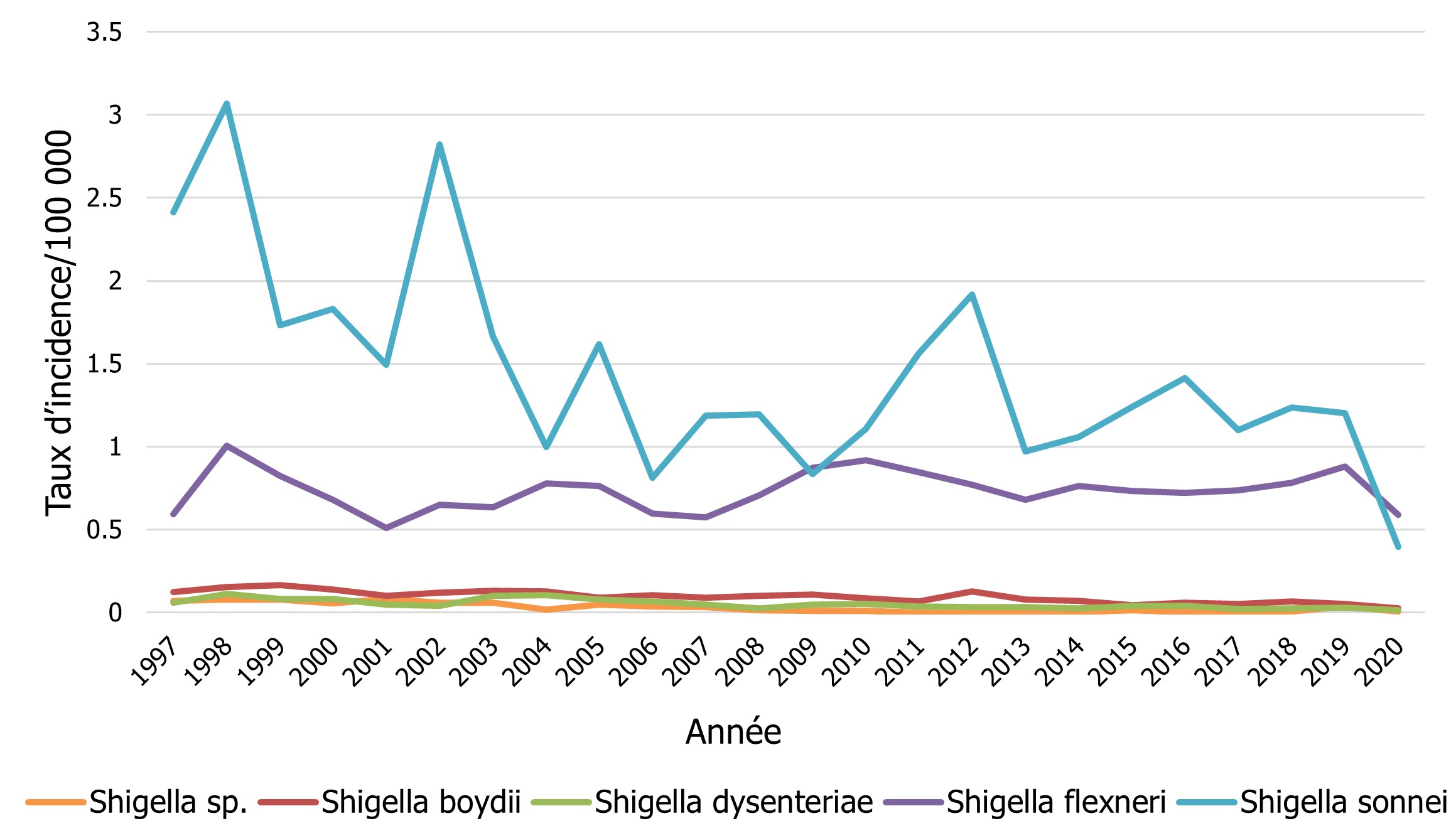
Figure 8 - Équivalent textuel
| Groupe de Shigella | Année | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1997 | 1998 | 1999 | 2000 | 2001 | 2002 | 2003 | 2004 | 2005 | 2006 | 2007 | 2008 | |
| Shigella sp. | 0,07 | 0,08 | 0,08 | 0,06 | 0,08 | 0,06 | 0,06 | 0,02 | 0,05 | 0,04 | 0,03 | 0,02 |
| Shigella boydii | 0,12 | 0,15 | 0,16 | 0,14 | 0,10 | 0,12 | 0,13 | 0,13 | 0,09 | 0,10 | 0,09 | 0,10 |
| Shigella dysenteriae | 0,06 | 0,11 | 0,08 | 0,08 | 0,05 | 0,04 | 0,10 | 0,11 | 0,08 | 0,07 | 0,05 | 0,03 |
| Shigella flexneri | 0,59 | 1,00 | 0,82 | 0,68 | 0,51 | 0,65 | 0,64 | 0,78 | 0,76 | 0,60 | 0,57 | 0,71 |
| Shigella sonnei | 2,41 | 3,07 | 1,73 | 1,83 | 1,49 | 2,82 | 1,66 | 1,00 | 1,62 | 0,81 | 1,19 | 1,19 |
| Groupe de Shigella | Année | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2009 | 2010 | 2011 | 2012 | 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| Shigella sp. | 0,01 | 0,01 | 0 | 0 | 0,01 | 0 | 0,01 | 0 | 0 | 0,01 | 0,04 | 0,01 |
| Shigella boydii | 0,11 | 0,09 | 0,07 | 0,13 | 0,08 | 0,07 | 0,04 | 0,06 | 0,05 | 0,07 | 0,05 | 0,03 |
| Shigella dysenteriae | 0,05 | 0,05 | 0,03 | 0,03 | 0,03 | 0,03 | 0,04 | 0,04 | 0,02 | 0,02 | 0,03 | 0,01 |
| Shigella flexneri | 0,87 | 0,92 | 0,84 | 0,77 | 0,68 | 0,76 | 0,73 | 0,72 | 0,74 | 0,78 | 0,88 | 0,59 |
| Shigella sonnei | 0,84 | 1,11 | 1,56 | 1,92 | 0,97 | 1,06 | 1,24 | 1,42 | 1,10 | 1,24 | 1,20 | 0,40 |
Hépatite A
Le taux d'incidence national de l'hépatite A en 2020 était inférieur à celui de 2019 (0,67 cas pour 100 000 personnes en 2020 comparativement à 1,55 cas pour 100 000 personnes en 2019), probablement en raison des conséquences de la pandémie de COVID-19 (figure 9). Chaque laboratoire provincial ou territorial détermine s'il faut signaler un cas en se basant uniquement sur les analyses de laboratoire, sans qu'un suivi de la santé publique soit effectué. Un IgM positif pourrait être imputable à un faux positif ou à une immunisation récente. Au moment du suivi effectué par la santé publique locale, on détermine si le cas satisfait à la définition de cas confirmé ou non. Si la santé publique locale détermine qu'il ne s'agit pas d'un cas (p. ex., immunisation récente), il est possible qu'elle ne communique pas toujours avec le laboratoire, ce qui fait que nous ne pouvons corriger nos chiffres de surveillance. Les augmentations illustrées à la figure 9 pourraient être attribuables à un changement des méthodes de détection en laboratoire. Inversement, étant donné que tous les spécimens ou isolats ne sont pas renvoyés par les laboratoires régionaux et locaux aux laboratoires de santé publique provinciaux, les virus, dont l'hépatite A,sont sous représentés dans le cadre du PNSME et les dénombrements des cas déclarés n'illustrent pas l'incidence réelle de la maladie au Canada.
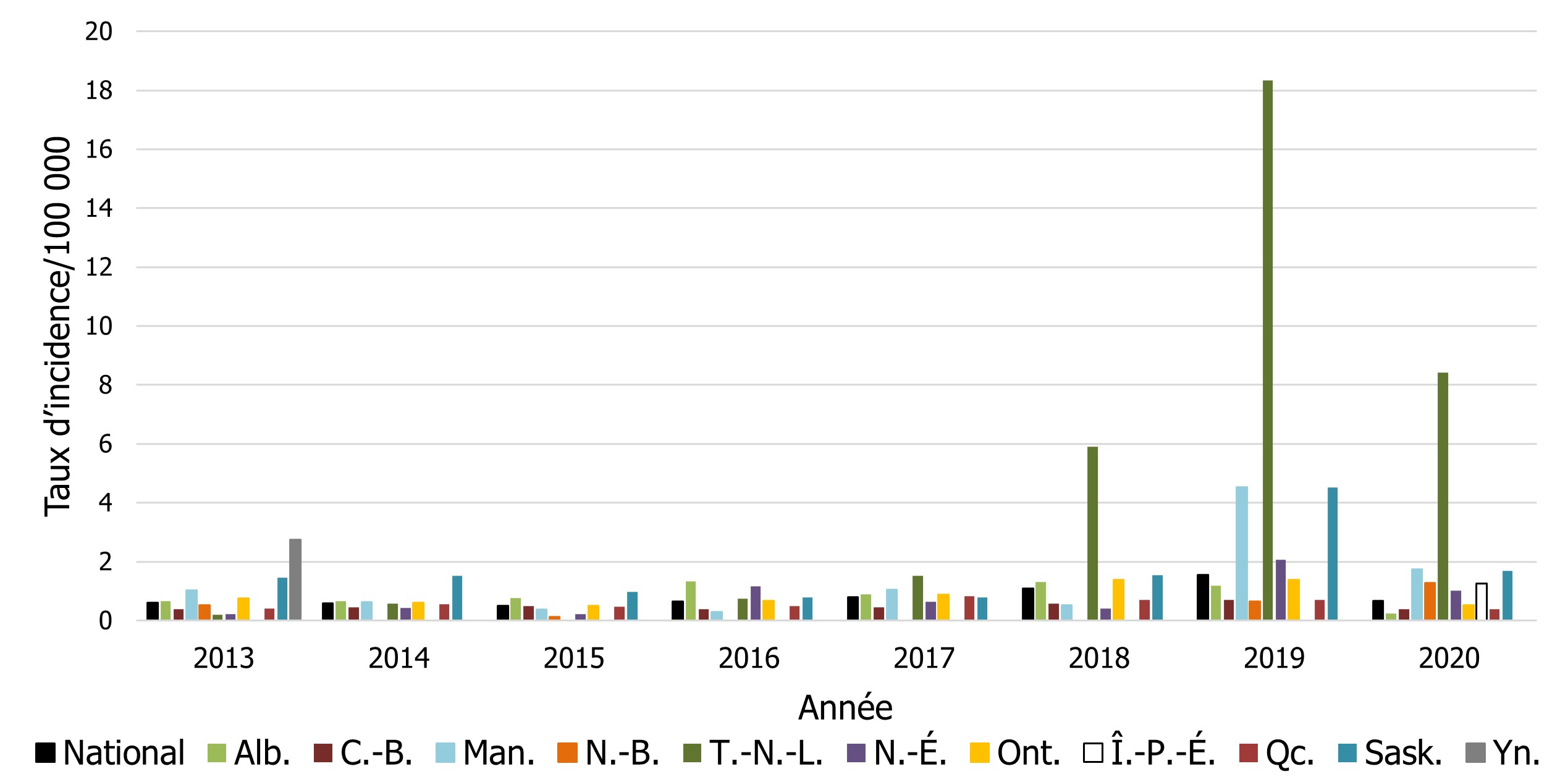
Figure 9 - Équivalent textuel
| Province / territoire | Année | |||||||
|---|---|---|---|---|---|---|---|---|
| 2013 | 2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | |
| National | 0,62 | 0,59 | 0,51 | 0,65 | 0,79 | 1,08 | 1,55 | 0,67 |
| Alb. | 0,65 | 0,66 | 0,77 | 1,33 | 0,90 | 1,30 | 1,19 | 0,32 |
| C.-B. | 0,39 | 0,45 | 0,50 | 0,39 | 0,45 | 0,58 | 0,71 | 0,39 |
| Man. | 1,03 | 0,63 | 0,39 | 0,30 | 1,05 | 0,52 | 4,53 | 1,74 |
| N.-B. | 0,53 | 0 | 0,13 | 0 | 0 | 0 | 0,64 | 1,28 |
| T.-N.-L. | 0,19 | 0,57 | 0 | 0,76 | 1,51 | 5,90 | 18,34 | 8,43 |
| N.-É. | 0,21 | 0,43 | 0,21 | 1,17 | 0,63 | 0,42 | 2,06 | 1,02 |
| Ont. | 0,75 | 0,61 | 0,50 | 0,67 | 0,88 | 1,38 | 1,38 | 0,53 |
| Î.-P.- É. | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1,25 |
| Qc. | 0,42 | 0,56 | 0,46 | 0,49 | 0,83 | 0,70 | 0,69 | 0,40 |
| Sask. | 1,45 | 1,53 | 0,98 | 0,79 | 0,78 | 1,55 | 4,52 | 1,70 |
| Yn. | 2,74 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
Annexe A. Comparaison des nombres totaux, des taux d’incidence (pour 100 000 personnes) et des pourcentages de cas de maladies entériques enregistrés au Canada, en 2019, par le Système de surveillance des maladies à déclaration obligatoire du Canada (SSMDOC) et le PNSMEannexe un tableau 1 note 1
| Maladies entériques et d’origine hydrique et alimentaire | Système de surveillance des maladies à déclaration obligatoire du Canada (SSMDOC) | Programme national de surveillance des maladies entériques (PNSME) | % des cas du SSMDOC saisis dans le PNSME (isolements signalés au PNSME/cas du SSMDOCannexe A tableau 1 note 8) | ||
|---|---|---|---|---|---|
| 2019 | N | Taux pour 100 000 personnes |
N | Taux pour 100 000 personnes |
|
| Botulisme | 7 | 0,02 | - | - | N/A |
| Campylobactérioseannexe A tableau 1 note 2 | 10 237 | 27,23 | 1 664 | - | 16,3 |
| Choléraannexe A tableau 1 note 3 | 3 | 0,01 | 2 | 0,005 | 66,7 |
| Cryptosporidioseannexe A tableau 1 note 2 | 1 490 | 3,96 | 457 | - | 30,7 |
| Cyclosporoseannexe A tableau 1 note 2 | 495 | 1,32 | 66 | - | 13,3 |
| Giardiaseannexe A tableau 1 note 2 | 3 889 | 10,34 | 739 | - | 19,0 |
| Hépatite A | 386 | 1,03 | 583 | 1,55 | 151,0annexe A tableau 1 note 8 |
| Listériose invasive | 172 | 0,43 | 174 | 0,46 | 101,2annexe A tableau 1 note 8 |
| Norovirusannexe A tableau 1 note 2annexe A tableau 1 note 4annexe A tableau 1 note 5 | 372 | 7,48 | 1 554 | - | N/A |
| Intoxication par phycotoxine paralysanteannexe A tableau 1 note 6 | 3 | 0,009 | - | - | N/A |
| Salmonellose | 5 845 | 15,55 | 6 350 | 16,9 | 108,6annexe A tableau 1 note 8 |
| Shigellose | 914 | 2,43 | 827 | 2,02 | 90,5 |
| Typhoïdeannexe A tableau 1 note 7 | 194 | 0,52 | 232 | 0,62 | 119,6annexe A tableau 1 note 8 |
| Infection aux E. coli producteurs de shigatoxines | 1 116 | 2,97 | 776annexe A tableau 1 note 9 | 2,06 | 69,5 |
Table 8 Notes
|
|||||
Annexe B. Données sur les espèces et les sérotypes déclarés au PNSME par province et territoire, 2020
Les cas visitant une autre province ou territoire sont entrés dans le décompte total de la province où le cas a été détecté.
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Campylobacter coli | 1 | 9 | 11 | 7 | 7 | 19 | 10 | 1 | 4 | 0 | 0 | 0 | 0 | 69 |
| Campylobacter concisus | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Campylobacter curvus | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 3 |
| Campylobacter fetus | 1 | 0 | 0 | 0 | 6 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 |
| Campylobacter gracilis | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Campylobacter hominis | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Campylobacter hyointestinalis | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Campylobacter jejuni | 15 | 240 | 157 | 40 | 37 | 159 | 170 | 85 | 43 | 0 | 4 | 1 | 0 | 951 |
| Campylobacter lari | 0 | 0 | 0 | 0 | 3 | 2 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 8 |
| Campylobacter rectus | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 2 |
| Campylobacter showae - like | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Campylobacter sp | 2 | 0 | 0 | 0 | 0 | 0 | 66 | 0 | 0 | 105 | 0 | 0 | 0 | 173 |
| Campylobacter upsaliensis | 1 | 2 | 1 | 1 | 32 | 8 | 3 | 2 | 1 | 0 | 0 | 0 | 0 | 51 |
| Campylobacter ureolyticus | 0 | 1 | 0 | 0 | 0 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Campylobacter volucris | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Total Campylobacter | 25 | 252 | 169 | 48 | 87 | 208 | 252 | 90 | 48 | 105 | 4 | 1 | 0 | 1 289 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| E. coli | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ECST/positifs au STX confirmés par TDSC | 6 | 0 | 0 | 0 | 0 | 40 | 0 | 0 | 0 | 9 | 0 | 0 | 0 | 55 |
| E. coli autres que O157 (ECST) | 5 | 0 | 38 | 18 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 63 |
| E. coli entéroagrégatif (ECEA) non typé | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 23 | 0 | 0 | 0 | 23 |
| E. coli entéropathogène (ECEP) non typé | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 167 | 0 | 0 | 0 | 167 |
| E. coli entérotoxinogène (ECET) non typé | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 13 | 0 | 0 | 0 | 13 |
| E. coli non typé (ECST) | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 14 |
| E. coli O indéterminé:H11 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O indéterminé:H16 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O indéterminé:H19 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O indéterminé:H2 | 0 | 1tableau 8 note * | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O indéterminé:H21 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O indéterminé:H45 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O indéterminé:H7 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O indéterminé:H8 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O indéterminé:HNM | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| E. coli O-rugueux:H12 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O-rugueux:H19 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O-rugueux:H2 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O100:HNM | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O103:H2 | 4 | 12tableau 8 note * | 0 | 0 | 6 | 4tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 26 |
| E. coli O103:H25 | 0 | 2tableau 8 note * | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| E. coli O103:HNM | 1 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O111 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| E. coli O111:HNM | 2 | 22tableau 8 note * | 0 | 0 | 9 | 5tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 38 |
| E. coli O113:H21 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O117:H7 | 4 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| E. coli O118:14 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O118:H16 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 2tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| E. coli O118:H2 | 0 | 3tableau 8 note * | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| E. coli O118:HNM | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O121 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O121:H19 | 9 | 23tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 32 |
| E. coli O123:H2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O128ab:H2 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O142:H rugueux | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O145 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O145:H25 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O145:HNM | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O146:H21 | 1 | 2tableau 8 note * | 0 | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| E. coli O152:H8 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O156:H25 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O157 | 0 | 0 | 4tableau 8 note * | 21tableau 8 note * | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 26 |
| E. coli O157:H7 | 32 | 41 | 0 | 1 | 75 | 40 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 189 |
| E. coli O157:HNM | 5 | 3tableau 8 note * | 0 | 0 | 10 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 22 |
| E. coli O168:H8 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O16:H5 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O181:H49 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O186:H11 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O186:H2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O25:H15 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O26:H11 | 5 | 33tableau 8 note * | 0 | 0 | 9 | 2tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 49 |
| E. coli O26:HNM | 0 | 8tableau 8 note * | 0 | 0 | 1 | 2tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 11 |
| E. coli O28ac:H25 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O38:H21 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O41:H2 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O45:H2 | 0 | 0 | 0 | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O48:H21 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O52:H45 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| E. coli O5:HNM | 0 | 5tableau 8 note * | 0 | 0 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 6 |
| E. coli O60:H7 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O69:H11 | 0 | 5tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| E. coli O69:HNM | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O70:HNM | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O71:H11 | 0 | 4tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| E. coli O71:H8 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O71:HNM | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O76:H19 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O82:H40 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O84:HNM | 0 | 4tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| E. coli O85:H2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O86:H2 | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O87:H16 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O91:H21 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| E. coli O91:HNM | 0 | 1tableau 8 note * | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Total E. coli | 97 | 201 | 42 | 40 | 124 | 107 | 1 | 1 | 1 | 216 | 0 | 0 | 0 | 830 |
Remarques du tableau 8
|
||||||||||||||
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Listeria monocytogenes | 12 | 9 | 6 | 7 | 66 | 44 | 6 | 5 | 1 | 2 | 0 | 0 | 0 | 158 |
| Total Listeria | 12 | 9 | 6 | 7 | 66 | 44 | 6 | 5 | 1 | 2 | 0 | 0 | 0 | 158 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Salmonella Aarhus | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Adelaide | 0 | 0 | 0 | 0 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Adjame | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Agama | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Agbeni | 1 | 0 | 2 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 6 |
| Salmonella Agona | 7 | 6 | 0 | 0 | 14 | 7 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 35 |
| Salmonella Alachua | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Albany | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Altona | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Amounderness | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Anatum | 2 | 1 | 0 | 0 | 15 | 7 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 26 |
| Salmonella Apapa | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Apeyeme | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Arechavaleta | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Austin | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Bareilly | 2 | 2 | 0 | 1 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 12 |
| Salmonella Berta | 0 | 0 | 0 | 0 | 4 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Blockley | 0 | 0 | 0 | 0 | 2 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Salmonella Bonariensis | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Bovismorbificans | 1 | 1 | 0 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Braenderup | 11 | 13 | 2 | 4 | 35 | 11 | 3 | 1 | 0 | 0 | 0 | 1 | 0 | 81 |
| Salmonella Brancaster | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Brandenburg | 4 | 3 | 3 | 0 | 5 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 23 |
| Salmonella Breda | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Bredeney | 0 | 0 | 1 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Brunei | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Cerro | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Chandans | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Chester | 2 | 0 | 0 | 0 | 4 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Salmonella Chicago | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Choleraesuis | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Choleraesuis var Decatur | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Choleraesuis var Kunzendorf | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Coeln | 1 | 3 | 0 | 0 | 3 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 11 |
| Salmonella Colindale | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Corvallis | 3 | 0 | 0 | 1 | 8 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 14 |
| Salmonella Cotham | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Cubana | 2 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Daytona | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Derby | 1 | 1 | 0 | 2 | 5 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 13 |
| Salmonella Dublin | 2 | 2 | 0 | 0 | 6 | 17 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 27 |
| Salmonella Durban | 0 | 1 | 0 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Ealing | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Eastbourne | 0 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Emek | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Enteritidis | 303 | 138 | 43 | 42 | 357 | 232 | 70 | 104 | 5 | 127 | 1 | 0 | 0 | 1 422 |
| Salmonella Essen | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Fluntern | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Freetown | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Gaminara | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Georgia | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Give | 0 | 2 | 1 | 3 | 8 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 |
| Salmonella Glostrup | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Grumpensis | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Hadar | 5 | 4 | 1 | 1 | 41 | 5 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 59 |
| Salmonella Haifa | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Hartford | 0 | 0 | 0 | 0 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 6 |
| Salmonella Havana | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Heidelberg | 9 | 13 | 2 | 5 | 113 | 50 | 6 | 3 | 0 | 6 | 0 | 0 | 0 | 207 |
| Salmonella Hvittingfoss | 0 | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Idikan | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Indiana | 2 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Infantis | 11 | 18 | 4 | 10 | 72 | 67 | 6 | 5 | 1 | 4 | 0 | 0 | 0 | 198 |
| Salmonella Isangi | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Javiana | 3 | 7 | 1 | 3 | 16 | 16 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 50 |
| Salmonella Jedburgh | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Johannesburg | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Kedougou | 0 | 0 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Kentucky | 6 | 1 | 0 | 0 | 7 | 3 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 18 |
| Salmonella Kiambu | 0 | 2 | 4 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 10 |
| Salmonella Kisarawe | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Kisii | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Kottbus | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Lattenkamp | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Lexington | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Litchfield | 1 | 1 | 0 | 1 | 13 | 6 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 24 |
| Salmonella Liverpool | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Livingstone | 0 | 0 | 0 | 0 | 5 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 6 |
| Salmonella Lomalinda | 0 | 0 | 0 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Lome | 0 | 0 | 0 | 0 | 6 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 9 |
| Salmonella London | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Salmonella Manhattan | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Matopeni | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Mbandaka | 3 | 1 | 2 | 0 | 3 | 1 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 12 |
| Salmonella Miami | 0 | 0 | 0 | 0 | 10 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 13 |
| Salmonella Michigan | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Mikawasima | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Minnesota | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Mississippi | 1 | 0 | 0 | 0 | 5 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 10 |
| Salmonella Molade | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Montevideo | 6 | 3 | 0 | 1 | 8 | 52 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 71 |
| Salmonella Muenchen | 6 | 7 | 1 | 4 | 25 | 10 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 55 |
| Salmonella Muenster | 4 | 4 | 1 | 1 | 10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 22 |
| Salmonella Nchanga | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Newport | 153 | 322 | 38 | 34 | 91 | 50 | 0 | 3 | 1 | 1 | 0 | 0 | 0 | 693 |
| Salmonella Norwich | 0 | 0 | 0 | 0 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Oakland | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Ohio | 0 | 0 | 0 | 0 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Okatie | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Oranienburg | 9 | 9 | 2 | 0 | 32 | 16 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 71 |
| Salmonella Orientalis | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Oslo | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Ouakam | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Panama | 5 | 3 | 0 | 0 | 11 | 3 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 24 |
| Salmonella Paratyphi A | 15 | 10 | 0 | 0 | 30 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 59 |
| Salmonella Paratyphi B | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Paratyphi B var. Java | 16 | 9 | 0 | 0 | 8 | 6 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 43 |
| Salmonella Pensacola | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Plymouth | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Pomona | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Poona | 1 | 4 | 0 | 0 | 13 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 22 |
| Salmonella Praha | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Putten | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Reading | 15 | 8 | 8 | 13 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 50 |
| Salmonella Richmond | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Rissen | 2 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Rubislaw | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella Saintpaul | 9 | 6 | 2 | 3 | 14 | 4 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 42 |
| Salmonella Sandiego | 0 | 0 | 0 | 0 | 8 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 13 |
| Salmonella Schwarzengrund | 2 | 9 | 0 | 7 | 6 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 25 |
| Salmonella Senftenberg | 4 | 2 | 1 | 0 | 7 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 16 |
| Salmonella Singapore | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Soerenga | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Splott | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Stanley | 10 | 4 | 0 | 1 | 10 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 29 |
| Salmonella Stanleyville | 0 | 0 | 0 | 0 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Telelkebir | 0 | 1 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella Tennessee | 0 | 1 | 0 | 0 | 5 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Salmonella Thompson | 7 | 2 | 0 | 2 | 69 | 40 | 2 | 4 | 0 | 0 | 0 | 0 | 0 | 126 |
| Salmonella Typhi | 20 | 11 | 7 | 5 | 59 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 113 |
| Salmonella Typhimurium | 49 | 55 | 10 | 18 | 208 | 116 | 2 | 3 | 3 | 4 | 0 | 0 | 0 | 468 |
| Salmonella Uganda | 1 | 2 | 0 | 3 | 7 | 3 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 19 |
| Salmonella Veneziana | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Virchow | 1 | 4 | 0 | 1 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 10 |
| Salmonella Wandsworth | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Wangata | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella Waral | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Waycross | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella Weltevreden | 3 | 5 | 1 | 0 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 |
| Salmonella Worthington | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella bongori 48:z35:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella sp | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 56 | 0 | 0 | 0 | 58 |
| Salmonella ssp I | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp I 13,23:b:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 17:-:- | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 3,10:-:1,6 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 4,[5],12:-:- | 0 | 0 | 0 | 0 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella ssp I 4,[5],12:b:- | 0 | 0 | 0 | 0 | 17 | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 27 |
| Salmonella ssp I 4,[5],12:d:1,7 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 4,[5],12:i:- | 14 | 24 | 4 | 2 | 76 | 113 | 11 | 8 | 0 | 4 | 0 | 0 | 0 | 256 |
| Salmonella ssp I 47:z4,z23:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 48:-:-:z58 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 6,7,[14]:c:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 6,7:-:- | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella ssp I 6,7:c:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 6,7:z4,z23:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 6,8,[20]:-:z6 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 6,8:-:1,5 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 9,12:-:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I 9,12:-:1,5 | 0 | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella ssp I O Untypeable:HNM | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella ssp I O Untypeable:g,m:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:H r:- | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp I Rough-O:H undetermined | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:HNM | 0 | 0 | 0 | 0 | 6 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Salmonella ssp I Rough-O:a:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:a:1,7 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:g,m:- | 0 | 0 | 0 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Salmonella ssp I Rough-O:i:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:i:1,2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:m,t:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp I Rough-O:z29:- | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 |
| Salmonella ssp II 16:m,t:[z42] | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIa 11:z4,z23:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIa 41:z4,z23:- | 0 | 0 | 0 | 0 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Salmonella ssp IIIa 48:g,z51:- | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIa 56:z4,z23:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp IIIb 38:l,v:z35 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp IIIb 50:k:z | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp IIIb 50:r:z53 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 50:r:z:[z67] | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Salmonella ssp IIIb 50:z:z52 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 53:z10:z35 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 58:z52:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 61:k:1,5 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 61:l,v:1,5,7:[z57] | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 61:r:z53:[z47],[z50 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb 61:z52:z53 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb Rough-O:l,v:z35 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb Rough-O:r:z | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IIIb Rough-O:z10:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IV | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IV 44:z4,z23:- | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IV 45:g,z51:- | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IV 45:z36,z38:- | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Salmonella ssp IV 48:g,z51:- | 0 | 1 | 0 | 0 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 |
| Total Salmonella | 760 | 741 | 148 | 180 | 1 631 | 968 | 122 | 146 | 11 | 209 | 2 | 1 | 0 | 4 919 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Shigella | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 3 |
| Shigella boydii 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella boydii 12 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Shigella boydii 18 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella boydii 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Shigella boydii 20 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella boydii 4 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella boydii 9 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella boydii | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella dysenteriae 2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Shigella dysenteriae 4 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella dysenteriae 9 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Shigella flexneri 1a | 0 | 0 | 0 | 0 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 |
| Shigella flexneri 1b | 4 | 3 | 0 | 0 | 12 | 32 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 52 |
| Shigella flexneri 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella flexneri 2a | 10 | 9 | 0 | 0 | 23 | 37 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 79 |
| Shigella flexneri 2b | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella flexneri 3a | 4 | 2 | 0 | 0 | 15 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 28 |
| Shigella flexneri 3b | 0 | 0 | 0 | 0 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Shigella flexneri 4a | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Shigella flexneri 4b | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella flexneri 4c | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Shigella flexneri 5a | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Shigella flexneri 6 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Shigella flexneri | 1 | 0 | 3 | 5 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 10 |
| Shigella flexneri Prov. SH-104 | 0 | 0 | 0 | 0 | 19 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 26 |
| Shigella flexneri var. Y | 2 | 0 | 0 | 0 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 6 |
| Shigella sonnei | 29 | 20 | 4 | 5 | 67 | 25 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 151 |
| Total Shigella | 56 | 37 | 7 | 10 | 156 | 121 | 0 | 2 | 0 | 3 | 0 | 1 | 0 | 393 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Vibrio alginolyticus | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 |
| Vibrio cholerae | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Vibrio cholerae non-O1/O139 | 3 | 0 | 2 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 8 |
| Vibrio fluvialis | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 3 |
| Vibrio harveyi | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Vibrio parahaemolyticus | 6 | 1 | 1 | 0 | 2 | 0 | 11 | 0 | 6 | 0 | 0 | 0 | 0 | 27 |
| Total Vibrio | 11 | 4 | 3 | 1 | 3 | 1 | 14 | 1 | 6 | 0 | 0 | 0 | 0 | 44 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Yersinia enterocolitica | 52 | 22 | 9 | 3 | 114 | 18 | 2 | 3 | 0 | 2 | 2 | 0 | 0 | 227 |
| Yersinia frederiksenii | 16 | 3 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 20 |
| Yersinia intermedia | 6 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 9 |
| Yersinia kristensenii | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Yersinia massiliensis | 19 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 19 |
| Yersinia mollaretii | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Yersinia pseudotuberculosis | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Yersinia rohdei | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| Total Yersinia | 101 | 27 | 10 | 4 | 114 | 18 | 2 | 3 | 0 | 2 | 2 | 0 | 0 | 283 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Cryptosporidium | 7 | 2 | 7 | 11 | 160 | 0 | 30 | 26 | 10 | 24 | 1 | 0 | 0 | 278 |
| Cyclospora | 1 | 0 | 0 | 0 | 55 | 0 | 2 | 0 | 0 | 9 | 0 | 1 | 0 | 68 |
| Entamoeba histolytica/dispar | 89 | 1 | 2 | 8 | 75 | 0 | 0 | 4 | 0 | 0 | 4 | 0 | 0 | 183 |
| Giardia | 47 | 7 | 33 | 49 | 177 | 0 | 51 | 65 | 4 | 50 | 5 | 0 | 0 | 488 |
| Total Parasites | 144 | 10 | 42 | 68 | 467 | 0 | 83 | 95 | 14 | 83 | 10 | 1 | 0 | 1 017 |
| Organisme | C.-B. | Alb. | Sask. | Man. | Ont. | Qc. | N.-B. | N.-É. | Î.-P.- É. | T.-N.-L. | Yn. | T.-N.-O. | Nt. | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Adénovirus | 1 | 1 | 0 | 7 | 24 | 0 | 0 | 2 | 0 | 12 | 0 | 0 | 0 | 47 |
| Astrovirus | 2 | 9 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 26 | 0 | 0 | 0 | 40 |
| Entérovirus | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Hépatite A | 20 | 14 | 20 | 24 | 78 | 34 | 10 | 10 | 2 | 44 | 0 | 0 | 0 | 256 |
| Norovirus | 73 | 43 | 16 | 52 | 280 | 0 | 21 | 35 | 15 | 85 | 2 | 0 | 0 | 622 |
| Rotavirus | 12 | 11 | 4 | 11 | 0 | 0 | 25 | 4 | 2 | 3 | 0 | 0 | 0 | 72 |
| Sapovirus | 4 | 0 | 0 | 14 | 0 | 0 | 0 | 2 | 0 | 8 | 0 | 0 | 0 | 28 |
| Total Virus | 112 | 78 | 40 | 113 | 382 | 34 | 56 | 53 | 19 | 178 | 2 | 0 | 0 | 1 067 |
Annexe C : Effets de la pandémie de COVID-19 – Comparaison du nombre d’isolats hebdomadaires soumis au PNSME en 2020 et du nombre moyen d’isolats hebdomadaires soumis de 2015 à 2019 en ce qui concerne certains agents pathogènes
En mars 2020, la pandémie de COVID-19 a été déclaréeNote de bas de page 4 et des mesures de santé publique ont été prises à l’échelle mondiale pour y faire face. Dans l’ensemble du Canada et dans certains territoires, provinces et régions, diverses mesures de santé publique ont été mises en place. Ces mesures comprennent notamment l’interdiction de voyager à l’étrangerNote de bas de page 5 et dans le pays, la fermeture des entreprises et des activités non essentielles (restaurants, salles de sport, salons, lieux de culte, etc.), la fermeture des écoles et le lancement de l’apprentissage virtuel, l’obligation de se couvrir le visage dans les lieux publics et intérieurs. En outre, des messages de santé publique ont été diffusés plus fréquemment sur le lavage des mains, les règles d’hygiène en cas de toux et d’éternuements ainsi que des rappels sur la nécessité de rester à la maison en cas de malaise et de se soumettre à un test de dépistage de la COVID-19. Ces mesures de santé publique et les mesures d’adaptation mises en œuvre par les Canadiens dans le contexte de la pandémie de COVID-19 ont non seulement permis de réduire la transmission du virus responsable de la maladie, mais ont également eu, à différents degrés, des répercussions sur d’autres maladies infectieuses signalées. Les données du PNSME pour 2020 doivent être interprétées en tenant compte de ces mesures de santé publique et des mesures d’adaptation adoptées dans le contexte de la pandémie de COVID-19.
Les figures suivantes mettent en évidence les différences observées dans les données du PNSME pour 2020 en ce qui concerne certains agents pathogènes par rapport à celles des cinq années précédentes (2015 à 2019). Les données du PNSME sont représentées avec le nombre de cas nationaux de COVID-19 en tant qu'indicateur du stade de la pandémie et des mesures de santé publique connexes et ne doivent pas être interprétées comme ayant un lien direct avec ces cas.
Les mesures de santé publique et les mesures d'adaptation mises en œuvre dans le contexte de la pandémie de COVID-19 ont eu des effets différents sur les agents pathogènes. Pour toutes les espèces de Salmonella (figure 10), la diminution drastique, qui s'observe à compter de la semaine 13, est probablement associée à la mise en œuvre initiale des mesures de santé publique et aux restrictions concernant les voyages internationaux. Le pic enregistré à partir de la semaine 28 est lié à l'éclosion d'infections à S. Newport. Les figures 11 et 12 mettent en évidence les différences constatées entre S. Enteritidis seulement et tous les autres sérotypes de Salmonella, à l'exception de S. Enteritidis. Les données de 2020 montrent une diminution liée à S. Enteritidis, laquelle se maintient tout au long de l'année et s'explique probablement par les effets de la pandémie de COVID-19 et les répercussions durables de la réglementation mise en œuvre par l'ACIA, en avril 2019, pour éviter la présence de Salmonella dans les produits surgelés de poulet cru pané. Dans les figures 13 et 14, on observe une nette diminution initiale vers les semaines 14 et 15 en ce qui concerne E. coli O157 et ECST autres que O157, respectivement. Au cours des semaines 24 à 41, la fréquence d'E. coli O157 semble connaître une légère diminution en 2020 par rapport à la moyenne historique, puis, vers la semaine 41 et jusqu'à la fin de 2020, une diminution plus importante se poursuit. En revanche, l'ECST autres que O157 semble rebondir autour de la semaine 22 et se maintenir au niveau de la moyenne historique jusqu'à la semaine 39, où il semble ensuite connaître une légère baisse en 2020 par rapport à la moyenne historique. Cette légère différence entre les tendances d'E. coli O157 et d'ECST autres que O157 peut en partie être due à la tendance générale à la hausse de l'identification et de la déclaration des ECST autres que O157 qui a été observée. La figure 15 montre que la tendance relative à Listeria monocytogenes en 2020 correspond plutôt à la moyenne historique pour 2015 à 2019 bien que, en fait, elle soit légèrement supérieure à celle-ci. Cela démontre le peu d'effets des mesures de santé publique liées à la COVID-19 sur cet organisme. Listeria monocytogenes n'est généralement pas associée aux voyages et provoque des maladies graves, ce qui peut expliquer en partie la cohérence des données rapportées avec les niveaux avant la pandémie de COVID-19. En ce qui concerne Shigella, comme c'est le cas de toutes les espèces de Salmonella, dans la figure 16, la forte diminution notée vers la semaine 13 est suivie d'une diminution soutenue tout au long de 2020. Shigella est souvent associée aux voyages et transmise par contact de personne à personne, ce qui peut expliquer en partie l'effet marqué possible des mesures de santé publique liées à la COVID-19 sur cet organisme.
En général, on observe des diminutions plus importantes de la fréquence des agents pathogènes qui sont généralement associés aux voyages, qui causent habituellement des maladies plus bénignes ou qui sont plus souvent transmis par contact de personne à personne. Citons, à titre de comparaison, Listeria monocytogenes qui cause habituellement des maladies graves, n'est pas associée aux voyages et ne se transmet pas par contact de personne à personne. Une analyse supplémentaire sera effectuée pour examiner plus en profondeur les effets de la pandémie de COVID-19 sur les données du PNSME.
Les mesures de santé publique qui ont été mises en œuvre en réponse à la pandémie de COVID-19 étaient multiples; il est donc difficile d'attribuer des mesures spécifiques à des impacts spécifiques des maladies entériques (à l'exception peut-être des impacts des restrictions de voyage). Le fait que les mesures de santé publique aient également entraîné un changement majeur dans les habitudes de consommation alimentaire (c.-à-d. une diminution des aliments achetés et consommés en dehors du foyer) complique encore notre capacité à discerner facilement ces impacts individuels. Enfin, tout changement dans les comportements de recherche de soins médicaux chez les Canadiens à la suite des ordonnances de maintien à domicile et des changements dans les systèmes de soins de santé locaux peut également avoir influencé ces observations; toutefois, cela reste inconnu à l'heure actuelle. Une analyse supplémentaire sera effectuée pour examiner plus en profondeur les effets de la pandémie de COVID-19 sur la fréquence des maladies entériques au Canada ainsi que les données recueillies et rapportées du Programme national de surveillance des maladies entériques.
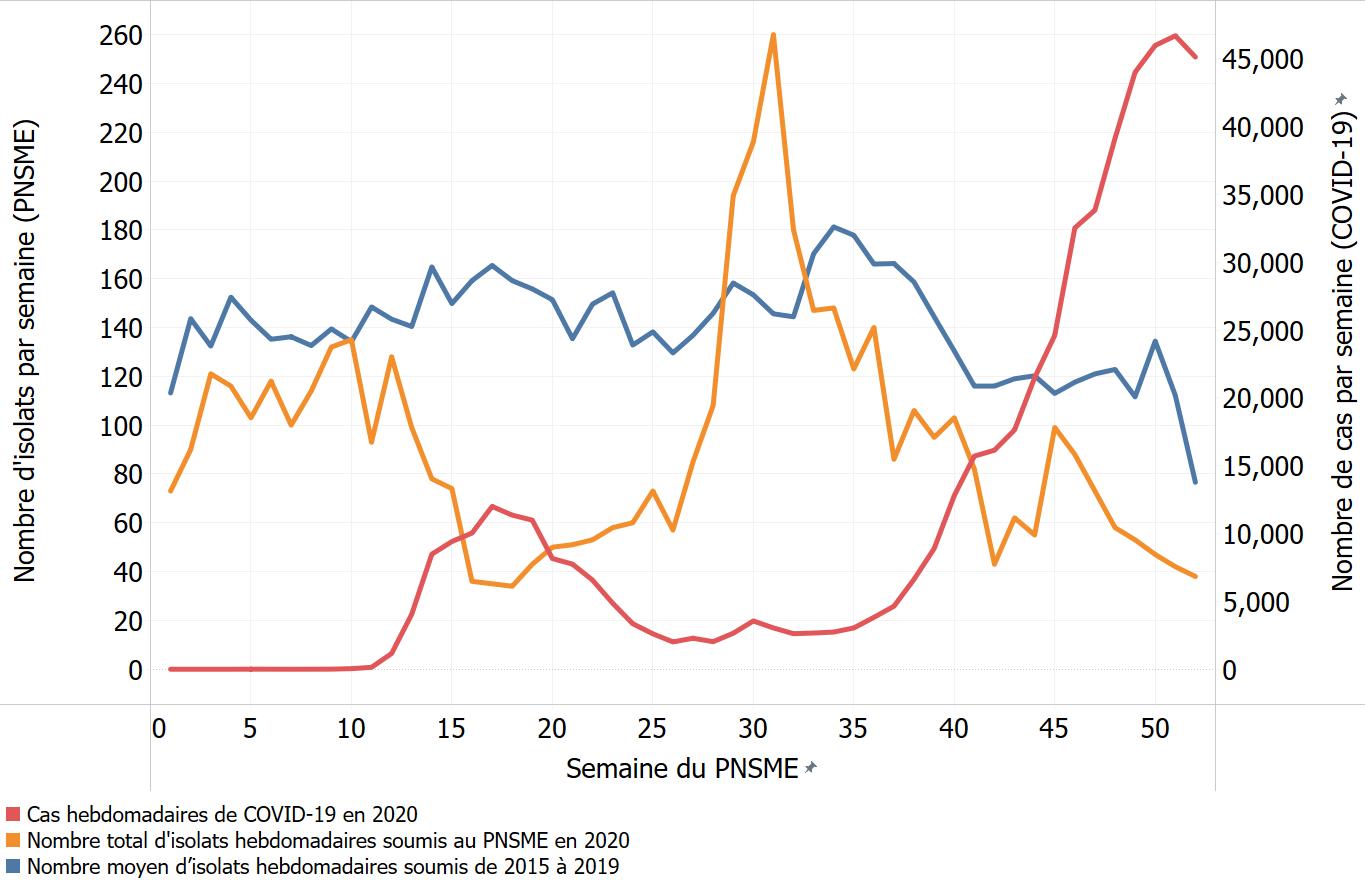
Figure 10 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 73 | 113,2 |
| 2 | 0 | 90 | 143,6 |
| 3 | 0 | 121 | 132,4 |
| 4 | 0 | 116 | 152,4 |
| 5 | 4 | 103 | 143 |
| 6 | 3 | 118 | 135,2 |
| 7 | 0 | 100 | 136,2 |
| 8 | 2 | 114 | 132,6 |
| 9 | 6 | 132 | 139,4 |
| 10 | 42 | 135 | 134,2 |
| 11 | 140 | 93 | 148,4 |
| 12 | 1161 | 128 | 143,4 |
| 13 | 4054 | 99 | 140,4 |
| 14 | 8479 | 78 | 164,8 |
| 15 | 9414 | 74 | 149,8 |
| 16 | 10035 | 36 | 159,2 |
| 17 | 11997 | 35 | 165,4 |
| 18 | 11359 | 34 | 159,2 |
| 19 | 10988 | 43 | 155,8 |
| 20 | 8159 | 50 | 151,4 |
| 21 | 7748 | 51 | 135,4 |
| 22 | 6564 | 53 | 149,6 |
| 23 | 4867 | 58 | 154,2 |
| 24 | 3353 | 60 | 132,8 |
| 25 | 2609 | 73 | 138,2 |
| 26 | 2013 | 57 | 129,6 |
| 27 | 2284 | 85 | 136,8 |
| 28 | 2030 | 108 | 145,8 |
| 29 | 2653 | 194 | 158,2 |
| 30 | 3557 | 216 | 153,4 |
| 31 | 3043 | 260 | 145,6 |
| 32 | 2622 | 180 | 144,4 |
| 33 | 2668 | 147 | 170,2 |
| 34 | 2740 | 148 | 181,2 |
| 35 | 3043 | 123 | 177,8 |
| 36 | 3822 | 140 | 166 |
| 37 | 4646 | 86 | 166,2 |
| 38 | 6633 | 106 | 158,6 |
| 39 | 8897 | 95 | 144,4 |
| 40 | 12800 | 103 | 130,4 |
| 41 | 15708 | 82 | 116 |
| 42 | 16142 | 43 | 116 |
| 43 | 17638 | 62 | 119 |
| 44 | 21485 | 55 | 120,2 |
| 45 | 24611 | 99 | 113 |
| 46 | 32530 | 88 | 117,6 |
| 47 | 33839 | 73 | 121 |
| 48 | 39136 | 58 | 122,8 |
| 49 | 44007 | 53 | 111,6 |
| 50 | 45982 | 47 | 134,4 |
| 51 | 46693 | 42 | 112,2 |
| 52 | 45124 | 38 | 76,6 |
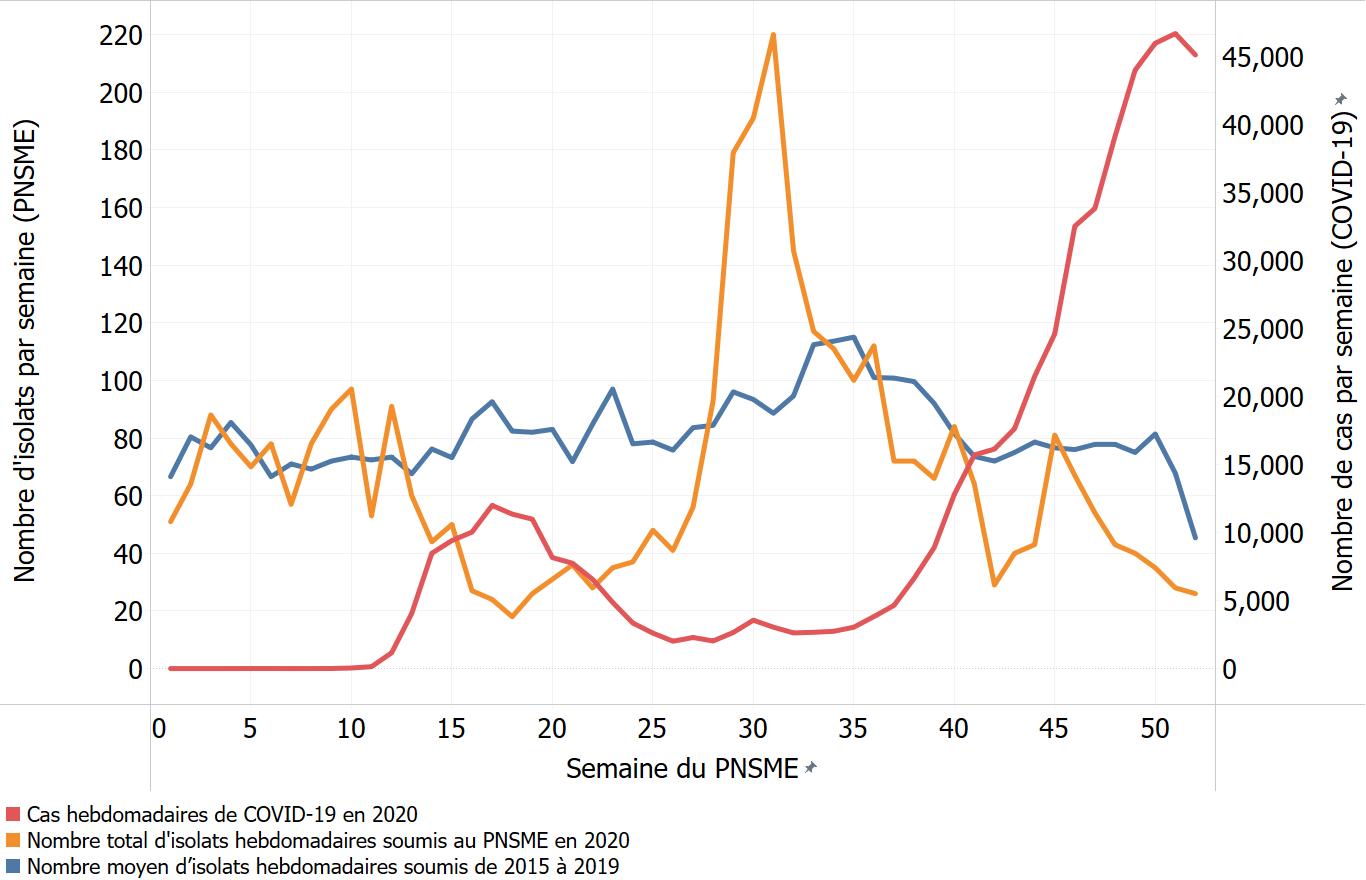
Figure 11 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 51 | 66,6 |
| 2 | 0 | 64 | 80,4 |
| 3 | 0 | 88 | 76,6 |
| 4 | 0 | 78 | 85,4 |
| 5 | 4 | 70 | 77,6 |
| 6 | 3 | 78 | 66,6 |
| 7 | 0 | 57 | 71 |
| 8 | 2 | 78 | 69,2 |
| 9 | 6 | 90 | 72 |
| 10 | 42 | 97 | 73,4 |
| 11 | 140 | 53 | 72,4 |
| 12 | 1161 | 91 | 73,4 |
| 13 | 4054 | 60 | 67,6 |
| 14 | 8479 | 44 | 76,2 |
| 15 | 9414 | 50 | 73,2 |
| 16 | 10035 | 27 | 86,6 |
| 17 | 11997 | 24 | 92,6 |
| 18 | 11359 | 18 | 82,4 |
| 19 | 10988 | 26 | 82 |
| 20 | 8159 | 31 | 83 |
| 21 | 7748 | 36 | 71,8 |
| 22 | 6564 | 28 | 84,8 |
| 23 | 4867 | 35 | 97 |
| 24 | 3353 | 37 | 78 |
| 25 | 2609 | 48 | 78,6 |
| 26 | 2013 | 41 | 75,8 |
| 27 | 2284 | 56 | 83,6 |
| 28 | 2030 | 93 | 84,4 |
| 29 | 2653 | 179 | 96 |
| 30 | 3557 | 191 | 93,4 |
| 31 | 3043 | 220 | 88,6 |
| 32 | 2622 | 145 | 94,6 |
| 33 | 2668 | 117 | 112,4 |
| 34 | 2740 | 111 | 113,6 |
| 35 | 3043 | 100 | 115 |
| 36 | 3822 | 112 | 101 |
| 37 | 4646 | 72 | 100,8 |
| 38 | 6633 | 72 | 99,6 |
| 39 | 8897 | 66 | 92 |
| 40 | 12800 | 84 | 81,6 |
| 41 | 15708 | 64 | 73,6 |
| 42 | 16142 | 29 | 72 |
| 43 | 17638 | 40 | 75 |
| 44 | 21485 | 43 | 78,6 |
| 45 | 24611 | 81 | 76,6 |
| 46 | 32530 | 67 | 76 |
| 47 | 33839 | 54 | 77,8 |
| 48 | 39136 | 43 | 77,8 |
| 49 | 44007 | 40 | 75 |
| 50 | 45982 | 35 | 81,4 |
| 51 | 46693 | 28 | 67,8 |
| 52 | 45124 | 26 | 45,4 |

Figure 12 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 22 | 46,6 |
| 2 | 0 | 26 | 63,2 |
| 3 | 0 | 33 | 55,8 |
| 4 | 0 | 38 | 67 |
| 5 | 4 | 33 | 65,4 |
| 6 | 3 | 40 | 68,6 |
| 7 | 0 | 43 | 65,2 |
| 8 | 2 | 36 | 63,4 |
| 9 | 6 | 42 | 67,4 |
| 10 | 42 | 38 | 60,8 |
| 11 | 140 | 40 | 76 |
| 12 | 1161 | 37 | 70 |
| 13 | 4054 | 39 | 72,8 |
| 14 | 8479 | 34 | 88,6 |
| 15 | 9414 | 24 | 76,6 |
| 16 | 10035 | 9 | 72,6 |
| 17 | 11997 | 11 | 72,8 |
| 18 | 11359 | 16 | 76,8 |
| 19 | 10988 | 17 | 73,8 |
| 20 | 8159 | 19 | 68,4 |
| 21 | 7748 | 15 | 63,6 |
| 22 | 6564 | 25 | 64,8 |
| 23 | 4867 | 23 | 57,2 |
| 24 | 3353 | 23 | 54,8 |
| 25 | 2609 | 25 | 59,6 |
| 26 | 2013 | 16 | 53,8 |
| 27 | 2284 | 29 | 53,2 |
| 28 | 2030 | 15 | 61,4 |
| 29 | 2653 | 15 | 62,2 |
| 30 | 3557 | 25 | 60 |
| 31 | 3043 | 40 | 57 |
| 32 | 2622 | 35 | 49,8 |
| 33 | 2668 | 30 | 57,8 |
| 34 | 2740 | 37 | 67,6 |
| 35 | 3043 | 23 | 62,8 |
| 36 | 3822 | 28 | 65 |
| 37 | 4646 | 14 | 65,4 |
| 38 | 6633 | 34 | 59 |
| 39 | 8897 | 29 | 52,4 |
| 40 | 12800 | 19 | 48,8 |
| 41 | 15708 | 18 | 42,4 |
| 42 | 16142 | 14 | 44 |
| 43 | 17638 | 22 | 44 |
| 44 | 21485 | 12 | 41,6 |
| 45 | 24611 | 18 | 36,4 |
| 46 | 32530 | 21 | 41,6 |
| 47 | 33839 | 19 | 43,2 |
| 48 | 39136 | 15 | 45 |
| 49 | 44007 | 13 | 36,6 |
| 50 | 45982 | 12 | 53 |
| 51 | 46693 | 14 | 44,4 |
| 52 | 45124 | 12 | 31,2 |
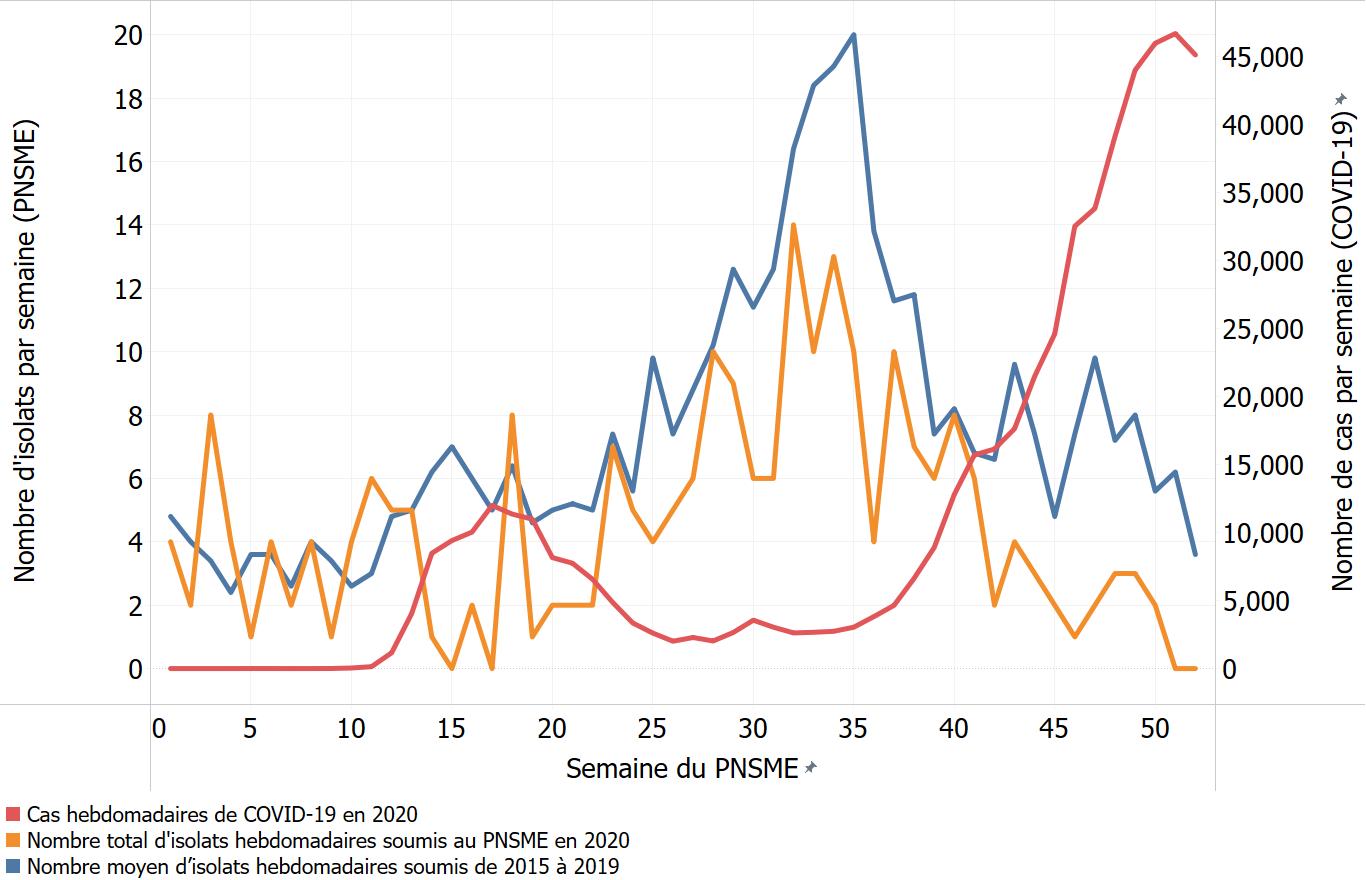
Figure 13 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 4 | 4,8 |
| 2 | 0 | 2 | 4 |
| 3 | 0 | 8 | 3,4 |
| 4 | 0 | 4 | 2,4 |
| 5 | 4 | 1 | 3,6 |
| 6 | 3 | 4 | 3,6 |
| 7 | 0 | 2 | 2,6 |
| 8 | 2 | 4 | 4 |
| 9 | 6 | 1 | 3,4 |
| 10 | 42 | 4 | 2,6 |
| 11 | 140 | 6 | 3 |
| 12 | 1161 | 5 | 4,8 |
| 13 | 4054 | 5 | 5 |
| 14 | 8479 | 1 | 6,2 |
| 15 | 9414 | 0 | 7 |
| 16 | 10035 | 2 | 6 |
| 17 | 11997 | 0 | 5 |
| 18 | 11359 | 8 | 6,4 |
| 19 | 10988 | 1 | 4,6 |
| 20 | 8159 | 2 | 5 |
| 21 | 7748 | 2 | 5,2 |
| 22 | 6564 | 2 | 5 |
| 23 | 4867 | 7 | 7,4 |
| 24 | 3353 | 5 | 5,6 |
| 25 | 2609 | 4 | 9,8 |
| 26 | 2013 | 5 | 7,4 |
| 27 | 2284 | 6 | 8,8 |
| 28 | 2030 | 10 | 10,2 |
| 29 | 2653 | 9 | 12,6 |
| 30 | 3557 | 6 | 11,4 |
| 31 | 3043 | 6 | 12,6 |
| 32 | 2622 | 14 | 16,4 |
| 33 | 2668 | 10 | 18,4 |
| 34 | 2740 | 13 | 19 |
| 35 | 3043 | 10 | 20 |
| 36 | 3822 | 4 | 13,8 |
| 37 | 4646 | 10 | 11,6 |
| 38 | 6633 | 7 | 11,8 |
| 39 | 8897 | 6 | 7,4 |
| 40 | 12800 | 8 | 8,2 |
| 41 | 15708 | 6 | 6,8 |
| 42 | 16142 | 2 | 6,6 |
| 43 | 17638 | 4 | 9,6 |
| 44 | 21485 | 3 | 7,4 |
| 45 | 24611 | 2 | 4,8 |
| 46 | 32530 | 1 | 7,4 |
| 47 | 33839 | 2 | 9,8 |
| 48 | 39136 | 3 | 7,2 |
| 49 | 44007 | 3 | 8 |
| 50 | 45982 | 2 | 5,6 |
| 51 | 46693 | 0 | 6,2 |
| 52 | 45124 | 0 | 3,6 |

Figure 14 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 3 | 3,8 |
| 2 | 0 | 3 | 4,8 |
| 3 | 0 | 9 | 5,6 |
| 4 | 0 | 10 | 6,2 |
| 5 | 4 | 7 | 5,6 |
| 6 | 3 | 8 | 5 |
| 7 | 0 | 10 | 4,2 |
| 8 | 2 | 6 | 7 |
| 9 | 6 | 7 | 6,8 |
| 10 | 42 | 8 | 3,8 |
| 11 | 140 | 4 | 4,4 |
| 12 | 1161 | 11 | 5,2 |
| 13 | 4054 | 3 | 6 |
| 14 | 8479 | 11 | 5,4 |
| 15 | 9414 | 1 | 6,4 |
| 16 | 10035 | 3 | 6 |
| 17 | 11997 | 1 | 8,4 |
| 18 | 11359 | 4 | 6,2 |
| 19 | 10988 | 3 | 7,6 |
| 20 | 8159 | 5 | 10 |
| 21 | 7748 | 2 | 4,6 |
| 22 | 6564 | 8 | 6,4 |
| 23 | 4867 | 5 | 6 |
| 24 | 3353 | 6 | 8,4 |
| 25 | 2609 | 9 | 7,8 |
| 26 | 2013 | 5 | 8,8 |
| 27 | 2284 | 12 | 9,4 |
| 28 | 2030 | 12 | 7,4 |
| 29 | 2653 | 9 | 7,6 |
| 30 | 3557 | 7 | 13,2 |
| 31 | 3043 | 15 | 8,6 |
| 32 | 2622 | 5 | 13,4 |
| 33 | 2668 | 8 | 12,2 |
| 34 | 2740 | 10 | 8 |
| 35 | 3043 | 15 | 10,2 |
| 36 | 3822 | 11 | 9,6 |
| 37 | 4646 | 6 | 9,2 |
| 38 | 6633 | 7 | 9,8 |
| 39 | 8897 | 8 | 8 |
| 40 | 12800 | 7 | 16,4 |
| 41 | 15708 | 3 | 10,6 |
| 42 | 16142 | 1 | 8,6 |
| 43 | 17638 | 3 | 6,8 |
| 44 | 21485 | 4 | 3,8 |
| 45 | 24611 | 3 | 5,6 |
| 46 | 32530 | 2 | 5,4 |
| 47 | 33839 | 4 | 6,8 |
| 48 | 39136 | 2 | 6,2 |
| 49 | 44007 | 6 | 6,2 |
| 50 | 45982 | 2 | 8,8 |
| 51 | 46693 | 2 | 4,8 |
| 52 | 45124 | 1 | 6 |

Figure 15 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 1 | 3 |
| 2 | 0 | 1 | 3,6 |
| 3 | 0 | 1 | 2,8 |
| 4 | 0 | 3 | 1,6 |
| 5 | 4 | 2 | 3,6 |
| 6 | 3 | 2 | 1,6 |
| 7 | 0 | 0 | 2,6 |
| 8 | 2 | 3 | 1,8 |
| 9 | 6 | 2 | 2,4 |
| 10 | 42 | 0 | 1,2 |
| 11 | 140 | 0 | 1,2 |
| 12 | 1161 | 4 | 1,4 |
| 13 | 4054 | 2 | 2 |
| 14 | 8479 | 2 | 2,2 |
| 15 | 9414 | 1 | 2,2 |
| 16 | 10035 | 4 | 2 |
| 17 | 11997 | 5 | 2,8 |
| 18 | 11359 | 3 | 0,6 |
| 19 | 10988 | 8 | 3,2 |
| 20 | 8159 | 1 | 2,4 |
| 21 | 7748 | 0 | 2,8 |
| 22 | 6564 | 1 | 3 |
| 23 | 4867 | 2 | 2,2 |
| 24 | 3353 | 0 | 2,8 |
| 25 | 2609 | 1 | 4 |
| 26 | 2013 | 0 | 1,6 |
| 27 | 2284 | 0 | 5,8 |
| 28 | 2030 | 1 | 5 |
| 29 | 2653 | 5 | 2,2 |
| 30 | 3557 | 6 | 3,8 |
| 31 | 3043 | 3 | 4 |
| 32 | 2622 | 5 | 3 |
| 33 | 2668 | 2 | 4,6 |
| 34 | 2740 | 6 | 3,2 |
| 35 | 3043 | 8 | 5,2 |
| 36 | 3822 | 5 | 4,4 |
| 37 | 4646 | 9 | 4,2 |
| 38 | 6633 | 5 | 5,6 |
| 39 | 8897 | 4 | 3,6 |
| 40 | 12800 | 8 | 3 |
| 41 | 15708 | 6 | 3,6 |
| 42 | 16142 | 4 | 3,2 |
| 43 | 17638 | 2 | 3,6 |
| 44 | 21485 | 3 | 2 |
| 45 | 24611 | 2 | 2,2 |
| 46 | 32530 | 3 | 2,6 |
| 47 | 33839 | 5 | 2,6 |
| 48 | 39136 | 4 | 2,4 |
| 49 | 44007 | 4 | 2 |
| 50 | 45982 | 1 | 2,6 |
| 51 | 46693 | 4 | 3 |
| 52 | 45124 | 2 | 1,8 |

Figure 16 - Équivalent textuel
| Semaine du PNSME | Cas hebdomadaires de COVID-19 en 2020 | Nombre total d'isolats hebdomadaires soumis au PNSME en 2020 | Nombre moyen d'isolats hebdomadaires soumis de 2015 à 2019 |
|---|---|---|---|
| 1 | 0 | 9 | 11,8 |
| 2 | 0 | 8 | 20,8 |
| 3 | 0 | 23 | 14,6 |
| 4 | 0 | 16 | 23,2 |
| 5 | 4 | 22 | 17,6 |
| 6 | 3 | 23 | 14,8 |
| 7 | 0 | 17 | 18,6 |
| 8 | 2 | 16 | 17,2 |
| 9 | 6 | 21 | 16,2 |
| 10 | 42 | 14 | 13,2 |
| 11 | 140 | 22 | 11,8 |
| 12 | 1161 | 14 | 15,2 |
| 13 | 4054 | 8 | 15,6 |
| 14 | 8479 | 10 | 18 |
| 15 | 9414 | 5 | 15,8 |
| 16 | 10035 | 3 | 15,8 |
| 17 | 11997 | 6 | 13,6 |
| 18 | 11359 | 3 | 16,4 |
| 19 | 10988 | 5 | 11,4 |
| 20 | 8159 | 2 | 13,4 |
| 21 | 7748 | 3 | 11,6 |
| 22 | 6564 | 3 | 13,8 |
| 23 | 4867 | 3 | 11,6 |
| 24 | 3353 | 3 | 10 |
| 25 | 2609 | 3 | 10,8 |
| 26 | 2013 | 1 | 12 |
| 27 | 2284 | 4 | 11,8 |
| 28 | 2030 | 2 | 11,4 |
| 29 | 2653 | 7 | 10 |
| 30 | 3557 | 6 | 12,6 |
| 31 | 3043 | 8 | 15 |
| 32 | 2622 | 4 | 14,4 |
| 33 | 2668 | 4 | 14,2 |
| 34 | 2740 | 8 | 16,8 |
| 35 | 3043 | 2 | 18 |
| 36 | 3822 | 2 | 16,2 |
| 37 | 4646 | 5 | 14,8 |
| 38 | 6633 | 9 | 19,4 |
| 39 | 8897 | 4 | 14,8 |
| 40 | 12800 | 5 | 16,8 |
| 41 | 15708 | 7 | 14 |
| 42 | 16142 | 1 | 16,4 |
| 43 | 17638 | 5 | 21,6 |
| 44 | 21485 | 3 | 13,4 |
| 45 | 24611 | 1 | 14,4 |
| 46 | 32530 | 4 | 11,6 |
| 47 | 33839 | 6 | 17,8 |
| 48 | 39136 | 10 | 11 |
| 49 | 44007 | 4 | 14 |
| 50 | 45982 | 1 | 17 |
| 51 | 46693 | 4 | 19,2 |
| 52 | 45124 | 2 | 10 |
Notes de bas de page
- Note de bas de page 1
-
Système canadien de surveillance des maladies à déclaration obligatoire, Agence de la santé publique du Canada : https://maladies.canada.ca/declaration-obligatoire/
- Note de bas de page 2
-
PulseNet Canada, Laboratoire national de microbiologie, Agence de la santé publique du Canada : http://www.nml-lnm.gc.ca/index-fra.htm
- Note de bas de page 3
-
Modalités canadiennes d’intervention lors de toxi-infection d’origine alimentaire (MITIOA) 2010 : en cas d’éclosion multijuridictionnelle. Agence de la santé publique du Canada : http://www.phac-aspc.gc.ca/zoono/fiorp-mitioa/index-fra.php
- Note de bas de page 4
-
https://www.who.int/fr/director-general/speeches/detail/who-director-general-s-opening-remarks-at-the-media-briefing-on-covid-19---11-march-2020 (consulté le 13 septembre 2021)
- Note de bas de page 5
-
https://pm.gc.ca/fr/nouvelles/communiques/2020/03/16/premier-ministre-annonce-de-nouvelles-mesures-cadre-de-la-reponse (consulté le 20 octobre 2021). Le 18 mars 2020, le Canada a imposé des restrictions concernant les voyages internationaux, ce qui correspond à la semaine 12 du PNSME.