Analyse de la protéase principale du SARS-CoV-2 et présence de mutants Omicron SARS-CoV-2 résistants au nirmatrelvir

 Téléchargez cet article en format PDF (214 ko)
Téléchargez cet article en format PDF (214 ko)Publié par : L'Agence de la santé publique du Canada
Numéro : RMTC : Volume 50-10, octobre 2024 : COVID-19 après la pandémie
Date de publication : octobre 2024
ISSN : 1719-3109
Soumettre un article
À propos du RMTC
Naviguer
Volume 50-10, octobre 2024 : COVID-19 après la pandémie
Surveillance
L'analyse à grande échelle de la protéase principale du virus SRAS-CoV-2 révèle la présence marginale de mutants du variant Omicron du SRAS-CoV-2 résistants au nirmatrelvir en Ontario, au Canada, de décembre 2021 à septembre 2023
Venkata Duvvuri1,2, Fatima Shire1,3, Sandra Isabel1, Thomas Braukmann1, Shawn Clark1, Alex Marchand-Austin1, Alireza Eshaghi1, Hina Bandukwala1, Nobish Varghese1, Ye Li1,3, Karthikeyan Sivaraman1, Hadia Hussain1, Kirby Cronin1, Ashleigh Sullivan1, Aimin Li1, Austin Zygmunt1,4, Karam Ramotar1, Julianne Kus1,2, Maan Hasso1, Antoine Corbeil1, Jonathan Gubbay1, Samir Patel1,2
Affiliations
1 Santé publique Ontario, Toronto, ON
2 Département médecine de laboratoire et pathobiologie, Faculté de médecine Temerty, Université de Toronto, Toronto, ON
3 Division des biostatistiques, École de santé publique Dalla Lana, Université de Toronto, Toronto, ON
4 Département de médecine familiale, Université d'Ottawa, Ottawa, ON
Correspondance
Citation proposée
Duvvuri VR, Shire F, Isabel S, Braukmann T, Clark ST, Marchand-Austin A, Eshaghi A, Bandukwala H, Varghese N, Li Y, Sivaraman K, Hussain H, Cronin K, Sullivan A, Li A, Zygmunt A, Ramotar K, Kus J, Hasso M, Corbeil A, Gubbay JB, Patel SN. L'analyse à grande échelle de la protéase principale du virus SRAS-CoV-2 révèle la présence marginale de mutants du variant Omicron du SRAS-CoV-2 résistants au nirmatrelvir en Ontario, au Canada, de décembre 2021 à septembre 2023. Relevé des maladies transmissibles au Canada 2024;50(10):399–409. https://doi.org/10.14745/ccdr.v50i10a05f
Mots-clés : SRAS-CoV-2, Omicron, Paxlovid, nirmatrelvir-ritonavir, gène de la protéase principale (Mpro), mutations résistantes in vitro, surveillance de la génomique, Ontario
Résumé
Contexte : En réponse à la pandémie de COVID-19, un nouvel antiviral oral appelé nirmatrelvir-ritonavir (PaxlovidMC) a été autorisé au Canada en janvier 2022. Des études in vitro ont signalé des mutations dans la protéine Mpro qui peuvent être associées au développement de la résistance au nirmatrelvir.
Objectifs : Étudier la prévalence, la pertinence et les modèles temporels des mutations de Mpro chez les lignées d'Omicron du SRAS-CoV-2 en Ontario, au Canada.
Méthodes : Au total, 93 082 séquences de gènes Mpro de décembre 2021 à septembre 2023 ont été analysées. Les mutations in vitro de Mpro ont été comparées à notre base de données à l'aide de pipelines de données internes pour déterminer la résistance au nirmatrelvir. Une régression binomiale négative a été réalisée pour analyser les tendances temporelles des chiffres de mutation de Mpro au cours de la période étudiée.
Résultats : Une tendance à la baisse a été observée dans les mutations non synonymes des séquences Mpro, montrant une réduction de 7,9 % (IC à 95 % : 6,5 %–9,4 %; p < 0,001) tous les 30 jours. La mutation P132H était la plus répandue (plus de 95 %) dans toutes les lignées d'Omicron. Des mutations in vitro résistantes au nirmatrelvir ont été trouvées dans 3,12 % (n = 29/929) des lignées d'Omicron avec un très faible dénombrement, allant de 1 à 19. Seules deux mutations, A7T (n = 19) et M82I (n = 9), ont montré une présence temporelle entre le BA.1.1 en 2022 et le BQ.1.2.3 en 2022, respectivement.
Conclusion : Les observations suggèrent qu'en septembre 2023, aucune résistance importante ou généralisée au nirmatrelvir ne s'était développée parmi les variants Omicron du SRAS-CoV-2 en Ontario. Cette étude souligne l'importance de créer des systèmes de surveillance automatisés pour suivre l'émergence de mutations résistantes au nirmatrelvir dans le virus SRAS-CoV-2, en utilisant les données génomiques générées en temps réel.
Introduction
Le nirmatrelvir-ritonavir (marque de commerce PaxlovidMC, Pfizer Inc.) est un traitement antiviral administré par voie orale. Cette combinaison a reçu une autorisation d'utilisation d'urgence de la Food and Drug Administration des États-Unis en décembre 2021 Note de bas de page 1Note de bas de page 2Note de bas de page 3. Le nirmatrelvir-ritonavir a par la suite été approuvé par Santé Canada pour les adultes atteints de la COVID-19 qui présentaient un risque élevé de devenir une maladie grave en janvier 2022 Note de bas de page 4Note de bas de page 5. Le nirmatrelvir (PF-07322332), un composant actif du Paxlovid, est un nouvel inhibiteur de la protéase 3 chymotrypsine du SRAS-CoV-2 (3CLpro) ou de la protéase principale (Mpro, aussi appelée protéine non structurale, nsp5), qui est essentielle pour la réplication et l'assemblage viraux. Ce mécanisme inhibiteur empêche la production de nouveaux virus dans les cellules infectées Note de bas de page 6. Fait important, le nirmatrelvir est très spécifique à la protéase virale, ce qui réduit le risque d'effets hors cible sur les protéases humaines Note de bas de page 7. Le ritonavir inhibe l'enzyme cytochrome P4503A4 (CYP3A4), une importante enzyme hépatique métabolisant les médicaments chez l'humain, augmentant les concentrations plasmatiques du nirmatrelvir in vivo Note de bas de page 8.
Les études d'efficacité clinique sur le nirmatrelvir-ritonavir ont fait état d'un nombre moins élevé de visites à l'urgence, d'un nombre moins élevé d'hospitalisations et d'un taux de mortalité moindre, toutes causes confondues, chez les patients infectés par les variants préoccupants du SRAS-CoV-2 (Delta B.1.617.2 et Omicron B.1.1.529, BA.2, BA2.12.1, BA.4 et BA.5) Note de bas de page 9Note de bas de page 10Note de bas de page 11Note de bas de page 12. Une étude d'observation rétrospective menée en Ontario, au Canada, a révélé une réduction importante des admissions à l'hôpital en raison de la COVID-19 et de la mortalité toutes causes confondues chez les patients externes qui ont utilisé le nirmatrelvir-ritonavir entre avril et août 2022, et des avantages plus importants ont été constatés chez les personnes qui étaient sous-vaccinées ou non vaccinées et âgées de 70 ans et plus Note de bas de page 13. Le Programme canadien de surveillance des infections nosocomiales a révélé que 13 % (n = 490/3 731) des patients adultes atteints de la COVID-19 ont reçu le nirmatrelvir-ritonavir, au moment de l'admission ou de l'hospitalisation au Canada, bien que les résultats sur l'efficacité du traitement ne soient toujours pas déclarés Note de bas de page 14.
L'efficacité thérapeutique du nirmatrelvir-ritonavir peut être influencée par l'émergence de variants résistants. Compte tenu de l'évolution continue du virus SRAS-CoV-2 et des pressions de sélection découlant de l'introduction du nirmatrelvir-ritonavir, une résistance est susceptible d'émerger Note de bas de page 15. Des données probantes démontrant l'existence in vitro de variants du SRAS-CoV-2 résistants au nirmatrelvir Note de bas de page 16Note de bas de page 17Note de bas de page 18, de la puissance variable du nirmatrelvir pour différents coronavirus humains Note de bas de page 19 et de la résistance d'autres virus aux inhibiteurs de la protéase Note de bas de page 20 appuient la nécessité d'une surveillance continue des séquences de gènes Mpro du SRAS-CoV-2 pour identifier rapidement les mutations qui peuvent affecter la puissance du nirmatrelvir. Une telle surveillance génomique pourrait fournir des informations sur les mécanismes de l'évasion antivirale qui sont cruciaux pour les orientations des politiques et pour le développement de la prochaine génération d'inhibiteurs Mpro Note de bas de page 18.
Cette étude visait à étudier la prévalence, la pertinence et les modèles temporels des mutations de Mpro chez les lignées du SRAS-CoV-2 en circulation en Ontario. Tout d'abord, nous avons effectué une revue de la littérature scientifique et grise (de mai 2022 à août 2023) afin de dresser une liste des mutations de Mpro qui ont été caractérisées comme conférant une résistance in vitro au nirmatrelvir Note de bas de page 21. Cette liste a ensuite été utilisée pour identifier la présence de mutations résistantes au nirmatrelvir dans l'ensemble de données. Nous avons ensuite analysé 93 082 séquences Mpro dérivées de spécimens cliniques positifs au variant Omicron du SRAS-CoV-2 séquencés en Ontario entre décembre 2021 et septembre 2023.
Méthodes
Sélection des échantillons cliniques et séquençage du génome entier du SRAS-CoV-2
Les laboratoires de diagnostic de l'Ontario ont fourni une proportion de tous les spécimens cliniques positifs au SRAS-CoV-2 à des laboratoires de séquençage du génome entier (SGE) désignés dans le cadre du Réseau génomique COVID-19 de l'Ontario Note de bas de page 22. Les critères acceptables pour l'échantillonnage du SGE comprenaient un seuil de cycle de réaction en chaîne par polymérase (PCR) du SRAS-CoV-2 de 30 ou moins et un volume d'échantillon suffisant. La proportion d'échantillonnage variait de 10 % à 100 % et a été rajustée au fil du temps en fonction du nombre de cas prévu et de la capacité de séquençage du Réseau génomique COVID-19 de l'Ontario de décembre 2021 à septembre 2023. Le test PCR diagnostique du SRAS-CoV-2/COVID-19 était limité aux populations à risque élevé Note de bas de page 23Note de bas de page 24 et, par conséquent, la surveillance représentative ne concerne que les populations testées au moment de l'échantillonnage.
Principales séquences de protéase du SRAS-CoV-2
Les données de séquence brutes de la plate-forme Illumina ont été analysées à l'aide du pipeline ARTIC (version 1.7) (pipeline de l'Institut ontarien de recherche sur le cancer) et de la version 1.4 du schéma d'amorçage ARTIC. Le filtrage de la qualité post-analyse a été effectué à l'aide de la version 1.8 de ncov-tools. Les échantillons ont été annotés pour la lignée avec Pangolin (version 4.3) en utilisant les constellations (version 0.1.12) (Pangolin-assignment [version 1.15.1], Scorpio 0.3.17 et usher 0.5.6). Le pipeline ARTIC nanopolish v1.3.0-dev (rustine +0.3.1) et la version associée de ncov-tools ont été utilisés pour les échantillons séquencés sur la plate-forme nanopore. Toutes les séquences disponibles du gène Mpro du variant Omicron du SRAS-CoV-2 (n = 93 082 séquences uniques) ont été recueillies entre le 1er décembre 2021 et le 21 septembre 2023 à partir de la base de données du SGE du SRAS-CoV-2 de Santé publique Ontario (base de données du SGE du SRAS-CoV-2 de SPO). Ces séquences Mpro ont été testées par rapport au génome de référence SRAS-COV-2, Wuhan-Hu-1 (accession no NC_045512.2), afin d'identifier les mutations synonymes et non synonymes dans toutes les lignées d'Omicron.
Suivi temporel des principales mutations de protéases dans les lignées d'Omicron
Un pipeline interne de données scientifiques a été élaboré dans Python (version 3.9.16) pour suivre la temporalité et la prévalence des mutations de Mpro observées dans les lignées d'Omicron en Ontario. Un modèle additif généralisé avec fente cubique restreinte a été adapté au dénombrement des mutations transformées en logarithmes. Nous avons examiné les tendances des mutations non synonymiques du Mpro au cours de la période de l'étude; à partir de ces tendances, une régression binomiale négative (paquet R mgcv version 1.9-0) a été utilisée pour modéliser le déclin du nombre de mutations non synonymiques au fil du temps.
Résultats
Un total de 93 082 séquences de gènes Mpro correspondant à 929 lignées d'Omicron du SRAS-CoV-2 en Ontario ont été analysées. Les lignées d'Omicron ont été regroupées en fonction de leur prévalence par rapport au nombre total de séquences analysées : faible (moins d'un pour cent) ou élevée (supérieure ou égale à un pour cent). Douze lignées du SRAS-CoV-2 ont été classées comme ayant une prévalence élevée. Les cinq lignées présentant la prévalence la plus élevée au cours de la période définie sont les suivantes : BA.1.1 (9,3 %), XBB.1.5 (8,3 %), BQ.1.1 (7,8 %), BA.2 (7,4 %) et BA.5.2.1 (6,0 %).
Nous avons étudié l'évolution des séquences de nucléotides Mpro du SRAS-CoV-2; nous avons observé des variations cycliques pour les comptages totaux des mutations synonymes (aucun changement dans la séquence des protéines) et non synonymes (changement dans la séquence des protéines). La régression binomiale négative sur les mutations non synonymiques de Mpro a montré une diminution de 7,9 % (IC à 95 % : 6,5 %–9,4 %; p < 0,001) du nombre de mutations tous les 30 jours (figure 1). La charge mutationnelle non synonyme, avec des séquences porteuses d'au moins une mutation dans la séquence de protéines Mpro, représentait environ 67,7 % (207 AA/306 AA de Mpro) (figure 2). Le tableau 1 présente les détails des lignées à prévalence faible et élevée avec des mutations non synonymes de Mpro signalées dans au moins 10 séquences des données de séquence totales recueillies pour chaque lignée. Par exemple, la mutation T21I est observée dans 31 des 2 801 séquences BA.2.12.1 totales au cours de la période d'étude. Seules six mutations, L67, L75, K90, A116, P184 et R279, se sont avérées communes dans les lignées à forte et faible prévalence; cependant, aucune de ces mutations n'était pertinente pour les mutations in vitro résistantes au nirmatrelvir.
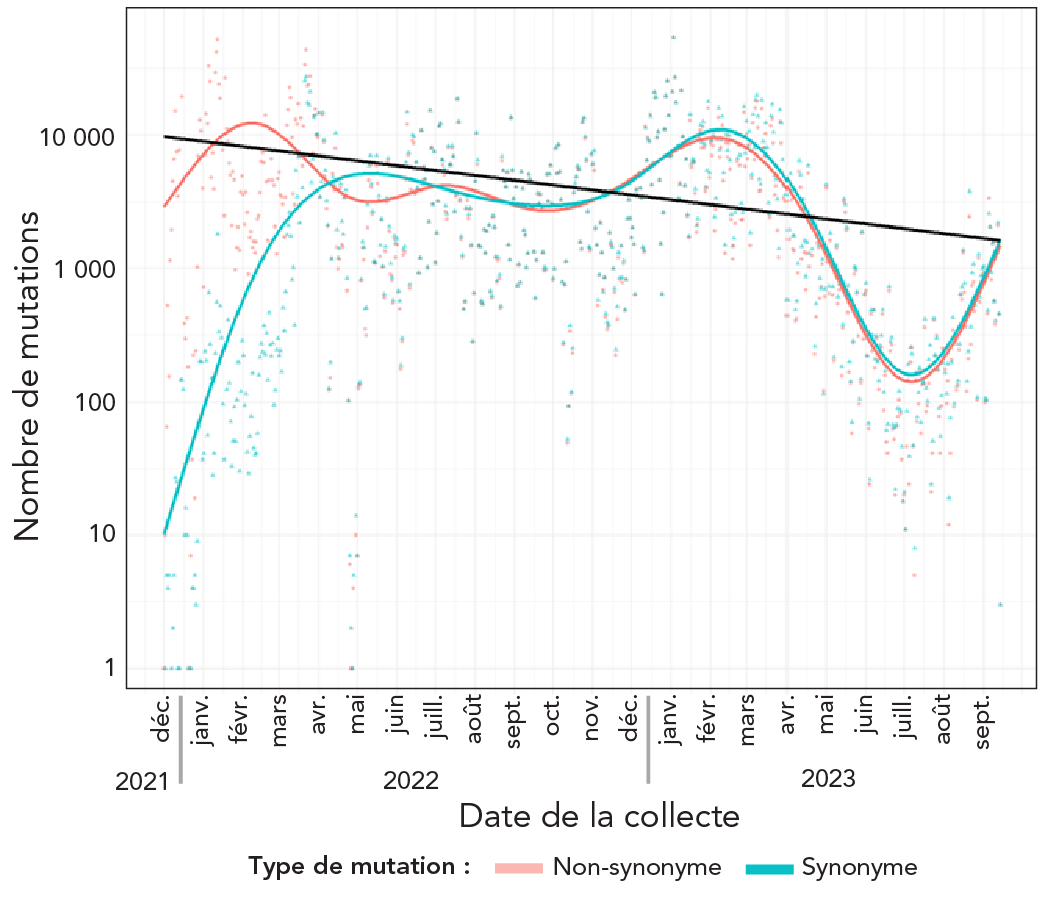
Figure 1 : Description textuelle
Cette figure présente le nombre de mutations observées au fil du temps dans la principale protéine protéase des lignées du variant Omicron du SRAS-CoV-2, classées par types de mutations non-synonymes (ligne rouge) et synonymes (ligne bleue). Au total, 93 082 séquences nucléotidiques ont été analysées. Les données montrent une augmentation graduelle des deux types de mutations dans les lignées circulant au début de 2022, suivie d'une baisse notable des lignées à partir de mai 2023. Dans le cas particulier des mutations non-synonymiques, il y a une diminution statistiquement significative du nombre de mutations tous les 30 jours, comme le montre la régression binomiale négative.
Abréviation : Mpro, protéase principale
- Note de bas de page a
-
Chaque cercle ou point plein représente une mutation et est codé en couleur sur la base des détails structurels de la protéine Mpro Note de bas de page 25. Le nombre de mutations est la valeur absolue observée des mutations à chaque position. L'axe des ordonnées transformé en logarithme présente le nombre de mutations
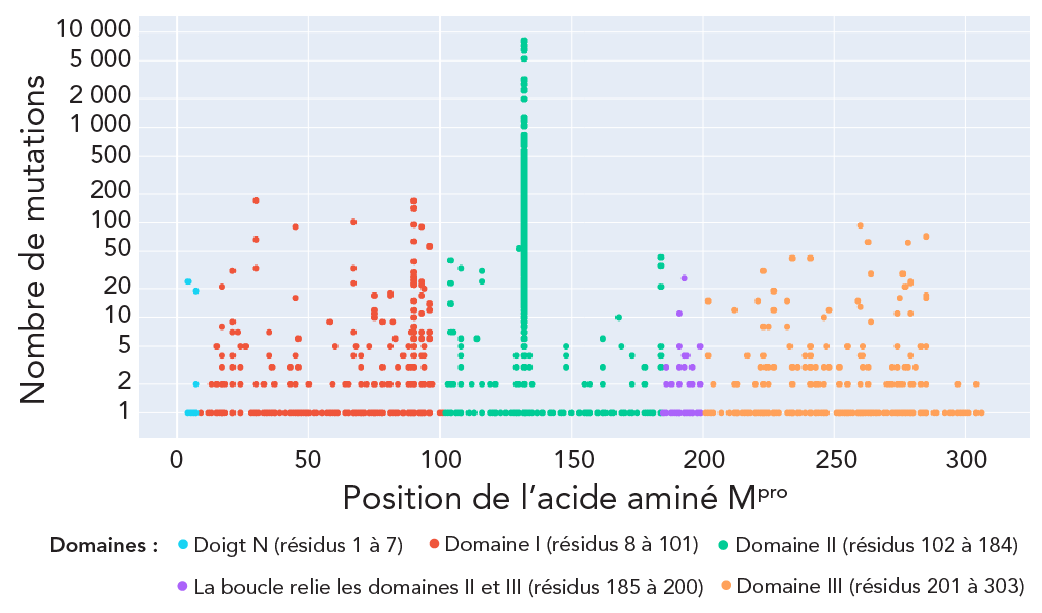
Figure 2 : Description textuelle
La figure présente une distribution visuelle détaillée des mutations non-synonymes observées dans les différents domaines de la principale protéine protéase des lignées du variant Omicron du SRAS-CoV-2. L'axe des abscisses indique les positions des acides aminés, et l'axe des ordonnées représente le nombre de mutations sur une échelle logarithmique. Conformément à la littérature sur les mutations les plus répandues chez les lignées d'Omicron, la 132e position dans le domaine II, qui correspond à la mutation P132H, a affiché le point culminant dans le dénombrement des mutations et était présente dans plus de 95 % des lignées d'Omicron analysées dans la présente étude.
Abréviation : Mpro, protéase principale
- Note de bas de page a
-
Chaque cercle solide ou point représente une mutation et est codé en couleur sur la base des détails structurels de la protéine Mpro Note de bas de page 25. Le nombre de mutations est la valeur absolue observée des mutations à chaque position. L'axe des ordonnées transformé en logarithme présente le nombre de mutations
| Région de structure de Mpro | MutationNote de bas de page a | Lignée Pango | Prévalence de la lignéeNote de bas de page b | Séquences totales | Nombre de séquences avec mutation | FréquenceNote de bas de page c | Observation de la littérature antérieure (référence) |
|---|---|---|---|---|---|---|---|
| Doigt N (1 à 7 AA) |
R4K | BQ.1.2 | Faible | 267 | 24 | 8,99 | La mutation contribue à la dimérisation de Mpro Note de bas de page 26 |
| A7T | BA.1.1 | Élevée | 8 143 | 19 | 0,23 | La mutation contribue à la dimérisation de Mpro Note de bas de page 27Note de bas de page 28Note de bas de page 29Note de bas de page 30 | |
| Domaine I (8 à 101 AA) |
M17V | XBB.1.5 | Élevée | 7 281 | 18 | 0,25 | - |
| T21I | BA.2.12.1 | Élevée | 2 801 | 31 | 1,11 | Étude in vitro signalée comme mutation fondatrice ou précurseur Note de bas de page 18 | |
| L30I | BQ.1.3 | Faible | 33 | 33 | 100 | L30F, une mutation résistante au nirmatrelvir signalée in vitro Note de bas de page 9, mais L30I n'a pas été testée | |
| BQ.1.3.1 | Faible | 170 | 170 | 100 | |||
| BQ.1.3.2 | Faible | 66 | 66 | 100 | |||
| T45N | BE.4 | Faible | 90 | 90 | 100 | - | |
| BE.4.1 | Faible | 16 | 16 | 100 | |||
| CQ.2 | Faible | 16 | 16 | 100 | |||
| L67S | BA.5.2.1 | Élevée | 5 312 | 23 | 0,43 | - | |
| L67V | BF.14 | Faible | 46 | 32 | 69,57 | - | |
| BQ.1.1.40 | Faible | 448 | 101 | 22,54 | |||
| L75F | BA.1.1 | Élevée | 8 143 | 11 | 0,14 | - | |
| BA.2 | Élevée | 6 551 | 12 | 0,18 | |||
| BA.4.6 | Élevée | 1 250 | 17 | 1,36 | |||
| BA.5.5 | Faible | 705 | 10 | 1,42 | |||
| S81C | XBB.1.5 | Élevée | 7 281 | 18 | 0,25 | - | |
| K90R | BA.1.1 | Élevée | 8 143 | 95 | 1,17 | Mutation prévalente dans les variants Beta (B.1.351) Note de bas de page 27 | |
| BA.2 | Élevée | 6 551 | 140 | 2,14 | |||
| BA.2.12.1 | Élevée | 2 801 | 23 | 0,82 | |||
| BA.5.2 | Élevée | 3 083 | 24 | 0,78 | |||
| BA.5.2.1 | Élevée | 5 312 | 25 | 0,47 | |||
| BQ.1 | Élevée | 1 987 | 12 | 0,6 | |||
| BQ.1.1 | Élevée | 6 513 | 39 | 0,6 | |||
| XBB.1.5 | Élevée | 7 281 | 27 | 0,37 | |||
| BA.2.3 | Faible | 751 | 166 | 22,1 | |||
| BA.5.9 | Faible | 70 | 10 | 14,29 | |||
| BF.14 | Faible | 46 | 29 | 63,04 | |||
| BF.21 | Faible | 104 | 12 | 11,54 | |||
| BQ.1.1.51 | Faible | 133 | 15 | 11,28 | |||
| T93I | BA.2.12.1 | Élevée | 2 801 | 11 | 0,39 | - | |
| XBB.1.5 | Élevée | 7 281 | 6 | 0,08 | |||
| A94V | BU.1 | Faible | 20 | 20 | 100 | - | |
| P96S | BQ.1.1 | Élevée | 6 513 | 56 | 0,86 | - | |
| P96L | XBB.1.5 | Élevée | 7 281 | 14 | 0,19 | - | |
| Domaine II (102 à 184 AA) |
V104I | BN.1.4 | Faible | 15 | 14 | 93,33 | - |
| P108T | BA.5.2.1 | Élevée | 5 312 | 33 | 0,62 | - | |
| A116V | XBB.1.5 | Élevée | 7 281 | 31 | 0,43 | - | |
| A116T | BQ.1.2 | Faible | 267 | 24 | 8,99 | - | |
| M130L | BA.5.2.9 | Faible | 534 | 53 | 9,93 | - | |
| P168S | BA.5.1 | Élevée | 2 452 | 10 | 0,41 | Mutation prévalente dans les lignées préalables à Omicron Note de bas de page 17 | |
| P184S | BA.1.1 | Élevée | 8 143 | 21 | 0,26 | - | |
| BA.2.3 | Faible | 751 | 43 | 5,73 | - | ||
| P184L | BQ.1.14 | Faible | 250 | 35 | 14 | - | |
| Boucle (185 à 200 AA) |
A193V | XBB.1.16.1 | Faible | 227 | 4 | 1,76 | - |
| Domaine III (201 à 303 AA) |
V202I | BQ.1.2.3 | Faible | 289 | 15 | 5,19 | - |
| V212I | FL.7 | Faible | 17 | 12 | 70,59 | - | |
| N221S | BQ.1.22 | Faible | 112 | 15 | 13,39 | - | |
| F223L | BN.1.3.1 | Faible | 31 | 31 | 100 | - | |
| L227F | BA.5.2.1 | Élevée | 5 312 | 19 | 0,36 | - | |
| BF.7 | Élevée | 999 | 12 | 1,2 | |||
| L232F | BA.5.2 | Élevée | 3 083 | 14 | 0,45 | - | |
| A234V | BQ.1.13 | Faible | 435 | 42 | 9,66 | - | |
| P241L | XBB.1.5 | Élevée | 7 281 | 42 | 0,58 | - | |
| H246Y | BA.5.1.15 | Faible | 10 | 10 | 100 | - | |
| D248N | BU.1 | Faible | 20 | 12 | 60 | - | |
| A260V | BQ.1.1 | Élevée | 6 513 | 92 | 1,41 | Aucun impact n'a été démontré sur le pouvoir réducteur du médicament dans les essais biochimiques Note de bas de page 2Note de bas de page 3 | |
| D263A | BQ.1.1 | Élevée | 6 513 | 62 | 0,95 | - | |
| M264I | XBB.1.5 | Élevée | 7 281 | 29 | 0,4 | - | |
| N274T | BQ.1.1 | Élevée | 6 513 | 11 | 0,17 | - | |
| N274S | BQ.1.14 | Faible | 250 | 10 | 4 | - | |
| G275S | BN.1.5.2 | Faible | 18 | 16 | 88,89 | - | |
| M276I | BA.5.1.2 | Faible | 154 | 28 | 18,18 | - | |
| N277I | BA.5.1.23 | Faible | 236 | 21 | 8,9 | - | |
| G278R | BF.7 | Élevée | 999 | 58 | 5,81 | - | |
| R279C | BF.7 | Élevée | 999 | 23 | 2,3 | - | |
| BA.5.5 | Faible | 705 | 22 | 3,12 | - | ||
| BF.1 | Faible | 95 | 11 | 11,58 | - | ||
| A285T | BA.2 | Élevée | 6 551 | 16 | 0,24 | La mutation contribue à la dimérisation de Mpro Note de bas de page 26 et à la diminution potentielle de l'efficacité catalytique de Mpro Note de bas de page 31 | |
| BF.1 | Faible | 95 | 11 | 11,58 | - | ||
Abréviations : Mpro, protéase principale; -, sans objet Notes de bas de page
|
|||||||
Profil des mutations hautement prévalentes documentées dans les lignées d'Omicron du SRAS-CoV-2, en Ontario
Des neuf mutations de Mpro les plus prévalentes dans le SRAS-CoV-2 (G15S, T21I, K88R, L89F, K90R, P108S, P132H, L205V et A260V) Note de bas de page 17Note de bas de page 26Note de bas de page 27Note de bas de page 32, mais avec une susceptibilité non altérée au nirmatrelvir Note de bas de page 2Note de bas de page 3, seule la P132H accumulée à une fréquence perceptible finit par représenter plus de 95 % des lignées d'Omicron en Ontario Note de bas de page 27. La mutation K90R a été observée dans les lignées d'Omicron suivantes : BA.1.1, BA.2, BA.2.12.1, BA.5.2, BA.5.2.1, BQ.1, BQ.1.1 et XBB.1.15 (les taux dans la lignée allaient de 0,37 % à 2,14 %). Bien que la substitution d'A260V ait été observée dans 1,41 % (n = 92/6 513 séquences) des variants BQ.1.1 circulent en 2022, la mutation T21I s'est accumulée dans la lignée BA.2.12.1 avec une fréquence de mutation de 1,1 % (n = 31/2 801 séquences).
Faible prévalence et absence de temporalité de la résistance au nirmatrelvir chez les lignées d'Omicron du SRAS-CoV-2, en Ontario
Seize des 34 mutations in vitro caractérisées comme résistantes au nirmatrelvir Note de bas de page 2Note de bas de page 3Note de bas de page 32Note de bas de page 33, correspondant à A7T/S/V (doigt N Mpro), G15S, L30F, L50F, M82I (domaine I de Mpro), P132S, T135I, E166V, A173S/T/V (domaine II de Mpro), Q189K, T196A (boucle reliant les domaines Mpro II et III de Mpro), W207S, D248E, A260T, D263E et A266V (domaine III de Mpro), ont été observés avec une spécificité de la lignée (3,12 %, n = 29/929 lignées) (figure 3). La charge de ces mutations variait de 1 à 19 comptages, l'A7T étant le plus fréquemment observé dans BA.1.1 (taux de lignée = 0,23 %, n = 19/8 143 séquences; observé seulement une fois dans FT.1, XBB.1.22 et XBB.1.5), suivi de M82I dans neuf séquences de BQ.1.2.3. Le reste a été observé dans n = 4 séquences de BA.4.6 pour la mutation A173T, selon le tableau A1 de l'appendice. Seuls A7T et M82I affichaient une certaine temporalité; A7T a été remarqué pendant les semaines 3, 4 et 10 à 15 en 2022 parmi les lignées BA.1.1 et M82I pendant les semaines 46 à 51 à la fin de 2022 parmi les lignées BQ.1.2.3 (figure 3).
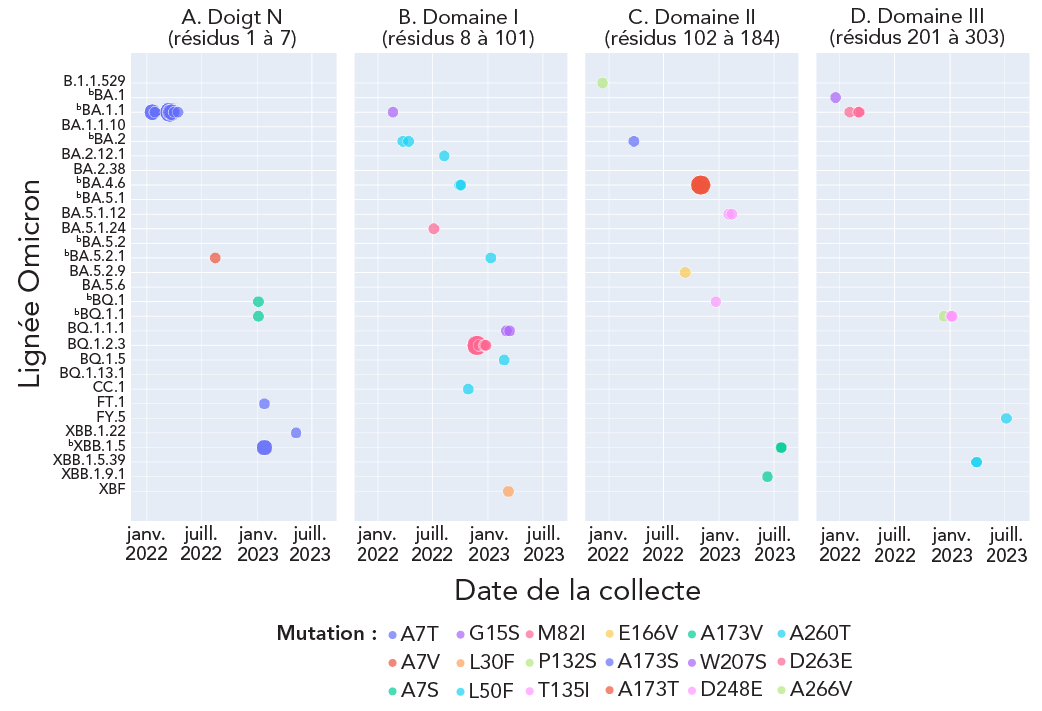
Figure 3 : Description textuelle
Cette figure illustre la prévalence et la temporalité des mutations Mpro qui ont conféré une résistance in vitro au nirmatrelvir dans les lignées d'Omicron recueillies de décembre 2021 à septembre 2023. Parmi les mutations analysées, seules A7T et M82I présentaient une prévalence perceptible dans les lignées BA.1.1 et BQ.1.2.3 d'Omicron, respectivement. En outre, A7T et M82I sont également les seules mutations qui présentent une temporalité. Les autres mutations ont été observées dans quatre séquences ou moins des lignées analysées, sans temporalité observable.
Notes de bas de page
- Note de bas de page a
-
Chaque cercle ou point plein représente un compte de la mutation Mpro codée par couleur correspondante et la taille du cercle plein indique sa valeur de comptage. Les mutations de Mpro répertoriées correspondent aux régions structurelles de Mpro de la mutation Note de bas de page 25
- Note de bas de page b
-
Indique les lignées très répandues. Le tableau A1 de l'appendice indique le nombre de chaque mutation et les lignées associées au fil du temps
Nous avons également examiné notre base de données pour les mutants doubles Note de bas de page 18Note de bas de page 21Note de bas de page 33Note de bas de page 34Note de bas de page 35, triples Note de bas de page 2Note de bas de page 3, quadruples et quintuples Note de bas de page 2Note de bas de page 3, comme on l'a signalé dans la littérature, puisque ces mutations multiples ont le potentiel de conférer une résistance synergique au nirmatrelvir. Cependant, aucune de ces mutations n'a été identifiée au sein des lignées de SRAS-CoV-2 en circulation au moment de l'échantillonnage en Ontario.
Discussion
Une analyse exhaustive des séquences de lignées de Mpro d'Omicron du SRAS-CoV-2 en Ontario a révélé qu'environ 3 % des lignées (n = 29/929) présentaient des mutations de Mpro caractérisées in vitro résistantes au nirmatrelvir, sans modèle temporel discernable.
Conformément à la littérature mondiale Note de bas de page 26Note de bas de page 27Note de bas de page 32, la mutation faux-sens P132H dans la région du domaine II de la structure de Mpro était la plus répandue, avec une prévalence supérieure à 95 % dans toutes les lignées d'Omicron de l'Ontario. En outre, K90R, la mutation la plus répandue des variants Bêta, a été observée avec une prévalence modeste dans les lignées d'Omicron de l'Ontario Note de bas de page 27. Cependant, malgré leur prédominance, ces deux mutations n'ont pas été signalées comme réduisant la puissance du nirmatrelvir Note de bas de page 2Note de bas de page 3. Les évaluations structurelles de Mpro ont révélé que les deux mutations (P132H et K90R) sont distales par rapport au site de liaison du nirmatrelvir et ne modifient donc pas la conformation structurelle au niveau ou autour du site de liaison Note de bas de page 34. La substitution A260V, une autre mutation de Mpro très prévalente observée dans les variants BQ.1.1 signalés comme un polymorphisme naturel peu fréquent, a été signalée dans l'essai clinique EPIC-HR avec impact sur la résistance au nirmatrelvir en attendant Note de bas de page 2Note de bas de page 3.
Dans notre ensemble de données, nous avons observé une faible fréquence de mutations ponctuelles de Mpro telles que T21I, P252L et T304I, qui sont connues pour fonctionner comme des mutations « précurseurs » de l'émergence de la résistance au nirmatrelvir dans le SRAS-CoV-2 Note de bas de page 18. Ces trois mutations peuvent limiter indépendamment la réplication du virus SRAS-CoV-2 Note de bas de page 32, mais aucune donnée n'est disponible sur leur contribution potentielle à la résistance. Aucune des mutations à faible prévalence observées dans notre ensemble de données, y compris A7T et M82I, n'est impliquée dans la résistance au nirmatrelvir Note de bas de page 35. Notamment, A7 est situé dans la région du doigt N, connue pour jouer un rôle dans la dimérisation qui est cruciale pour l'activité de l'enzyme de Mpro Note de bas de page 28Note de bas de page 29. Selon Iketani et al. Note de bas de page 30, les variants présentant des mutations de A7 à V/C/S/T ont une activité protéasique comparable à celle du type sauvage. De manière cohérente, les études structurales suggèrent que la substitution de l'alanine par la thréonine en position 7 n'a qu'un effet modeste sur l'activité protéasique de Mpro, soit une réduction de l'efficacité 1,5 fois Note de bas de page 29. Dans l'ensemble, ces études suggèrent que les mutations A7V/S/T observées dans les variants BA.1.1 au début de 2022 n'ont probablement pas contribué à la résistance au nirmatrelvir ou à l'activité protéasique. Aucune mutation in vitro connue résistante au nirmatrelvir n'a été trouvée (du 17 au 30 septembre 2023) dans les variants qui circulaient récemment en Ontario, EG.5.1.1, FL.1.5.1, HV.1, HK.3 et XBB.1.16.6 Note de bas de page 36.
La tendance à la baisse observée dans les mutations Mpro non synonymes (figure 1) suggère la possibilité d'une réduction de l'hétérogénéité parmi les variants viraux circulants de l'Ontario ou d'une diminution de la propension de la protéine Mpro à évoluer en réponse à une pression sélective Note de bas de page 27. Par ailleurs, Schwartz et al. Note de bas de page 13 a rapporté que seulement 5 % des patients (n = 8 876/177 545) avaient été traités par le nirmatrelvir-ritonavir entre le 4 avril et le 31 août 2022, en Ontario. Ces données, bien que spécifiques à une brève période d'étude dans le cadre de notre étude, suggèrent une pression de sélection limitée, contribuant potentiellement à la plus faible prévalence des variants Omicron résistants aux antiviraux observés dans la population étudiée. Dans l'ensemble, nos observations suggèrent que les variants Omicron analysés au moment de la période d'étude n'ont pas encore développé de résistance significative et généralisée au nirmatrelvir Note de bas de page 37.
Points forts et limites
L'un des principaux points forts de l'étude est l'analyse à grande échelle des séquences de Mpro des lignées d'Omicron qui ont circulé entre décembre 2021 et septembre 2023 en Ontario. Une analyse exhaustive a permis d'obtenir des renseignements sur les mutations in vitro pertinentes à la résistance au nirmatrelvir (fréquences de mutation et temporalité), l'activité de la protéase et l'identification des mutations de la fonction inconnue propre à notre ensemble de données qui peuvent être étudiées plus à fond dans des études expérimentales. L'une des principales limites de notre étude est sa généralisabilité, parce que seule une proportion d'échantillonnage définie a été séquencée à un moment donné (i.e., population ciblée pour les tests de diagnostic de la COVID-19, proportions de spécimens séquencés qui varient dans le temps, spécimens avec un seuil de cycle de PCR de moins de 30). En raison de ces critères stricts pour le séquençage des échantillons, notre ensemble de données d'étude pourrait ne pas être directement représentatif des séquences de Mpro de l'Ontario. De plus, l'absence de données sociodémographiques, cliniques et thérapeutiques a limité l'interprétation de nos résultats dans le contexte du traitement au nirmatrelvir-ritonavir.
Conclusion
Dans l'ensemble, nous avons constaté une très faible présence de souches mutantes résistantes au nirmatrelvir et un manque de temporalité. Nos données suggèrent que l'utilisation actuelle du nirmatrelvir-ritonavir ciblant des populations spécifiques en Ontario pourrait ne pas fournir de pression sélective pour l'émergence de mutants résistants Note de bas de page 37. Enfin, cette étude sous-tend la nécessité d'une surveillance génomique continue et constitue également le fondement de la création d'un système de surveillance automatisé conçu pour suivre l'émergence de mutations résistantes au nirmatrelvir dans le virus du SRAS-CoV-2, en utilisant des données temporelles sur le génome. La capacité de suivre, en temps quasi réel, la fréquence des mutations associées à la résistance aux antimicrobiens peut éclairer la gestion des antimicrobiens nécessaire pour maintenir l'efficacité des médicaments sur une plus longue période.
Déclaration des auteurs
- V. D. — Conceptualisation, logiciels, analyse formelle, rédaction de la version originale, rédaction–révision et édition
- F. S. — Logiciels, analyse formelle, rédaction–révision et édition
- S. I. — Analyse formelle, rédaction–révision et édition
- T. B. — Rédaction–révision et édition
- S. C. — Analyse formelle, rédaction–révision et édition
- A. M. A. — Rédaction–révision et édition
- A. E. — Rédaction–révision et édition
- H. B. — Rédaction–révision et édition
- N. V. — Rédaction–révision et édition
- Y. L. — Logiciels, analyse formelle, rédaction–révision et édition
- K. S. — Rédaction–révision et édition
- H. H. — Rédaction–révision et édition
- K. C. — Rédaction–révision et édition
- A. S. — Rédaction–révision et édition
- A. L. — Rédaction–révision et édition
- A. Z. — Rédaction–révision et édition
- K. R. — Rédaction–révision et édition
- J. K. — Rédaction–révision et édition
- M. H. — Rédaction–révision et édition
- A. C. — Rédaction–révision et édition
- J. G. — Rédaction–révision et édition
- S. P. — Conceptualisation, rédaction–révision et édition
Le contenu de cet article et les opinions qui y sont exprimées n'engagent que les auteurs et ne reflètent pas nécessairement ceux du gouvernement du Canada.
Intérêts concurrents
J. B. Gubbay est un rédacteur scientifique consultant rémunéré pour GIDEON Informatics, Inc., qui n'a aucun lien avec les travaux actuels. Tous les autres auteurs n'ont aucun intérêt concurrent à déclarer.
Remerciements
Nous remercions l'équipe du Service des bibliothèques de Santé publique Ontario qui a appuyé la recension des écrits et l'équipe de Santé publique Ontario qui a contribué à cette étude.
Références
- Notes de bas de page 1
-
U.S. Food and Drug Administration. Coronavirus (COVID-19) Update: FDA Authorizes First Oral Antiviral for Treatment of COVID-19. Washington, DC: FDA; 2021. [Consulté le 21 août 2023]. https://www.fda.gov/news-events/press-announcements/coronavirus-covid-19-update-fda-authorizes-first-oral-antiviral-treatment-covid-19
- Notes de bas de page 2
-
U.S. Food and Drug Administration. Emergency Use Authorization (EUA) for Paxlovid (nirmatrelvir tablets co-packaged with ritonavir tablets) - Center for Drug Evaluation and Research (CDER) Review. Silver Springs, MD: USDHHS; 2021. [Consulté le 21 août 2023]. https://www.fda.gov/media/155194/download
- Notes de bas de page 3
-
Pfizer Labs. Fact sheet for healthcare providers: Emergency Use Authorization for PaxlovidTM. New York, NY: Pfizer; 2024. [Consulté le 26 août 2023]. https://www.fda.gov/media/155050/download
- Notes de bas de page 4
-
Santé Canada. Santé Canada homologue PAXLOVIDTM pour les patients atteints d’une forme légère ou modérée de COVID-19 qui présentent un risque élevé d’évolution vers une forme grave de la maladie. Ottawa, ON : SC; 2022. [Consulté le 23 août 2023]. https://www.canada.ca/fr/sante-canada/nouvelles/2022/01/sante-canada-homologue-paxlovidtm-pour-les-patients-atteints-dune-forme-legere-ou-moderee-de-covid-19-qui-presentent-un-risque-eleve-devolution-ver.html
- Notes de bas de page 5
-
Ontario Health. Recommendations for Antiviral Therapy for Adults with Mild to Moderate COVID-19. [Consulté le 7 juil. 2024]. https://www.ontariohealth.ca/sites/ontariohealth/files/Recommendations-for-Antiviral-Therapy-for-Adults-with-Mild-to-Moderate-COVID-19.pdf
- Notes de bas de page 6
-
Hilgenfeld R. From SARS to MERS: Crystallographic Studies on Coronaviral Proteases Enable Antiviral Drug Design. FEBS J 2014; 281(18):4085–96. https://doi.org/10.1111/febs.12936
- Notes de bas de page 7
-
Anand K, Ziebuhr J, Wadhwani P, Mesters JR, Hilgenfeld R. Coronavirus main proteinase (3CLpro) structure: Basis for design of anti-SARS drugs. Science 2003;300(5626):1763–7. https://doi.org/10.1126/science.1085658
- Notes de bas de page 8
-
Owen DR, Allerton CMN, Anderson AS, Aschenbrenner L, Avery M, Berritt S, Boras B, Cardin RD, Carlo A, Coffman KJ, Dantonio A, Di L, Eng H, Ferre R, Gajiwala KS, Gibson SA, Greasley SE, Hurst BL, Kadar EP, Kalgutkar AS, Lee JC, Lee J, Liu W, Mason SW, Noell S, Novak JJ, Obach RS, Ogilvie K, Patel NC, Pettersson M, Rai DK, Reese MR, Sammons MF, Sathish JG, Singh RSP, Steppan CM, Stewart AE, Tuttle JB, Updyke L, Verhoest PR, Wei L, Yang Q, Zhu Y. An Oral SARS-CoV-2 Mpro Inhibitor Clinical Candidate for the Treatment of COVID-19. Science 2021;374(6575):1586–93. https://doi.org/10.1126/science.abl4784
- Notes de bas de page 9
-
Hammond J, Leister-Tebbe H, Gardner A, Abreu P, Bao W, Wisemandle W, Baniecki M, Hendrick VM, Damle B, Simón-Campos A, Pypstra R, Rusnak JM; EPIC-HR Investigators. Oral Nirmatrelvir for High-Risk, Nonhospitalized Adults with Covid-19. N Engl J Med 2022;386(15):1397–408. https://doi.org/10.1056/NEJMoa2118542
- Notes de bas de page 10
-
Wong CKH, Au ICH, Lau KTK, Lau EHY, Cowling BJ, Leung GM. Real-World Effectiveness of Molnupiravir and Nirmatrelvir plus Ritonavir against Mortality, Hospitalisation, and in-Hospital Outcomes among Community-Dwelling, Ambulatory Patients with Confirmed SARS-CoV-2 Infection during the Omicron Wave in Hong Kong: An Observational Study. Lancet 2022;400(10359):1213–22. https://doi.org/10.1016/S0140-6736(22)01586-0
- Notes de bas de page 11
-
Aggarwal NR, Molina KC, Beaty LE, Bennett TD, Carlson NE, Mayer DA, Peers JL, Russell S, Wynia MK, Ginde AA. Real-World Use of Nirmatrelvir–Ritonavir in Outpatients with COVID-19 during the Era of Omicron Variants Including BA. 4 and BA. 5 in Colorado, USA: A Retrospective Cohort Study. Lancet Infect Dis 2023;23(6):696–705. https://doi.org/10.1016/S1473-3099(23)00011-7
- Notes de bas de page 12
-
Shah MM, Joyce B, Plumb ID, Sahakian S, Feldstein LR, Barkley E, Paccione M, Deckert J, Sandmann D, Gerhart JL, Hagen MB. Paxlovid Associated with Decreased Hospitalization Rate among Adults with COVID-19—United States, April–September 2022. MMWR Morb Mortal Wkly Rep 2022;71(48):1531–7. https://doi.org/10.15585/mmwr.mm7148e2
- Notes de bas de page 13
-
Schwartz KL, Wang J, Tadrous M, Langford BJ, Daneman N, Leung V, Gomes T, Friedman L, Daley P, Brown KA. Population-Based Evaluation of the Effectiveness of Nirmatrelvir–Ritonavir for Reducing Hospital Admissions and Mortality from COVID-19. CMAJ 2023;195(6):E220–6. https://doi.org/10.1503/cmaj.221608
- Notes de bas de page 14
-
Mitchell R, Lee D, Pelude L, Comeau J, Conly J, Ellis C, Ellison J, Embil J, Evans G, Johnston L, Johnstone J, Katz K, Kibsey P, Lee B, Lefebvre MA, Longtin Y, McGeer A, Mertz D, Minion J, Smith S, Srigley J, Suh K, Tomlinson J, Wong A, Thampi N, Frenette C. Utilisation du nirmatrelvir/ritonavir chez les adultes admis à l’hôpital en raison de la COVID-19 pendant la phase omicron de la pandémie de COVID-19, Programme canadien de surveillance des infections nosocomiales. Relevé des maladies transmissibles au Canada 2023;49(7/8):351–7. https://doi.org/10.14745/ccdr.v49i78a07f
- Notes de bas de page 15
-
Banerjee A, Mossman K, Grandvaux N. Molecular Determinants of SARS-CoV-2 Variants. Trends Microbiol 2021;29(10):871–3. https://doi.org/10.1016/j.tim.2021.07.002
- Notes de bas de page 16
-
Hu Y, Lewandowski EM, Tan H, Zhang X, Morgan RT, Zhang X, Jacobs LMC, Butler SG, Gongora MV, Choy J, Deng X, Chen Y, Wang J. Naturally Occurring Mutations of SARS-CoV-2 Main Protease Confer Drug Resistance to Nirmatrelvir. ACS Cent Sci 2023;9(8):1658–69. https://doi.org/10.1021/acscentsci.3c00538
- Notes de bas de page 17
-
Moghadasi SA, Heilmann E, Khalil AM, Nnabuife C, Kearns FL, Ye C, Moraes SN, Costacurta F, Esler MA, Aihara H, von Laer D, Martinez-Sobrido L, Palzkill T, Amaro RE, Harris RS. Transmissible SARS-CoV-2 Variants with Resistance to Clinical Protease Inhibitors. Sci Adv 2023;9(13):eade8778. https://doi.org/10.1126/sciadv.ade8778
- Notes de bas de page 18
-
Iketani S, Mohri H, Culbertson B, Hong SJ, Duan Y, Luck MI, Annavajhala MK, Guo Y, Sheng Z, Uhlemann AC, Goff SP, Sabo Y, Yang H, Chavez A, Ho DD. Multiple Pathways for SARS-CoV-2 Resistance to Nirmatrelvir. Nature 2023;613(7944):558–64. https://doi.org/10.1038/s41586-022-05514-2
- Notes de bas de page 19
-
Li J, Wang Y, Solanki K, Atre R, Lavrijsen M, Pan Q, Baig MS, Li P. Nirmatrelvir Exerts Distinct Antiviral Potency against Different Human Coronaviruses. Antiviral Res 2023;211:105555. https://doi.org/10.1016/j.antiviral.2023.105555
- Notes de bas de page 20
-
Clavel F, Hance AJ. HIV Drug Resistance. N Engl J Med 2004;350(10):1023–35. https://doi.org/10.1056/NEJMra025195
- Notes de bas de page 21
-
Santé publique Ontario. Impact des mutations de la protéase principale du SRAS-CoV-2 sur la résistance au nirmatrelvir/ritonavir (Paxlovid). Toronto, ON : SPO; 2022. [Consulté le 23 août 2023]. https://www.publichealthontario.ca/-/media/Documents/nCoV/ipac/2022/06/sars-cov2-protease-mutations-paxlovid-resistance.pdf?sc_lang=fr
- Notes de bas de page 22
-
Public Health Ontario. Coronavirus Disease 2019 (COVID-19) - Variant of Concern Screening and Whole Genome Sequencing Surveillance. Toronto, ON: PHO; 2022. [Consulté le 9 oct. 2023]. https://www.publichealthontario.ca/en/Laboratory-Services/Test-Information-Index/COVID-19-VoC
- Notes de bas de page 23
-
Ministère de la santé de l’Ontario. Directives provinciales pour les tests de dépistage de COVID-19. Toronto, ON : MSO; 2023. [Consulté le 9 oct. 2023]. https://files.ontario.ca/moh-covid-19-provincial-testing-guidance-fr.pdf
- Notes de bas de page 24
-
Gouvernement de l’Ontario. Mise à jour de l’admissibilité au test PCR et des directives sur la gestion des cas et des contacts en Ontario. Toronto, ON : Gouvernement de l’Ontario; 2021. [Consulté le 23 oct. 2023]. https://news.ontario.ca/en/backgrounder/1001387/updated-eligibility-for-pcr-testing-and-case-and-contact-management-guidance-in-ontario
- Notes de bas de page 25
-
Kneller DW, Phillips G, O'Neill HM, Jedrzejczak R, Stols L, Langan P, Joachimiak A, Coates L, Kovalevsky A. Structural Plasticity of SARS-CoV-2 3CL Mpro Active Site Cavity Revealed by Room Temperature X-Ray Crystallography. Nat Commun 2020;11(1):3202. https://doi.org/10.1038/s41467-020-16954-7
- Notes de bas de page 26
-
Ullrich S, Ekanayake KB, Otting G, Nitsche C. Main Protease Mutants of SARS-CoV-2 Variants Remain Susceptible to Nirmatrelvir. Bioorg Med Chem Lett 2022;62:128629. https://doi.org/10.1016/j.bmcl.2022.128629
- Notes de bas de page 27
-
Lee JT, Yang Q, Gribenko A, Perrin BS Jr, Zhu Y, Cardin R, Liberator PA, Anderson AS, Hao L. Surveillance of SARS-CoV-2 Mpro Reveals High Sequence and Structural Conservation Prior to the Introduction of Protease Inhibitor Paxlovid. mBio 2022;13(4):e0086922. https://doi.org/10.1128/mbio.00869-22
- Notes de bas de page 28
-
Arutyunova E, Khan MB, Fischer C, Lu J, Lamer T, Vuong W, van Belkum MJ, McKay RT, Tyrrell DL, Vederas JC, Young HS, Lemieux MJ. N-Terminal Finger Stabilizes the S1 Pocket for the Reversible Feline Drug GC376 in the SARS-CoV-2 Mpro Dimer. J Mol Biol 2021;433(13):167003. https://doi.org/10.1016/j.jmb.2021.167003
- Notes de bas de page 29
-
Chen SA, Arutyunova E, Lu J, Khan MB, Rut W, Zmudzinski M, Shahbaz S, Iyyathurai J, Moussa EW, Turner Z, Bai B, Lamer T, Nieman JA, Vederas JC, Julien O, Drag M, Elahi S, Young HS, Lemieux MJ. SARS-CoV-2 Mpro Protease Variants of Concern Display Altered Viral Substrate and Cell Host Target Galectin-8 Processing but Retain Sensitivity toward Antivirals. ACS Cent Sci 2023;9(4):696–708. https://doi.org/10.1021/acscentsci.3c00054
- Notes de bas de page 30
-
Iketani S, Hong SJ, Sheng J, Bahari F, Culbertson B, Atanaki FF, Aditham AK, Kratz AF, Luck MI, Tian R, Goff SP, Montazeri H, Sabo Y, Ho DD, Chavez A. Functional Map of SARS-CoV-2 3CL Protease Reveals Tolerant and Immutable Sites. Cell Host Microbe 2022;30(10):1354–62.e6. https://doi.org/10.1016/j.chom.2022.08.003
- Notes de bas de page 31
-
Zhang L, Lin D, Sun X, Curth U, Drosten C, Sauerhering L, Becker S, Rox K, Hilgenfeld R. Crystal Structure of SARS-CoV-2 Main Protease Provides a Basis for Design of Improved α-Ketoamide Inhibitors. Science 2020;368(6489):409–12. https://doi.org/10.1126/science.abb3405
- Notes de bas de page 32
-
Ip JD, Wing-Ho Chu A, Chan WM, Cheuk-Ying Leung R, Umer Abdullah SM, Sun Y, Kai-Wang To K. Global Prevalence of SARS-CoV-2 3CL Protease Mutations Associated with Nirmatrelvir or Ensitrelvir Resistance. EBioMedicine 2023;91:104559. https://doi.org/10.1016/j.ebiom.2023.104559
- Notes de bas de page 33
-
Zhou Y, Gammeltoft KA, Ryberg LA, Pham LV, Tjørnelund HD, Binderup A, Duarte Hernandez CR, Fernandez-Antunez C, Offersgaard A, Fahnøe U, Peters GHJ, Ramirez S, Bukh J, Gottwein JM. Nirmatrelvir-Resistant SARS-CoV-2 Variants with High Fitness in an Infectious Cell Culture System. Sci Adv 2022;8(51):eadd7197. https://doi.org/10.1126/sciadv.add7197
- Notes de bas de page 34
-
Greasley SE, Noell S, Plotnikova O, Ferre R, Liu W, Bolanos B, Fennell K, Nicki J, Craig T, Zhu Y, Stewart AE, Steppan CM. Structural Basis for the in Vitro Efficacy of Nirmatrelvir against SARS-CoV-2 Variants. J Biol Chem 2022;298(6):101972. https://doi.org/10.1016/j.jbc.2022.101972
- Notes de bas de page 35
-
Heilmann E, Costacurta F, Moghadasi SA, Ye C, Pavan M, Bassani D, Volland A, Ascher C, Weiss AKH, Bante D, Harris RS, Moro S, Rupp B, Martinez-Sobrido L, von Laer D. SARS-CoV-2 3CLpro Mutations Selected in a VSV-Based System Confer Resistance to Nirmatrelvir, Ensitrelvir, and GC376. Sci Transl Med 2023;15(678):eabq7360. https://doi.org/10.1126/scitranslmed.abq7360
- Notes de bas de page 36
-
Santé publique Ontario. Résumé épidémiologique hebdomadaire : Surveillance génomique du SRAS-CoV-2 en Ontario, le 6 août 2024. Toronto, ON : SPO; 2023. [Consulté le 22 oct. 2023]. https://www.publichealthontario.ca/-/media/Documents/nCoV/epi/covid-19-sars-cov2-whole-genome-sequencing-epi-summary.pdf?rev=66a6cdcde04046b0abb44b0eaf7d648f&sc_lang=fr
- Notes de bas de page 37
-
Sjaarda CP, Lau L, Simpson JT, Fattouh R, Biondi MJ, Maguire F, Campigotto A, Feng Y, Tozer K, Wong H, Sung WWL, Kim S, Marshall CR, Sheth PM, Kozak R. Prevalence of Low-Frequency, Antiviral Resistance Variants in SARS-CoV-2 Isolates in Ontario, Canada, 2020-2023. JAMA Netw Open 2023;6(7):e2324963. https://doi.org/10.1001/jamanetworkopen.2023.24963
Appendice
| Mutation de Mpro | Données actuelles | Lignée associée | Année de circulation de la lignée | Mutations résistantes au nirmatrelvir in vitro signalées dans chaque lignée |
|---|---|---|---|---|
| A7T | Émergence post-traitement | BA.1.1 | 2022 | 19 |
| FT.1 | 2023 | 1 | ||
| XBB.1.22 | 2023 | 1 | ||
| XBB.1.5 | 2023 | 2 | ||
| A7V | Émergence post-traitement | BA.5.2.1 | 2022 | 1 |
| A7S | Émergence post-traitement | BQ.1 | 2023 | 1 |
| BQ.1.1 | 2023 | 1 | ||
| G15S | Essai biochimique, étude de sélection de la résistance | BA.1.1 | 2022 | 1 |
| BQ.1.1.1 | 2023 | 2 | ||
| L30F | Émergence post-traitement | XBF | 2023 | 1 |
| L50F | Étude sur la sélection de la résistance | BA.2 | 2022 | 2 |
| BA.2.12.1 | 2022 | 1 | ||
| BA.4.6 | 2022 | 2 | ||
| CC.1 | 2022 | 1 | ||
| BA.5.2.1 | 2023 | 1 | ||
| BQ.1.5 | 2023 | 1 | ||
| M82I | Émergence post-traitement | BA.5.1.24 | 2022 | 1 |
| BQ.1.2.3 | 2022 | 9 | ||
| T135I | Essai biochimique | BQ.1 | 2022 | 1 |
| BA.5.1.12 | 2023 | 2 | ||
| E166V | Analyse biochimique, émergence post-traitement | BA.5.2.9 | 2022 | 1 |
| A173S | Essai biochimique, essai de culture cellulaire | BA.2 | 2022 | 1 |
| A173T | Essai biochimique, essai de culture cellulaire | BA.4.6 | 2022 | 4 |
| A173V | Essai biochimique, essai de culture cellulaire | XBB.1.5 | 2023 | 2 |
| XBB.1.9.1 | 2023 | 1 | ||
| Q189K | Essai biochimique | XBB.1.5 | 2023 | 1 |
| T196A | Émergence post-traitement | BA.2 | 2022 | 1 |
| BA.2.3 | 2022 | 1 | ||
| BA.4.1 | 2022 | 1 | ||
| W207S | Émergence post-traitement | BA.1 | 2021 | 1 |
| D248E | Essai biochimique | BQ.1.1 | 2023 | 3 |
| A260T | Émergence post-traitement | FY.5 | 2023 | 1 |
| XBB.1.5.39 | 2023 | 2 | ||
| D263E | Émergence post-traitement | BA.1.1 | 2022 | 3 |
| A266V | Émergence post-traitement | BQ.1.1 | 2022 | 1 |
Abréviation : Mpro, protéase principale |
||||

Cette œuvre est mise à disposition selon les termes de la Licence Creative Commons Attribution 4.0 International