Comparaison des tests utilisés par le programme SRAS-CoV-2 du RCLI, mai 2020 à juin 2021

 Téléchargez cet article en format PDF
Téléchargez cet article en format PDFPublié par : L'Agence de la santé publique du Canada
Numéro : Volume 49-5, mai 2023 : Les technologies innovatrices en santé publique
Date de publication : mai 2023
ISSN : 1719-3109
Soumettre un article
À propos du RMTC
Naviguer
Volume 49-5, mai 2023 : Les technologies innovatrices en santé publique
Science de la mise en œuvre
Comparaison de quinze tests d'amplification de l'acide nucléique du SRAS-CoV-2 utilisés dans le cadre du Programme national de vérification de la compétence en matière de SRAS-CoV-2 du Réseau canadien de laboratoires d'intervention, mai 2020 à juin 2021
Charlene Ranadheera1, Kym Antonation1, Cindi Corbett1
Affiliation
1 Division de la sécurité et de l'intervention en santé, Laboratoire national de microbiologie, Agence de la santé publique du Canada, Winnipeg, MB
Correspondance
Citation proposée
Ranadheera C, Antonation K, Corbett C. Comparaison de quinze tests d'amplification de l'acide nucléique du SRAS-CoV-2 utilisés dans le cadre du Programme national de vérification de la compétence en matière de SRAS-CoV-2 du Réseau canadien de laboratoires d'intervention, mai 2020 à juin 2021. Relevé des maladies transmissibles au Canada 2023;49(5):203–13. https://doi.org/10.14745/ccdr.v49i05a03f
Mots-clés : PCR, SRAS-CoV-2, test d'amplification des acides nucléiques, COVID-19
Résumé
Contexte : Le 11 mars 2020, l'Organisation mondiale de la Santé a déclaré une pandémie causée par le coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2), récemment apparu. Cela a entraîné une augmentation des tests cliniques et une décentralisation de ces tests des laboratoires de santé provinciaux vers les établissements régionaux et privés. S'appuyant sur les résultats du programme national de vérification des compétences (VC) en matière de SRAS-CoV-2 du Réseau canadien de laboratoires d'intervention, cette étude compare plusieurs tests d'amplification des acides nucléiques commerciaux et développés en laboratoire, en évaluant à la fois la sensibilité et la spécificité chez de nombreux utilisateurs.
Méthodes : Chaque panel était composé de six échantillons cliniques simulés en aveugle. Les panels ont été distribués aux laboratoires internationaux, provinciaux et territoriaux, puis aux installations partenaires. Les laboratoires participants ont été invités à soumettre ces échantillons à leurs procédures d'extraction/PCR respective et à communiquer les résultats au Laboratoire national de microbiologie, en indiquant la plateforme d'extraction de l'acide nucléique et le test d'amplification des acides nucléiques utilisés, ainsi que la cible du gène viral et les valeurs de Ct ou équivalentes obtenues. Les données ont été compilées pour chaque plateforme moléculaire et chaque cible génétique utilisée.
Résultats : Les dispositifs des VC ont été déployés en mai 2020, novembre 2020 et juin 2021, ce qui a permis d'obtenir 683 ensembles de données utilisant 37 tests d'amplification d'acide nucléique différents. Au cours des trois programmes d'essais, les participants ont obtenu une note moyenne de 99,3 %, ce qui témoigne de la cohérence des essais entre les laboratoires et les plateformes d'essai.
Conclusion : Cette étude a confirmé la mise en œuvre rapide et réussie d'un programme canadien de VC et a fourni une analyse comparative des différents tests autorisés en cas d'urgence et développés en laboratoire pour la détection du SRAS-CoV-2, et a démontré une concordance globale des tests de 99,3 % à l'échelle nationale.
Introduction
Fin 2019, un nouveau virus respiratoire, le coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2), est apparu dans la province chinoise de Hubei et a ensuite provoqué la pandémie mondiale de maladie à coronavirus 2019 (COVID-19). Le nombre de cas augmentant rapidement, il est devenu nécessaire de décentraliser les tests afin de soutenir les tests aux niveaux fédéral, provincial/territorial et municipal, y compris les laboratoires privés, les hôpitaux et les établissements de soins de santé.
Le Réseau des laboratoires d'intervention du Canada (RCLI) du Laboratoire national de microbiologie (LNM) de Winnipeg, au Canada, fournit des panels de compétence à haut risque pour les agents de menace biologique afin de s'assurer que les laboratoires de santé publique sont prêts à réagir avec des tests de diagnostic de haute qualité. Pendant la pandémie de la COVID-19, le RCLI a été mis à contribution pour développer un programme de vérification des compétentes (VC) afin de soutenir les établissements qui effectuent des tests cliniques sur le SRAS-CoV-2 à l'aide de méthodes moléculaires. À l'instar d'autres efforts internationaux, le programme national de VC en matière de SRAS-CoV-2 soutient la capacité des installations de tests de santé publique à établir leur compétence et à obtenir ou maintenir leur accréditation pour effectuer des tests cliniques sur le SRAS-CoV-2 par rapport à une norme de référence connue afin de garantir la cohérence entre les plateformes de test et les laboratoires dans tout le pays et dans le monde entier Note de bas de page 1 Note de bas de page 2 Note de bas de page 3. Les tests d'amplification des acides nucléiques (TAAN) sont considérés comme la méthode de référence pour la détection des cas actifs de SRAS-CoV-2. Depuis l'émergence du SRAS-CoV-2 en décembre 2019, divers TAAN ont été mis au point, qu'il s'agisse de tests développés en laboratoire ou de tests commerciaux. Cette étude compare les différentes plateformes du TAAN utilisées au Canada au cours de trois programmes de VC, de mai 2020 à juin 2021.
Matériels et méthodes
Production, contrôle de la qualité et distribution des panels
Les virus irradiés ont été dilués dans un mélange de sécrétions nasales humaines négatives servant de matrice de fond, à des concentrations variables, et immédiatement aliquotés dans des tubes pré-étiquetés. Chaque panel était composé de six échantillons cliniques simulés en aveugle. Les échantillons ont été triés par numéro de site, emballés de manière appropriée pour le transport et stockés à -80 °C jusqu'à leur distribution.
Avant la distribution, des mesures de contrôle de la qualité ont été prises pour garantir l'homogénéité et la stabilité de l'échantillon. En bref, dix aliquotes de chaque échantillon ont été retirées du stockage, les acides nucléiques ont été extraits conformément aux instructions du fabricant (MagMaxMD CORE Nucleic Acid Purification Kit, Applied BiosystemsMD, Ontario) et analysés par amplification en chaîne par polymérasequantitative en temps réel (qRT-PCR) (QuantiNovaMD Probe RT-PCR Kit, QiagenMD, Ontario) ciblant le gène E du SRAS-CoV-2 Note de bas de page 4. Les coefficients de variation ont été calculés pour chaque série d'échantillons du panel à l'aide des statistiques descriptives de GraphPadMD Prism. Une valeur moyenne de Ct avec un coefficient de variation inférieur à 10 % était nécessaire pour passer les contrôles de qualité de l'homogénéité de l'échantillon. Les tests de stabilité ont commencé le premier jour après la production et se sont poursuivis à des intervalles spécifiés pendant la durée du programme de VC en utilisant la même approche que celle décrite ci-dessus. Si les contrôles de qualité ont satisfait aux tests d'homogénéité et de stabilité le premier et le septième jour après la production, les panels ont été autorisés à être distribués. Les essais de stabilité se sont poursuivis pendant toute la durée du programme d'essais.
Les panels ont été emballés dans de la glace sèche et distribués aux laboratoires internationaux, provinciaux et territoriaux, qui les ont ensuite distribués à leurs partenaires au sein de leur administration. La chaîne du froid a été contrôlée et, si elle n'était pas maintenue, un nouveau panel a été expédié directement par le LNM.
Sélection des participants et utilisation prévue
Les membres provinciaux et territoriaux du Réseau de laboratoires de santé publique du Canada (RLSPC) ont demandé au LNM de contribuer à la réponse à la pandémie en produisant et en administrant un programme de VC pour le SRAS-CoV-2, car il n'y en avait pas de disponible à l'époque. Les partenaires provinciaux et territoriaux du RLSPC ont fourni au LNM une liste de participants et ont été chargés de la distribution des panels de test dans leurs administrations respectives. Les participants comprenaient des laboratoires provinciaux et territoriaux, des laboratoires de santé publique, des hôpitaux et des établissements de soins de santé dans des communautés urbaines et rurales. Le LNM n'a pas reçu de métadonnées spécifiques ni de détails sur l'autorisation et l'accréditation des sites individuels pour le SRAS-CoV-2.
Le panel de VC a été conçu pour être utilisé comme validation interne des processus moléculaires SRAS-CoV-2, qui sont réalisés en conjonction avec une méthode d'extraction de l'acide nucléique. Ce panel n'a pas été conçu pour être utilisé sur des plateformes nécessitant du matériel d'écouvillonnage frais ou la détection d'antigènes viraux ou d'anticorps spécifiques de virus.
Présentation et analyse des résultats des tests
Les laboratoires participants ont soumis leurs résultats au LNM en précisant la plateforme d'extraction d'acide nucléique et le TAAN utilisés, ainsi que la cible du gène viral et les valeurs de Ct ou équivalentes obtenues. Les données ont été compilées pour chaque plateforme moléculaire et chaque cible génétique utilisée. Le coefficient de variation pour chaque cible génétique au sein d'une même plateforme a été déterminé à l'aide des statistiques descriptives de GraphPadMD Prism. Une analyse par les méthodes des probits utilisant un seuil de 95 % a été utilisée pour déterminer la limite de détection en fonction de la détection de l'échantillon Note de bas de page 5.
Résultats et discussion
Les dispositifs de VC ont été déployés en mai 2020, novembre 2020 et juin 2021, ce qui a permis d'obtenir 683 ensembles de données utilisant 37 TAAN différents (tableau 1). Chaque dispositif de VC a permis d'évaluer la sensibilité et la spécificité de l'essai. Les plateformes les plus couramment utilisées étaient des tests à faible débit entièrement automatisés, tels que le test moléculaire direct pour la COVID-19 de DiaSorin SimplexaMD, Cepheid XpertMD Xpress SRAS-CoV-2, Cepheid XpertMD Xpress SRAS-CoV-2/Flu/RSV et le panel de tests BioFireMD FilmArray RP2.1. Ces systèmes étaient principalement utilisés dans les laboratoires des hôpitaux et dans les communautés rurales. Les centres de diagnostic plus importants, tels que les laboratoires provinciaux et les centres de référence, ont généralement utilisé des tests à haut débit, notamment le test Roche CobasMD SRAS-CoV-2 (pour Cobas 6800/8800), le test 2019 nCoV de Seegene AllplexMD, la trousse Thermo Fisher TaqPathMD COVID-19 Combo et le test développé en laboratoire ciblant le gène E (tableau 1).
| Plateforme de test d'amplification des acides nucléiques | Système de vérification des compétences, Nombre de sites/plateforme |
|||
|---|---|---|---|---|
| Fabricant | Nom du produit | Mai 2020 | Nov 2020 | Juin 2021 |
| AbbottMD | Trousse d'amplification du SRAS-CoV-2 de AlinityMD | 0 | 5 | 16 |
| PCR SRAS-CoV-2 en temps réel | 1 | 3 | 3 | |
| Agena Bioscience | Panel SRAS-CoV-2 de MassARRAYMD | 0 | 0 | 1 |
| Altona | Trousse 1.5 RT-PCR pour le SRAS-CoV-2 d'AltoStarMD | 1 | 1 | 2 |
| BD | Réactifs SRAS-CoV-2 pour le système BD MAXMD | 2 | 9 | 4 |
| BGIMD | Trousse RT-PCR fluorescente en temps réel pour la détection du SRAS-CoV-2 | 0 | 2 | 1 |
| BioFireMD | Panel 2.1 respiratoire de Film ArrayMD | 0 | 20 | 49 |
| Biomeme | SRAS-CoV-2 Go StripsMD | 0 | 1 | 1 |
| Cepheid | XpertMD Xpress SRAS-CoV-2 | 34 | 36 | 52 |
| XpertMD Xpress SRAS-CoV-2/Flu/RSV | 0 | 0 | 29 | |
| DiaSorin | Analyse moléculaire directe de la COVID-19 de SimplexaMD | 5 | 42 | 81 |
| Hologic | Test SRAS-CoV-2 de Panther FusionMD | 0 | 2 | 2 |
| Test SRAS-CoV-2 (system Panther) de AptimaMD | 0 | 6 | 8 | |
| Hyris | bKitMD Virus Finder COVID-19 | 0 | 0 | 1 |
| Test développé en laboratoire | Cible du rapport de fonctionnement 3' | 0 | 0 | 1 |
| Cible du rapport de fonctionnement 5' | 0 | 2 | 4 | |
| Test PCR en temps réel de CDC CoVPlex | 0 | 0 | 1 | |
| Cible du gène E | 12 | 27 | 49 | |
| Cible du gène N | 1 | 1 | 10 | |
| Cible du gène ORF1a/b (RdRp) | 5 | 5 | 8 | |
| Cible du gène S | 0 | 1 | 0 | |
| Cibles des ensembles de gènes E et N | 0 | 1 | 6 | |
| Cibles regroupées des gènes E et ORF1a/b | 0 | 0 | 1 | |
| Cibles regroupées des gènes N, ORF1a/b et S | 0 | 1 | 1 | |
| Luminex | Test SRAS-CoV-2 d'AriesMD | 0 | 1 | 1 |
| Panel d'agents pathogènes respiratoires + SRAS-CoV-2 de NxTAGMD | 0 | 1 | 1 | |
| LuminUltra | Test RT-qPCR pour la COVID-19 de GeneCountMD | 0 | 0 | 1 |
| Quidel | Test SRAS-CoV-2 de LyraMD | 0 | 0 | 3 |
| Test SRAS-CoV-2 de SolanaMD | 0 | 0 | 1 | |
| RIDAMD Gene | Test SRAS-CoV-2 | 0 | 2 | 1 |
| Roche | Test SRAS-CoV-2 (pour Cobas 6800/8800) de CobasMD | 13 | 6 | 19 |
| Test pour SRAS-CoV-2 et Influenza A/B (pour Cobas 6800/8800) de CobasMD | 0 | 0 | 1 | |
| SRAS-CoV-2 de CobasMD (pour LiatMD) | 0 | 0 | 1 | |
| Test pour SRAS-CoV-2 et Influenza A/B de CobasMD (pour Liat) | 0 | 0 | 9 | |
| Seegene | Test 2019 nCoV d'AllplexMD | 4 | 19 | 19 |
| Test SRAS-CoV-2/FluA/FluB/RSV d'AllplexMD | 0 | 0 | 1 | |
| Thermo Fisher Scientific | Trousse combinée COVID-19 de TaqPathMD | 1 | 6 | 15 |
| Nombre total de résultats soumis | 79 | 200 | 404 | |
|
||||
Les résultats du panel obtenus à l'aide de TAAN disponibles dans le commerce et comportant au moins trois ensembles de données dans un schéma de test donné sont présentés dans la figure 1. Les plateformes peu utilisées n'ont pas fait l'objet d'une évaluation plus poussée. Abbott produit deux tests moléculaires de laboratoire à haut débit pour la détection du SRAS-CoV-2 : la trousse Alinity m SRAS-CoV-2 AMP utilisée avec le système Alinity m et le SRAS-CoV-2 RealTime PCR utilisant le système m2000 RealTime. Les deux systèmes ont obtenu les résultats escomptés pour tous les échantillons dans les trois dispositifs d'essai. Tous les sites ont obtenu des résultats cohérents entre novembre 2021 et juin 2021, avec un coefficient de variation inférieur à 10 % (figure 1).
Figure 1 - Équivalent textuel
Cette figure est composée de 11 panels. Chaque panel représente un test commercial d’amplification des acides nucléiques pour la détection du SRAS-CoV-2. Les données présentées comprennent les valeurs Ct pour chaque échantillon obtenu lors du programme de vérification des compétences sur le SRAS-CoV-2 du Réseau canadien de laboratoires d’intervention, de mai 2020 à juin 2021. Les valeurs Ct pour chaque gène cible spécifique dans un test commercial sont représentées par un cercle noir, un carré bleu ou un triangle rouge. Les points de données situés sur l’axe des x indiquent qu’il n’y avait pas d’ARN du SRAS-CoV-2 détectable dans l’échantillon. Le texte au-dessus du point de données est le coefficient de variation (CV) pour chaque ensemble de gènes cibles en utilisant la même couleur représentative. Dans un même échantillon, les cibles génétiques sont généralement regroupées, ce qui indique une détection similaire de l’ARN viral. Dans certains cas, les cibles génétiques sont plus dispersées, ce qui indique une certaine variation dans la détection de l’ARN viral. Les échantillons contenant de faibles quantités de virus avaient des valeurs Ct comprises entre 35 et 40, les échantillons contenant des quantités modérées de virus avaient des valeurs Ct comprises entre 25 et 34, et les échantillons contenant des quantités élevées de virus avaient des valeurs Ct inférieures à 25.
Les réactifs BD SRAS-CoV-2 du système BD MAXMD ax ciblant le gène N ont été utilisés pour la détection du SRAS-CoV-2. Le système BD MAX est un système entièrement automatisé qui permet à l'utilisateur d'analyser jusqu'à 24 échantillons à la fois. Sur une période de 13 mois, les réactifs BD SRAS-CoV-2 pour le système BD MAX ont donné des résultats d'une précision variable. Lors du test de mai 2020, les échantillons ont été détectés avec précision dans tous les cas, mais le coefficient de variation était compris entre 4,8 % et 12,3 %, ce qui indique une variation accrue entre les utilisateurs. Des résultats discordants ont été observés lors du test de novembre 2021; 6/7 échecs de détection du SRAS-CoV-2 ont été attribués à une erreur de l'utilisateur (figure 1, tableau 2); par conséquent, les données obtenues pour l'échantillon G à L ont été faussées et la précision et la cohérence ont été affectées négativement. En supprimant ces points de données, on retrouve une précision globale de 100 % pour la cible N1 et de 99 % pour la cible N2; cette dernière cible n'a pas permis d'identifier la présence de l'échantillon I (figure 1, tableau 2). Lors du test de juin 2021, les réactifs BD SRAS-CoV-2 pour le système BD MAX ont fonctionné avec une précision de 100 %. Les valeurs de Ct étaient cohérentes entre tous les utilisateurs, ce qui se traduit par un coefficient de variation inférieur à 5 % (figure 1).
| Plateforme PCR |
Cible de l'essai | Résultats discordants pour le SRAS-CoV-2 pour la plateforme PCR (%) | Sensibilité : 95 % de détectionNote de bas de page a Note de bas de page b (copies/ml) |
Spécificité | |
|---|---|---|---|---|---|
| Accord positif (%)Note de bas de page b | Accord négatif (%)Note de bas de page b | ||||
Réactifs BD SRAS-CoV-2 pour le système BD MAXMD |
N1 | 6/96 (6,25 %) | 1 100 ou moins | 100 | 100 |
| N2 | 7/96 (7,29 %) | 1 100 ou moins | 100 | 100 | |
| BioFireMD Film ArrayMD RP2.1 | M/S | 1/414 (0,24 %) | 1 100 ou moins | 100 | 100 |
Cepheid XpertMD Xpress SRAS-CoV-2 |
E | 0/730 (0,00 %) | 1 100 ou moins | 100 | 100 |
| N | 6/730 (0,82 %) | 1 100 ou moins | 100 | 97 | |
Test direct moléculaire pour la COVID-19 de DiaSorin SimplexaMD |
ORF1a/b | 3/768 (0,39 %) | 1 100 ou moins | 99,7 | 100 |
| S | 4/768 (0,52 %) | 1 100 ou moins | 99,6 | 100 | |
Panel d'agents pathogènes respiratoires + SRAS-CoV-2 de NxTAGMD |
ORF1a/b | 0/12 (0,00 %) | 1 100 ou moins | 100 | 100 |
| M | 1/12 (8,33 %) | 1 100 ou moins | 100 | 100 | |
| Test pour le gène du SRAS-CoV-2 de RIDAMD | E | 2/18 (11,11 %) | 1 100 ou moins | 100 | 100 |
Test Seegene AllplexMD 2019 nCoV |
E | 11/252 (4,37 %) | 1 100 ou moins | 100 | 100 |
| RdRp | 14/252 (5,56 %) | 1 358 | 99,1 | 100 | |
| N | 9/252 (3,57 %) | 1 100 ou moins | 100 | 100 | |
|
|||||
La trousse de test BioFire Film Array RP2.1 utilise un système entièrement automatisé pour tester la présence de 22 pathogènes différents, dont le SRAS-CoV-2. Ce test comporte une étape d'extraction de l'acide nucléique suivie d'une étape de transcription inverse/de PCR nouée couplée à la technologie de la courbe de fusion de l'acide désoxyribonucléique afin d'identifier qualitativement la présence des pathogènes cibles. Sur les 414 échantillons testés, il n'a pas identifié la présence du SRAS-CoV-2 dans un seul échantillon, ce qui démontre un taux de concordance de 99,8 (tableau 3). Un site n'a pas été en mesure de détecter le SRAS-CoV-2 dans l'échantillon P; cependant, il a été déterminé qu'un mélange insuffisant de l'échantillon de test était probablement responsable des résultats divergents. En outre, ce site a correctement identifié la présence d'autres agents pathogènes cibles, présents dans les échantillons, tels que le rhinovirus (échantillon M), le virus respiratoire syncytial (échantillon K), le virus de la grippe A (échantillons H et O) et le virus de la grippe B (échantillon R) (tableau 3).
| Plateforme | ID de l'échantillon | Échantillon G | Échantillon H | Échantillon I | Échantillon J | Échantillon K | Échantillon L |
|---|---|---|---|---|---|---|---|
| Panel respiratoire 2,1 BioFireMD Film ArrayMD | Résultats attendus | SRAS-CoV-2 détecté | Grippe A détectée | SRAS-CoV-2 détecté | Aucun agent détecté | SRAS-CoV-2 détecté VRS |
SRAS-CoV-2 détecté |
| Exemple de concordance | 100 % (20/20) |
100 % (20/20) |
100 % (20/20) |
100 % (20/20) |
100 % (20/20) |
100 % (20/20) |
|
| ID de l'échantillon | Échantillon M | Échantillon N | Échantillon O | Échantillon P | Échantillon Q | Échantillon R | |
| Résultats attendus | Rhinovirus SRAS-CoV-2 détecté | SRAS-CoV-2 détecté | Grippe A détectée | SRAS-CoV-2 détecté | Aucun agent détecté | SRAS-CoV-2 détecté Grippe B |
|
| Exemple de concordance | 100 % (n = 49/49) |
100 % (n = 49/49) |
100 % (n = 49/49) |
98,6 % (n = 48/49) |
100 % (n = 49/49) |
100 % (n = 49/49) |
|
| Concordance globale | 99,8 % (413/414) | ||||||
| Test pour SRAS-CoV-2 et Influenza A/B de Roche CobasMD (pour Liat) | ID de l'échantillon | Échantillon M | Échantillon N | Échantillon O | Échantillon P | Échantillon Q | Échantillon R |
| Résultats attendus | SRAS-CoV-2 détecté | SRAS-CoV-2 détecté | Grippe A détectée | SRAS-CoV-2 détecté | Aucun agent détecté | SRAS-CoV-2 détecté Grippe B |
|
| Exemple de concordance | 100 % (n = 9/9) |
100 % (n = 9/9) |
100 % (n = 9/9) |
100 % (n = 9/9) |
100 % (n = 9/9) |
100 % (n = 9/9) |
|
| Concordance globale | 100 % (n = 54/54) | ||||||
|
|||||||
La plateforme GeneXpert de Cepheid est déjà utilisée au Canada pour la détection du SRAS-CoV-2 à l'aide des tests Xpert Xpress SRAS-CoV-2 et Xpert Xpress SRAS-CoV-2/Flu/RSV. Le test Xpert Xpress SRAS-CoV-2 E a fonctionné avec précision (taux de détection de 100 %) et cohérence (coefficient de variation inférieur à 5 %) pour tous les échantillons. Toutefois, des résultats discordants ont été observés en utilisant la cible N, en particulier pour l'échantillon H. L'échantillon H ne contenait pas de SRAS-CoV-2, mais une quantité modérée de virions de grippe A (Ct 27); dans six cas, la cible SRAS-CoV-2 N2 a produit un Ct supérieur à 40, ce qui a été considéré comme positif pour le SRAS-CoV-2 par le logiciel GeneXpert (figure 1, tableau 2). À l'exception de l'échantillon H, les valeurs de Ct pour la cible N étaient cohérentes et avaient un coefficient de variation inférieur à 10 %, figure 1. Le test Cepheid Xpert Xpress SRAS-CoV-2/Flu/RSV récemment mis au point a été utilisé dans le cadre du programme de tests de juin 2021 et les résultats concernant le SRAS-CoV-2 ont été combinés pour les cibles E et N2. La plateforme avait une précision de 100 % et produisait des résultats très cohérents avec un coefficient de variation inférieur à 2 % parmi tous les utilisateurs (figure 1). Le test Xpert Xpress SRAS-CoV-2/Flu/RSV a également correctement identifié la présence de l'influenza A et B dans les échantillons O et R, respectivement (données non montrées).
Le test moléculaire direct Diasorin Simplexa COVID-19 est un système automatisé à faible débit qui peut analyser jusqu'à huit échantillons à la fois. Sa principale distinction par rapport à d'autres systèmes similaires, tels que les plateformes BioFire Film Array et Cepheid GeneXpert, est qu'il élimine l'étape d'extraction/purification de l'acide nucléique. Des résultats discordants ont été observés pour les échantillons G et R, la cible ORF1a/b n'ayant pas détecté le SRAS-CoV-2 n = 2/768 fois (0,26 %), tandis que la cible S n'a pas détecté le SRAS-CoV-2 n = 3/768 fois (0,39 %) (figure 1, tableau 2). Selon le fabricant, le test S a un taux de détection de 95,8 % à 500 copies/ml (2 000 copies/ml pour une détection à 100 %) et l'ORF1a/b est détecté dans 93,8 % des cas à 1 000 copies/ml (2 000 copies/ml pour une détection à 100 %) Note de bas de page 6. Des observations similaires ont été faites ici : le test S a donné de meilleurs résultats que le test ORF1a/b (tableau 2). Les échantillons G et R sont respectivement à environ 1 100 et 3 500 copies/ml, ce qui correspond à la limite de détection (LD) du test pour les deux cibles, et est la cause probable des résultats divergents (tableau 4). En outre, un résultat discordant supplémentaire a été obtenu pour chaque cible en raison d'une erreur logicielle qui a indiqué « pas de résultat » lorsque des valeurs Ct ont été obtenues pour les deux cibles (tableau 2). Pour les échantillons où toutes les cibles ont été correctement identifiées (échantillons A–F et H–Q), les coefficients de variation étaient inférieurs ou égaux à 5 %, à l'exception de l'échantillon F qui présentait des coefficients de variation de 11,1 % et 10,1 % pour les cibles ORF1a/b et S, respectivement (figure 1).
| Échantillon | Identité | SRAS-CoV-2 E Nombre approximatif de copies/ml |
Valeur Ct approximative (cible SRAS-CoV-2 E)Note de bas de page a |
|---|---|---|---|
| Programme de vérification des compétences sur le SRAS-CoV-2 du RCLI – mai 2020 | |||
| A | SRAS-CoV-2 de type sauvage | 120 000 000 | 20 |
| B | Blanc | 0 | 0 |
| C | SRAS-CoV-2 de type sauvage | 1 600 | 36 |
| D | SRAS-CoV-2 de type sauvage | 2 700 000 | 25 |
| E | SRAS-CoV-2 de type sauvage | 3 900 | 35 |
| F | SRAS-CoV-2 de type sauvage | 216 000 | 29 |
| Programme de vérification des compétences sur le RCLI SRAS-CoV-2 – novembre 2020 | |||
| G | SRAS-CoV-2 de type sauvage | 1 100 | 36 |
| H | Virus de l'influenza A | 0 | 0 |
| I | SRAS-CoV-2 de type sauvage | 54 000 | 31 |
| J | Blanc | 0 | 0 |
| K | SRAS-CoV-2 de type sauvage | 10 800 | 33 |
| Virus respiratoire syncytial | 0 | ||
| L | SRAS-CoV-2 de type sauvage | 13 000 000 | 22 |
| Programme de vérification des compétences sur le SRAS-CoV-2 du RCLI – juin 2021 | |||
| M | SRAS-CoV-2 B.1.351 | 280 000 | 28 |
| Rhinovirus | 0 | ||
| N | SRAS-CoV-2 B.1.1.7 | 2 100 | 35 |
| O | Virus de l'influenza A | 0 | 0 |
| P | SRAS-CoV-2 P.1 | 1 600 | 36 |
| Q | Blanc | 0 | 0 |
| R | SRAS-CoV-2 de type sauvage | 3 500 | 35 |
| Virus de l'influenza B | 0 | ||
|
|||
Hologic produit deux tests pour le SRAS-CoV-2 qui ont été utilisés dans le cadre des programmes de VC du RCLI pour le SRAS-CoV-2 : Test Panther Fusion SRAS-CoV-2 et test Aptima SRAS-CoV-2. Le test Panther Fusion SRAS-CoV-2 n'a pas été présenté ici, car seuls deux sites emploient cette plateforme, tandis que le test Aptima SRAS-CoV-2 a été utilisé lors des programmes de tests de novembre 2020 et de juin 2021, avec respectivement six et huit utilisateurs (tableau 1). Cette plateforme a démontré une concordance de 100 % (n = 90/90 échantillons). Cependant, les valeurs de Ct obtenues étaient très variables, avec des coefficients de variation allant de 5 % à 19,5 % d'un échantillon à l'autre (figure 1).
Au cours du programme de VC du RCLI de juin 2021, le test Lyra SRAS-CoV-2 de Quidel ciblant l'ORF1a/b a été utilisé pour la première fois par trois participants (tableau 1). Ce test a permis d'identifier correctement tous les échantillons testés (n = 18). Cependant, la variabilité entre les valeurs de Ct était importante, avec un coefficient de variation allant de 17,9 à 27,8 (figure 1). Cette variation des valeurs de Ct est largement attribuée à une série de résultats du panel de test, qui a fourni des valeurs de Ct nettement inférieures à celles des autres participants, ce qui indique des différences dans le réglage des seuils entre les participants.
Le test Seegene Allplex 2019 nCoV est un test RT-PCR multiplex qui détecte les cibles E, N et RdRp et qui peut être automatisé pour les tests à haut volume. Ce test a donné de bons résultats lors des essais de mai 2020 et de juin 2021, avec une concordance de 100 % et des résultats cohérents traduits par un coefficient de variation inférieur à 10 % (figure 1). Toutefois, un certain nombre de résultats discordants ont été observés lors de l'essai de novembre 2020, ce qui a entraîné une baisse de la reproductibilité et une augmentation des coefficients de variation. L'échantillon G a été associé à n = 3/19 échecs de cibles E, n = 4/19 échecs de cibles RdRp et n = 1/19 échecs de cibles N. Alors que n = 2/19 échecs de cibles RdRp ont été associés à l'utilisation d'une plateforme d'extraction nucléique, les autres échecs ont été associés à une divergence par rapport aux recommandations du fabricant et n'ont pas utilisé d'étape d'extraction de l'acide nucléique. En outre, la limite de détection rapportée pour le test Seegene Allplex 2019 nCoV est d'environ 4 000 copies/ml, ce qui est plus élevé que le titre de l'échantillon G et est probablement à l'origine de la non-détection du SRAS-CoV-2 dans cet échantillon Note de bas de page 7 (tableau 4). Inversement, l'échantillon I a été associé à n = 1/1 échecs pour la cible E et n = 2/19 échecs pour les cibles RdRp et N; tandis que l'échantillon K a eu n = 2/19 échecs pour la cible E, n = 3/19 échecs pour la cible RdRp et n = 1/19 échecs pour la cible N. Les échantillons L, H et J ont également été associés à un résultat discordant pour chaque cible en raison de l'impossibilité d'obtenir un résultat valide. Ces échecs de détection du SRAS-CoV-2 ont tous été associés à une utilisation non conforme à l'étiquetage, c'est-à-dire sans procédure d'extraction des acides nucléiques, et sont probablement à l'origine des résultats discordants puisque les titres des échantillons étaient tous supérieurs à 4 000 copies/ml. La pratique consistant à ne pas mettre en œuvre un protocole d'extraction n'a pas été observée dans le programme d'essai suivant. Dans l'ensemble, les cibles E, RdRp et N ont produit des discordances de 4,37 %, 5,56 % et 3,52 %, respectivement (figure 1, tableau 2).
Deux tests Roche différents ont été utilisés dans le cadre des programmes de VC du RCLI sur le SRAS-CoV-2, le test Roche Cobas SRAS-CoV-2, un test entièrement automatisé à haut débit destiné à être utilisé avec le Roche Cobas 5800/6800/8800, et le test sur le SRAS-CoV-2 et l'influenza A/B Roche Cobas pour Liat, un test qualitatif entièrement automatisé destiné à être utilisé sur le Cobas Liat. Le test Roche Cobas SRAS-CoV-2 pour Cobas 5800/6800/8800 a été utilisé dans les trois dispositifs de test, produisant des résultats précis et cohérents avec un coefficient de variation inférieur à 3 % (figure 1). Le test Roche Cobas SRAS-CoV-2 pour Liat a détecté avec précision tous les échantillons de neuf utilisateurs (tableau 1 et tableau 3). Dans l'ensemble, le test Roche Cobas SRAS-CoV-2 à utiliser sur le Cobas 5800/6800/8800 a obtenu les meilleurs résultats lors de la comparaison des plateformes commerciales dans les dispositifs de VC du RCLI sur le SRAS-CoV-2; il a fait preuve d'une précision de 100 % et a produit les résultats les plus reproductibles d'un utilisateur à l'autre.
Le TDL a également été utilisé dans le cadre du programme de VC du RCLI sur le SRAS-CoV-2 de mai 2020 à juin 2021. Les ensembles de données obtenus à l'aide de TDL qui présentent au moins trois séries de résultats dans un dispositif d'essai donné sont présentés (figure 2). Dans tous les cas, tous les tests ont permis de détecter efficacement et précisément le SRAS-CoV-2 à partir des échantillons fournis (figure 2). Les cibles E et RdRp ont été utilisées dans tous les dispositifs d'essai (tableau 1). La reproductibilité de la cible E et de la cible RdRp était assortie de coefficients de variation compris entre 3,9 % et 8,4 % et entre 3,2 % et 10,2 %, respectivement (figure 2). L'utilisation de la cible du rapport de fonctionnement 5' est apparue au cours du programme d'essai de novembre 2020 et les résultats ont été détectés de manière cohérente avec des coefficients de variation inférieurs à 7 % (figure 2). Les laboratoires ont commencé à utiliser le test de la cible N au cours du programme d'essais de juin 2021, avec des coefficients de variation compris entre 6,9 % et 9,6 % (figure 2). Il convient de noter que, hormis le gène ciblé, nous ne disposons pas des détails spécifiques concernant les séquences d'amorces/sondes mises en œuvre par chaque utilisateur et qu'il est possible que les séquences utilisées soient différentes. En général, les valeurs Ct étaient similaires entre tous les tests cibles, ce qui indique des affinités de détection similaires. Toutefois, une analyse comparative directe plus détaillée n'a pas été réalisée, car les tests n'étaient pas identiques. En outre, on s'attend à des changements entre les cibles génétiques, étant donné que l'expression des gènes individuels peut varier au cours de la réplication virale; mais cette constatation pourrait également être attribuée à des variations techniques dans les paramètres de seuil/détection des différents laboratoires. Dans l'ensemble, la cible du rapport de fonctionnement 5' a donné les résultats les plus cohérents avec un coefficient de variation moyen de 4,3 %, suivie par les cibles RdRp (4,7 %), E (5,2 %) et N (7,9 %). Tous les objectifs ont été atteints dans le respect des spécifications prévues, à savoir des coefficients de variation inférieurs à 10 %.
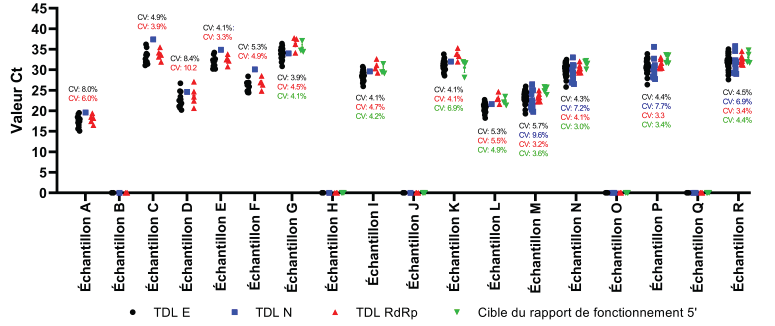
Figure 2 - Équivalent textuel
Cette figure présente les données obtenues à l’aide de tests d’amplification de l’acide nucléique développés en laboratoire pour la détection du SRAS-CoV-2. Les données présentées comprennent les valeurs Ct pour chaque échantillon basé sur le gène cible qui a été obtenu pendant le programme de vérification des compétences sur le SRAS-CoV-2 du Réseau canadien de laboratoires d’intervention de mai 2020 à juin 2021. Tous les tests développés en laboratoire ciblant le gène E (cercle noir), le gène N (carré bleu), le gène RdRp (triangle rouge) et la région 5' non traduite (triangle inversé vert) sont présentés. Les points de données situés sur l’axe des x indiquent qu’il n’y avait pas d’ARN du SRAS-CoV-2 détectable dans l’échantillon. Le texte au-dessus du point de données est le coefficient de variation (CV) pour chaque ensemble de gènes cibles en utilisant la même couleur représentative. Dans un même échantillon, les cibles génétiques sont généralement regroupées, ce qui indique une détection similaire de l’ARN viral. Les échantillons contenant de faibles quantités de virus avaient des valeurs Ct comprises entre 35 et 40, les échantillons contenant des quantités modérées de virus avaient des valeurs Ct comprises entre 25 et 34, et les échantillons contenant des quantités élevées de virus avaient des valeurs Ct inférieures à 25.
Dans l'ensemble, ces résultats donnent une idée de la sensibilité des tests; chaque schéma de test consistait à tester un échantillon contenant de faibles concentrations de particules virales, comprises entre 1 100 et 1 600 copies/ml (échantillon C, 1 600 copies/ml, échantillon G, 1 100 copies/ml ou échantillon P, 1 600 copies/ml). Une sensibilité efficace a été observée pour tous les tests commerciaux et les tests développés en laboratoire utilisés dans le pays. Un taux de concordance de 100 % pour ces échantillons à faible concentration a été observé pour toutes les cibles du SRAS-CoV-2, à quelques exceptions près. La trousse de test BioFire Film Array RP2.1 n'a pas détecté l'échantillon G 1/414 fois (tableau 3). Toutefois, cette erreur est due à une mauvaise manipulation de l'échantillon et, après avoir été répétée à des fins de remédiation, elle a été détectée. Cette erreur n'a donc pas été prise en compte dans l'évaluation générale de la sensibilité (tableau 3).
Le test moléculaire direct pour la COVID-19 de Diasorin Simplexa n'a pas détecté deux échantillons à faible concentration, les deux cibles n'ont pas pu détecter l'échantillon G à deux reprises et la cible S n'a pas détecté l'échantillon R (3 500 copies/ml) à un moment donné (tableau 2). Toutefois, ces résultats discordants n'ont pas affecté la limite de détection de 95 %. Le test Seegene Allplex 2019 nCoV a été associé à un certain nombre d'échecs de détection de l'échantillon G. La majorité de ces échecs ont été attribués à une utilisation non conforme à l'étiquetage, où un processus d'extraction de l'acide nucléique requis a été omis. Pour cette raison, ces résultats ont été retirés de l'analyse de sensibilité qui a suivi. Toutefois, dans deux cas associés à une utilisation correcte, la cible RdRp n'a pas identifié le SRAS-CoV-2 et a été incluse dans l'analyse. Ces résultats discordants ont eu un effet mineur sur la sensibilité du test; une limite de détection de 95 % a été fixée à 1 358 copies/ml (tableau 2). À l'exception du test Seegene Allplex 2019 nCoV, tous les autres tests présentaient des limites de détection de 95 % inférieures à 1 100 copies/ml. Ces résultats observés sont conformes aux limites de détection indiquées par les fabricants pour leurs tests respectifs Note de bas de page 6 Note de bas de page 7 Note de bas de page 8 Note de bas de page 9 Note de bas de page 10 Note de bas de page 11 Note de bas de page 12 Note de bas de page 13 Note de bas de page 14 Note de bas de page 15 Note de bas de page 16. Bien que n'entrant pas dans le cadre de l'utilisation prévue de ce système de VC, cette étude n'a pas été en mesure de calculer la limite de détection pour tous les essais en raison de l'absence d'échantillons présentant des niveaux inférieurs aux niveaux détectables, et il n'a donc pas été possible de comparer davantage la sensibilité des tests.
Outre la sensibilité des tests, leur spécificité a également été évaluée dans le cadre des programmes de VC. Plus précisément, le test de VC de mai 2020 s'est concentré sur la concordance positive et négative, tandis que les tests de novembre 2020 ont ajouté une composante pour la détection d'autres agents pathogènes respiratoires importants, et enfin les tests de juin 2021 se sont appuyés sur le dernier en incluant les variants SRAS-CoV-2 préoccupants (tableau 4). La concordance négative pour l'échantillon B était de 100 % sur toutes les plateformes. Le programme de tests de novembre 2020 comprenait deux échantillons, dont aucun ne contenait le SRAS-CoV-2 : l'échantillon H contenait une dose modérée de virus de la grippe A (Ct 27) et l'échantillon J contenait uniquement la matrice négative de sécrétions nasales/UTM. L'échantillon J a présenté une concordance négative de 100 % sur toutes les plateformes. Cependant, l'échantillon H a présenté quelques incohérences lorsque la plateforme Cepheid Xpert Xpress SRAS-CoV-2 a été utilisée. Dans six cas, conformément aux instructions du fabricant, la cible N a incorrectement identifié la présence du SRAS-CoV-2 dans un échantillon qui ne contenait que le virus de la grippe A (tableau 2). Dans chaque cas, les valeurs Ct étaient supérieures à 40 et suggéraient un certain degré de réactivité croisée avec le virus de la grippe A, car ce phénomène n'a jamais été observé avec aucun des échantillons négatifs. Étant donné que tous les résultats discordants étaient supérieurs à la valeur de 40 Ct, il a été recommandé d'étudier la possibilité de modifier le seuil de Ct en le fixant à 40 au lieu de 45, comme le recommande le fabricant, afin d'éviter de signaler des faux positifs Note de bas de page 17. Au cours des trois programmes de VC, la plateforme Cepheid Xpert Xpress SRAS-CoV-2 a obtenu une concordance négative de 100 % pour la cible E et une concordance négative de 97 % pour la cible N. La concordance négative pour les échantillons O et Q était de 100 % sur toutes les plateformes.
Tous les tests commerciaux et développés en laboratoire ont permis de détecter les variantes concernées. Il convient de noter que la trousse Thermo Fisher TaqPath COVID-19 Combo Kit a enregistré une baisse pour l'un de ses trois gènes cibles; le gène S n'a pas pu détecter la variante B.1.1.7, alors que les deux autres gènes cibles ont été identifiés avec succès. Selon les recommandations du fabricant, un résultat positif exige que n = 2/3 cibles aient des valeurs de Ct inférieures à 37. Par conséquent, la perte du gène S n'a pas entravé la capacité des tests à détecter la présence du SRAS-CoV-2 dans l'échantillon N Note de bas de page 14. L'échec du BioFire Film Array RP2.1 à détecter le SRAS-CoV-2 P.1 a été attribué à une erreur technique et non à un échec du test; ce test n'a donc pas été inclus dans l'analyse. Le BioFire Film Array RP2.1 a détecté avec succès le variant P.1 dans toutes les autres tentatives (n = 48).
Dans l'ensemble, la spécificité du test était comparable pour les trois programmes et plateformes de VC; une concordance négative de 99,5 % a été observée.
Conclusion
Au cours de trois programmes de VC menés à travers le Canada entre mai 2020 et juin 2021, la note moyenne obtenue par les participants a été de 99,3 %, ce qui démontre la cohérence des tests entre les laboratoires et les plateformes d'essai. Des niveaux d'accord aussi élevés ont été observés au niveau international. L'American Proficiency Institute a mené une étude dans l'ensemble des États-Unis et a obtenu une note globale supérieure à 97 % Note de bas de page 3. De même, le « Royal College of Pathologists of Australasia » a mené trois programmes de VC en Australie et en Nouvelle-Zélande entre mars 2020 et novembre 2020, avec un score initial de 75 % de concordance au début de la pandémie, mais qui a ensuite augmenté de façon spectaculaire pour atteindre 95 % de concordance dans les deux derniers programmes de test Note de bas de page 2. Enfin, un troisième programme de Corée du Sud a démontré une concordance de 93 % Note de bas de page 1. Bien que chaque programme ait varié dans la composition de l'échantillon et les utilisations prévues, il est encourageant de constater que le déploiement rapide des tests de dépistage du SRAS-CoV-2 a donné lieu à des degrés de concordance élevés et constants dans le monde entier.
La capacité à soutenir l'assurance qualité des mesures d'essai par le biais d'un programme externe de VC est essentielle en cas de menace nouvelle ou émergente pour la santé publique. Le RCLI fournit un cadre pour soutenir l'assurance qualité nécessaire à la décentralisation et à l'augmentation de la capacité de test au Canada. Tous les laboratoires de santé publique canadiens suivent un programme de gestion de la qualité exigé par leurs administrations respectives, et les systèmes de vérification et de validation sur place sont essentiels à la réalisation de ces processus. En outre, la comparaison des résultats du panel de VC permet d'évaluer diverses plateformes NAAT à différents endroits et pour plusieurs utilisateurs, ce qui permet d'obtenir une évaluation globale des performances de la plateforme. Les performances cumulées du TAAN utilisé au cours des trois programmes de VC du RCLI pour le SRAS-CoV-2 ont été concordantes à 99,3 %. On pourrait envisager à l'avenir de collecter des données supplémentaires auprès des participants afin d'avoir une vision plus large des données démographiques, des statistiques de population et du statut d'accréditation. Cette étude démontre la mise en œuvre rapide et réussie d'un programme canadien de VC et fournit une analyse comparative des différents tests autorisés en cas d'urgence et développés en laboratoire pour la détection du SRAS-CoV-2.
Déclaration des auteurs
C. R. — Conceptualisation, analyse des données, rédaction de la version originale, rédaction de la révision
K. A. — Conceptualisation, rédaction-révision
C. C. — Conceptualisation, rédaction-révision
Intérêts concurrentiels
Aucun.
Remerciements
Nous tenons à remercier les personnes, les organisations et les réseaux suivants qui ont participé à l'exécution du Programme de vérification des compétences sur le SRAS-CoV-2 du Réseau canadien de laboratoires d'intervention (RCLI) :
Le Laboratoire national de microbiologie : N. Bastien (section influenza, virus respiratoires et coronavirus), le programme des agents pathogènes spéciaux, l'équipe d'expédition/réception des échantillons, le bureau de la gestion de la propriété intellectuelle et du développement commercial, les services de sécurité et d'environnement et le centre d'opérations d'urgence.
Groupe de travail sur les infections par les virus respiratoires du Réseau des laboratoires de santé publique du Canada : N. Bastien (Laboratoire national de microbiologie, Winnipeg, Manitoba), P. Levett (BC Centre for Disease Control Public Health Laboratory, Vancouver, Colombie-Britannique), N. Zeylas (Alberta Provincial Laboratory for Public Health, Alberta Precision Laboratories, Edmonton, Alberta), A. Lang (Roy Romanow Provincial Laboratory, Regina, Saskatchewan), K. Dust (Cadham Provincial Laboratory, Winnipeg, Manitoba), J. Gubbay (Public Health Ontario Laboratories, Toronto, Ontario), J. Fafard et V. Dikimpe (Laboratoire de santé publique du Québec, Ste-Anne-de-Bellevue, Québec), G. German et V. Arseneau (Queen Elizabeth Hospital-Health Prince Edward Island, Charlottetown, Île-du-Prince-Édouard), J. LeBlanc (Queen Elizabeth II Health Science Centre, Halifax, Nouvelle-Écosse), Y. Yu (Newfoundland and Labrador Public Health Laboratory, St. John's, Terre-Neuve-et-Labrador), G. Desnoyers (Centre hospitalier universitaire Dr. Georges L. Dumont, Moncton, Nouveau-Brunswick), K. Dionne (Qikiqtani General Hospital, Iqaluit, Nunavut), L. Steven (Stanton Territorial Hospital, Yellowknife, Territoires du Nord-Ouest), et P. Rodgers (Whitehorse General Hospital, Whitehorse, Yukon).
Financement
Ce travail a été soutenu par l'Agence de la santé publique du Canada.
Références
- Note de bas de page 1
-
Sung H, Han MG, Yoo CK, Lee SW, Chung YS, Park JS, Kim MN, Lee H, Hong KH, Seong MW, Lee K, Chun S, Lee WG, Kwon GC, Min WK. Nationwide External Quality Assessment of SARS-CoV-2 Molecular Testing, South Korea. Emerg Infect Dis 2020;26(10):2353–60. https://doi.org/10.3201/eid2610.202551
- Note de bas de page 2
-
Lau KA, Kaufer A, Gray J, Theis T, Rawlinson WD. Proficiency testing for SARS-CoV-2 in assuring the quality and overall performance in viral RNA detection in clinical and public health laboratories. Pathology 2022;54(4):472–8. https://doi.org/10.1016/j.pathol.2022.01.006
- Note de bas de page 3
-
Edson DC, Casey DL, Harmer SE, Downes FP. Identification of SARS-CoV-2 in a Proficiency Testing Program. Am J Clin Pathol 2020;154(4):475–8. https://doi.org/10.1093/ajcp/aqaa128
- Note de bas de page 4
-
Corman VM, Landt O, Kaiser M, Molenkamp R, Meijer A, Chu DK, Bleicker T, Brünink S, Schneider J, Schmidt ML, Mulders DG, Haagmans BL, van der Veer B, van den Brink S, Wijsman L, Goderski G, Romette JL, Ellis J, Zambon M, Peiris M, Goossens H, Reusken C, Koopmans MP, Drosten C. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill 2020;25(3):2000045. https://doi.org/10.2807/1560-7917.ES.2020.25.3.2000045
- Note de bas de page 5
-
Public Health Ontario. Probit Analysis Tool for LOD data. [Consulté le 23 janv. 2023]. https://biostats.shinyapps.io/LOD_probit/
- Note de bas de page 6
-
DiaSorin Molecular LLC. Simplexa® COVID-19 Direct Kit. Cypress, CA; Diasorin; 2021. [Consulté le 25 fév. 2022]. https://molecular.diasorin.com/us/kit/simplexa-covid-19-direct-kit/
- Note de bas de page 7
-
Seegene Inc. AllplexTM 2019-nCoV Assay (version 2.3; Dec 13, 2022) Instructions for Use. Seoul (KR): Seegene; 2020. [Consulté le 25 fév. 2022]. https://www.fda.gov/media/137178/download
- Note de bas de page 8
-
Ag RD. cobas® SARS-CoV-2. Qualitative assay for use on the cobas® 6800/8800 Systems. Roche; 2022; [Consulté le 27 janv. 2023]. https://www.fda.gov/media/136049/download
- Note de bas de page 9
-
Hologic. Aptima SARS-CoV-2 Assay (Panther® system). Hologic; 2022. [Consulté le 27 janv. 2023]. https://www.fda.gov/media/138096/download
- Note de bas de page 10
-
GeneXpert. Xpert® Xpress SARS-CoV-2 Instructions for Use. Cepheid Innovation; April 2022. [Consulté le 27 janv. 2023].https://www.fda.gov/media/136314/download#:~:text=Insert%20the%20swab%20into%20either,or%203%20mL%20of%20saline
- Note de bas de page 11
-
Molecular Diagnostics BD. BD SARS-CoV-2 Reagents for BD MAX™ System. BD Max; 2022. [Consulté le 27 janv. 2023]. https://www.fda.gov/media/136816/download
- Note de bas de page 12
-
Abbott Molecular Inc. Abbott RealTime SARS-CoV-2 Instructions for Use. Abbott; 2022. [Consulté le 27 janv. 2023]. https://www.fda.gov/media/136258/download
- Note de bas de page 13
-
Abbott Molecular Inc. Alinity m SARS-CoV-2 AMP Kit. Abbott; 2022. [Consulté le 27 janv. 2023]. https://www.fda.gov/media/137979/download
- Note de bas de page 14
-
Thermo Fisher Scientific. Applied Biosystems. TaqPath™ COVID-19 Combo Kit, Instructions for use. Multiplex real-time RT-PCR test intended for the qualitative detection of nucleic acid from SARS-CoV-2. Thermo Fisher; 2020. [Consulté le 25 fév. 2022]. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0019211_TaqPath_COVID-19_IFU_Canada.pdf
- Note de bas de page 15
-
BioFire Diagnostics L. BioFire Respiratory Panel 2.1 (RP2.1). Instructions for Use. Biofire; 2022. https://www.biofiredx.qarad.eifu.online/ITI/CA/en/all?keycode=ITI0105
- Note de bas de page 16
-
Quidel Corp. Lyra® SARS-CoV-2 Assay. Instructions For Use. Quidel; 2022. [Consulté le 27 janv. 2023]. https://www.fda.gov/media/136820/download
- Note de bas de page 17
-
Public Health Ontario. An Overview of Cycle Threshold Values and their Role in SARS-CoV-2 Real-Time PCR Test Interpretation. Toronto, ON: PHO; 2020. [Consulté le 27 janv. 2023]. https://www.publichealthontario.ca/-/media/documents/ncov/main/2020/09/cycle-threshold-values-sars-cov2-pcr.pdf?la=en

Cette œuvre est mise à disposition selon les termes de la Licence Creative Commons Attribution 4.0 International